转自: https://club.1688.com/threadview/50123159.htm
简单介绍一下利用单分子标签(Unique Molecular Identifier,UMI)对残留噪音进行矫正或纠错的原理。
UMI矫正的原理有两个关键点:
第一,以单个DNA分子为测序样本,而不是传统胚系突变检测时所用的样本DNA整体。
第二,通过数量换质量,对标记后的单个DNA分子多次测序(也称为高深度测序),进一步提高单个分子的单碱基准确性。
流程下图所示,先将UMI标签添加至DNA分子(双端添加可用于区分正负链,进一步矫正),然后进行文库富集,这时候,不管是指数还是线性,单个分子都被复制成千万个带有相同标记的分子,然后再进行高通量测序。在测序数据中,通过单分子标记将他们重新聚集在一起统一分析,聚合酶错误和测序错误在高深度测序数据中是零散分布的,统计一致性序列后便得到了矫正后的精确序列,可用于对整个样本中的突变频率进行“绝对定量”。值得一提的是,单个UMI的效率,UMI自身的突变纠错以及不同UMI的均匀性等性能均需要在实际操作中进行优化,但这些都不影响该解决方案的实施。
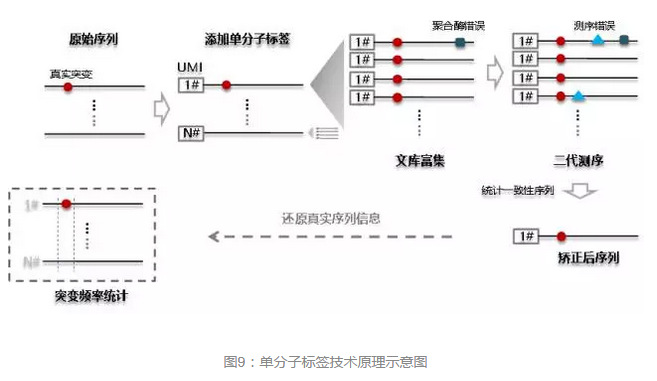
//关键是通过对很多具有相同umi标签的序列进行 一致性统计,这样才能够确定真实序列信息吧。过滤掉测序错误的或者聚合酶错误的等等。