Deep learning with word embeddings 论文阅读笔记
1.将三个模型进行了对比:
i)LSTM-CRF(tagger-master);
ii)使用通用特征集的CRF+词嵌入向量 ;
iii)对每个类的特定实体NER方法。
2.基线模型
2.1CRF模型
CRFSuite:(http://www.chokkan.org/software/crfsuite/)(Okazaki, 2007 )
对所有实体类型使用相同的特征,特征是CRFsuite工具提取的,是和特定领域无关的,默认训练一序线性链CRF模型 。
2.2NER工具
- Chemicals: we employed tmChem (https://www.ncbi.nlm.nih.gov/CBBresearch/Lu/Demo/tmTools/)(Leaman et al., 2015)
- Genes/proteins: we utilized Gimli (http://bioinformatics.ua.pt/software/gimli/) (Campos et al., 2013)
- Species: we used SPECIES (http://species.jensenlab.org/) (Pafiliset al., 2013)
- Diseases: we used DNorm (https://www.ncbi.nlm.nih.gov/CBBresearch/Lu/Demo/tmTools/) (Leaman et al., 2013)
- Cell Lines: we used a model presented in (Kaewphan et al.,2016)
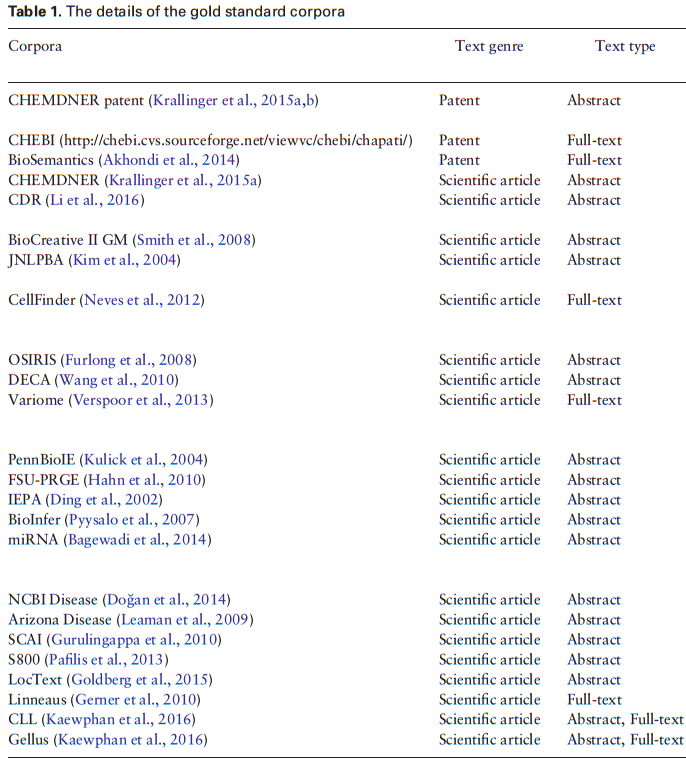
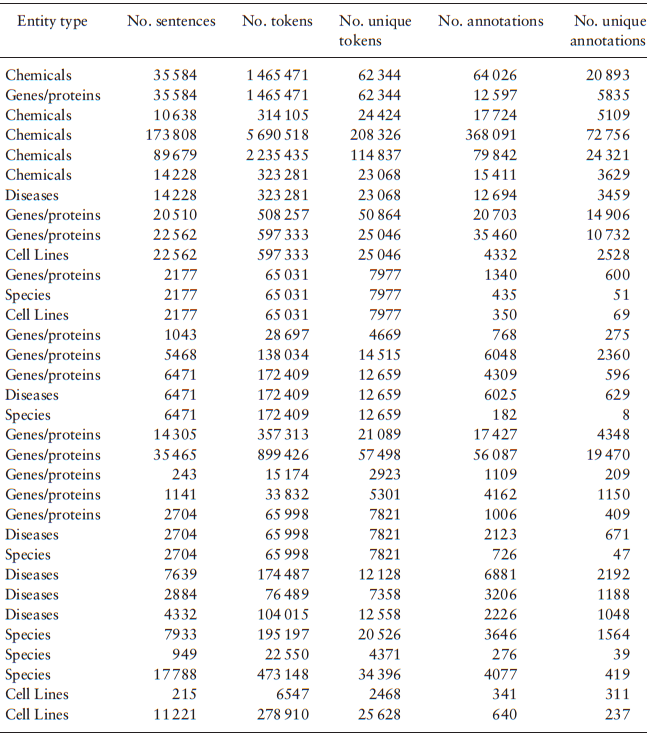
3.数据集
所识别的就是上述5种类型。
包括24个数据集。
4.词向量
i)200维的PubMed-PMC
ii)200维的Wiki-PubMed-PMC
iii)50维的自训练的向量,使用的数据是生物医药主题的欧洲专利,共20000个,工具是Genism;并将Chemd作为开发集调整超参。
5.文本预处理
统一格式:
a)所有文本和标注都存储在一个文件中
b)一个doc:docID doc-text
c)标注,一个标注占一行:字符起始位置 字符终止位置 entity-text 实体类型
d)空行表示doc的结束。
5.1基线模型输入格式:
a)之后再根据每个2.2NER工具的格式进行相应的转换。
b)最终的CRFstuie和LSTM-CRF输入格式是:CoNLL2003 format。使用Apache OpenNLP (https://opennlp.apache.org/) 进行分句、分token和POS标注。
CoNLL2003 格式:
6.评估标准
60%训练集、10%发展集、30%测试集。
Macro and micro averaged performance values in terms of precision, recall and F1-score
CRF适用于特定类的识别性能更好。
7.结论
1)对于专利型的内容,专门使用专利语料库训练了词嵌入向量,发现F1值好了一点点;
对于科学文章,使用Wiki-PubMed-PMC 效果最好。
2)训练数据集越大,得到的词向量效果越好。但是对于具体任务使用什么语料库进行训练词向量仍有待研究。