1.bowtie
短序列比对工具,blast也是短序列比对工具,速度快,结果易理解。
输入可以是fastq或者fasta文件。
生成比对结果文件sam格式的吧。
2.bwa
转自:https://www.jianshu.com/p/1552cc6ac3be
将DNA序列比对到参考基因组上的软件,包含三种算法:
BWA-backtrack:适合比对长度不超过100bp的序列;
BWA-SW:合于长度为70-1M bp的序列;
BWA-MEM:合于长度为70-1M bp的序列,高质量的测序数据,其比对的速度更快,精确度更高。
使用whereis bwa找到其安装路径:
/data1/zzl$ whereis bwa bwa: /usr/bin/bwa /usr/share/bwa /usr/share/man/man1/bwa.1.gz
输入bwa得到以下帮助:
Usage: bwa <command> [options] Command: index index sequences in the FASTA format mem BWA-MEM algorithm fastmap identify super-maximal exact matches pemerge merge overlapping paired ends (EXPERIMENTAL) aln gapped/ungapped alignment samse generate alignment (single ended) sampe generate alignment (paired ended) bwasw BWA-SW for long queries shm manage indices in shared memory fa2pac convert FASTA to PAC format pac2bwt generate BWT from PAC pac2bwtgen alternative algorithm for generating BWT bwtupdate update .bwt to the new format bwt2sa generate SA from BWT and Occ Note: To use BWA, you need to first index the genome with `bwa index'. There are three alignment algorithms in BWA: `mem', `bwasw', and `aln/samse/sampe'. If you are not sure which to use, try `bwa mem' first. Please `man ./bwa.1' for the manual.
步骤:
1.对参照基因组建索引:
bwa index –a bwtsw hg19.fasta
此处构建索引使用的是bwtsw算法,最终输出的结果文件:
会生成:bwt,pac,ann,amb,sa五种类型的文件:
/data1/GRCm38$ ls
GRCm38_68.fa GRCm38_68.fa.amb GRCm38_68.fa.ann GRCm38_68.fa.bwt GRCm38_68.fa.fai GRCm38_68.fa.pac GRCm38_68.fa.sa
2.使用bwa-mem算法进行比对:
bwa mem –t 4 hg19.fasta read1.fq read2.fq > aln-pe.sam
我使用了这条命令:
bwa mem -t 4 ../hg19/hg19.fasta ERR580012_1.fastq.gz ERR580012_2.fastq.gz > aln-pe.sam
使用了mem算法,-t是选择几个线程,增加线程,减少运行时间;然后是参照基因组的fasta文件。以及其他参数:
-p 忽略第二个输入序列,默认情况下,输入一个序列文件认为是单端测序,输入两个序列文件则是双端测序,加上这个参数后,会忽略第二个输入序列文件,把第一个文件当做单端测序的数据进行比对;
将最终结果存入到了sam文件中。
那么什么是单端测序和双端测序:
转自:https://www.cnblogs.com/Formulate0303/p/7843082.html
1、单端测序(Single-ead)首先将DNA样本进行片段化处理形成200-500p的片段,引物序列连接到DNA片段的一端,然后末端加上接头,将片段固定在flowcell上生成DNA簇,上机测序单端读取序列。
2、Paied-end方法是指在构建待测DNA文库时在两端的接头上都加上测序引物结合位点,在第一轮测序完成后,去除第一轮测序的模板链,用对读测序模块(Paied-End Module)引导互补链在原位置再生和扩增,以达到第二轮测序所用的模板量,进行第二轮互补链的合成测序。
//其实这个第二点还不太明白.[1]
3.将sam文件压缩为bam格式
samtools view –bS aln-pe_reorder.sam –o aln-pe.bam
查找samtools帮助:
Usage: samtools <command> [options] Command: view SAM<->BAM conversion sort sort alignment file mpileup multi-way pileup depth compute the depth faidx index/extract FASTA tview text alignment viewer index index alignment idxstats BAM index stats (r595 or later) fixmate fix mate information flagstat simple stats calmd recalculate MD/NM tags and '=' bases merge merge sorted alignments rmdup remove PCR duplicates reheader replace BAM header cat concatenate BAMs bedcov read depth per BED region targetcut cut fosmid regions (for fosmid pool only) phase phase heterozygotes bamshuf shuffle and group alignments by name
-b 表示输出为bam文件格式 –S默认下输入是 BAM 文件,若是输入是 SAM 文件,则最好加该参数,否则有时候会报错。-o 输出文件名
最终生成了bam文件,其中b指binary,运算快。
使用下面命令来查看文件头:
samtools view -H ESCell#8.sam
3.下载并安装gatk
对下载的进行解压。
unzip filename.zip
将本地文件传给服务器直接使用xftp-5软件即可。
解压缩zp2类型压缩文件:
tar -xjf GenomeAnalysisTK-3.3-0.tar.bz2
首次运行命令,提示:
##### ERROR MESSAGE: Fasta index file /data/data0/rawdata/../hg19/hg19.fasta.fai for reference /data/data0/rawdata/../hg19/hg19.fasta does not exist. Please see http://gatkforums.broadinstitute.org/discussion/1601/how-can-i-prepare-a-fasta-file-to-use-as-reference for help creating it.
有个关于参照组的.fai文件找不到,放进去了,然后又说没有dict文件
##### ERROR MESSAGE: Fasta dict file /data/data0/rawdata/../hg19/hg19.dict for reference /data/data0/rawdata/../hg19/hg19.fasta does not exist. Please see http://gatkforums.broadinstitute.org/discussion/1601/how-can-i-prepare-a-fasta-file-to-use-as-reference for help creating it.
5. .fasta.fai文件
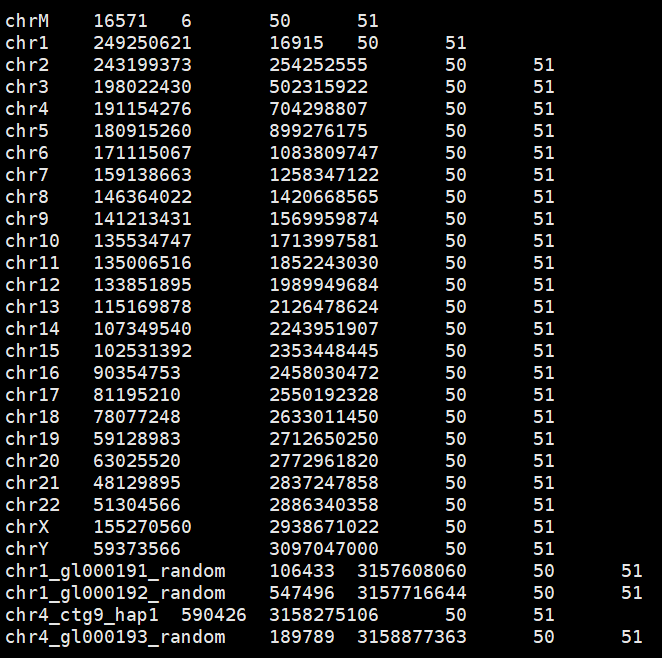
查看其中的内容,每行都有5列
name 序列长度(单位:bp) offset(第一个碱基的偏移量,从0计数,换行符也进行统计) LINEBASES (除了最后一行,其他代表序列的行的碱基数) LINEWIDTH(除了最后一行外, 其他代表序列的行的长度, 包括换行符, 在windows系统中换行符为 , 要在序列长度的基础上加2)
***根据这个.fai 文件和原始的fastsa文件, 能够快速的提取任意区域的序列 。