本分享为脑机学习者Rose整理发表于公众号:脑机接口社区(微信号:Brain_Computer).QQ交流群:903290195

简介
将CanICA应用于静息状态数据的示例。此示例将其应用于ADHD200数据集的30个主题。
然后,它绘制一个地图,所有的组件一起和轴向切割的每个组件分开。
CanICA是一种用于fMRI数据组级分析的ICA方法。与其他策略相比,它带来了一个良好控制的组模型,以及一个阈值算法控制特异性和敏感性的显式模型的信号。
案例
1.加载ADHD200数据
from nilearn import datasets
adhd_dataset = datasets.fetch_adhd(n_subjects=30)
func_filenames = adhd_dataset.func # list of 4D nifti files for each subject
# 数据集基本信息
print('First functional nifti image (4D) is at: %s' %
func_filenames[0]) # 4D data
2.将CanICA应用在核磁共振图像
from nilearn.decomposition import CanICA
canica = CanICA(n_components=20, smoothing_fwhm=6.,
memory="nilearn_cache", memory_level=2,
threshold=3., verbose=10, random_state=0)
canica.fit(func_filenames)
# 检索大脑空间中的独立成分。可通过属性components_img_直接访问。
components_img = canica.components_img_
#components_img是一个Nifti Image对象,可以使用以下行保存到文件中:
components_img.to_filename('canica_resting_state.nii.gz')
3.可视化
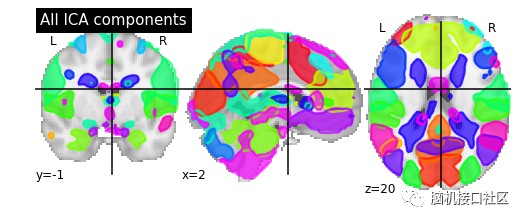
3.1将所有的组件都放在同一组图片上
from nilearn.plotting import plot_prob_atlas
import warnings
warnings.filterwarnings("ignore")
# Plot all ICA components together
plot_prob_atlas(components_img, title='All ICA components')
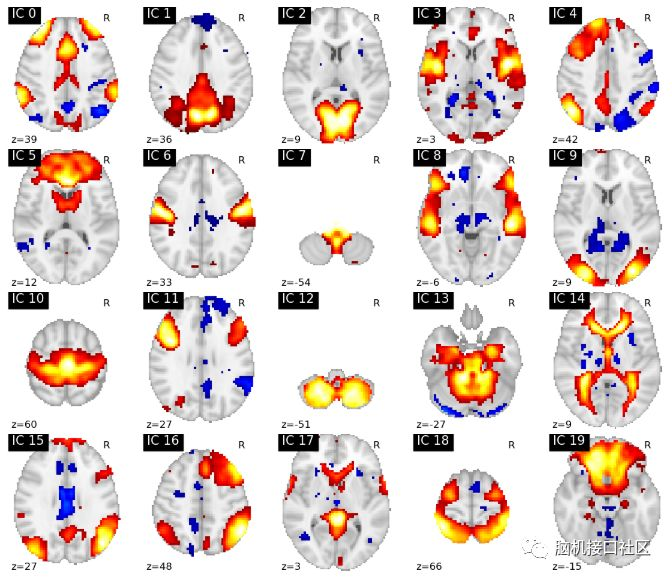
3.2将所有的ICA组件单独绘制
from nilearn.image import iter_img
from nilearn.plotting import plot_stat_map, show
import warnings
warnings.filterwarnings("ignore")
for i, cur_img in enumerate(iter_img(components_img)):
plot_stat_map(cur_img, display_mode="z", title="IC %d" % i,
cut_coords=1, colorbar=False)
show()
文献参考:
G. Varoquaux et al. "ICA-based sparse features recovery from fMRI datasets", IEEE ISBI 2010, p. 1177
G. Varoquaux et al. "A group model for stable multi-subject ICA on fMRI datasets", NeuroImage Vol 51 (2010), p. 288-299
脑机学习者Rose笔记分享,QQ交流群:903290195
更多分享,请关注公众号
