一 简单介绍
作为标准科学计算程序库,SciPy类似于Matlab的工具箱,它是Python科学计算程序的核心包,它用于有效地计算NumPy矩阵,与NumPy矩阵协同工作。
SciPy库由一些特定功能的子模块构成,如下表所示:
| 模块 | 功能 |
| cluster | 矢量量化 / K-均值 |
| constants | 物理和数学常数 |
| fftpack | 傅里叶变换 |
| integrate | 积分程序 |
| interpolate | 插值 |
| io | 数据输入输出 |
| linalg | 线性代数程序 |
| ndimage | n维图像包 |
| odr | 正交距离回归 |
| optimize | 优化 |
| signal | 信号处理 |
| sparse | 稀疏矩阵 |
| spatial | 空间数据结构和算法 |
| special | 任何特殊数学函数 |
| stats | 统计 |
import numpy as np
from scipy import stats
以上代码表示从SciPy模块中导入stats子模块,SciPy的其他子模块导入方式与之相同,限于机器学习研究领域及篇幅限制,本章将重点介绍linalg、optimize、interpolate及stats模块。
>>> from scipy import linalg
>>> arr = np.array([[1, 2], [3, 4]])
>>> linalg.det(arr)
-2.0
>>> arr = np.array([[3, 2],[6, 4]])
>>> linalg.det(arr)
0.0
>>> linalg.det(np.ones((3, 4))) #无论行列式还是逆矩阵只适用于n阶矩阵的求解
Traceback (most recent call last):
...
ValueError: expected square matrix
scipy.linalg.inv()函数计算方阵的逆,示例代码:
>>> arr = np.array([[1, 2], [3, 4]])
>>> iarr = linalg.inv(arr)
>>> iarr
array([[-2. , 1. ],
[ 1.5, -0.5]])
>>>np.allclose(np.dot(arr, iarr), np.eye(2)) #numpy.allclose()函数用于比较两方阵所有对应元素值,如果完全相同返回真(True),否则返回假(False)
True
以下计算奇异阵(行列式为0)的逆,其结果将会报错(LinAlgError),示例代码:
>>>arr = np.array([[3, 2], [6, 4]])
>>>linalg.inv(arr)
Traceback (most recent call last):
...
...LinAlgError: singular matrix
scipy.linalg.norm()函数计算方阵的范数,示例代码:
>>>A = np.matrix(np.random.random((2, 2)))
>>>A
>>>linalg.norm(A) #默认2范数
>>>linalg.norm(A, 1) #1范数
>>>linalg.norm(A, np.inf) #无穷范数
(2)解线性方程组
在一些矩阵公式中经常会出现类似于A-1B的运算,它们都可以用solve(A, B)计算,这要比直接逆矩阵然后做矩阵乘法更快捷一些,下面的程序比较solve()和逆矩阵的运算速度,示例代码:
>>> import numpy as np
>>> from scipy import linalg
>>> m, n = 500, 50
>>> A = np.random.rand(m, m)
>>> B = np.random.rand(m, n)
>>> X1 = linalg.solve(A, B)
>>> X2 = np.dot(linalg.inv(A), B)
>>> print(np.allclose(X1, X2))
>>> %timeit linalg.solve(A, B)
13.3 ms ± 834 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
>>> %timeit np.dot(linalg.inv(A), B)
22.4 ms ± 1.48 ms per loop (mean ± std. dev. of 7 runs, 10 loops each)
(3) 特征值和特征向量
下面以二维平面上的线性变换矩阵为例,演示特征值和特征向量的几何含义。通过linalg.eig(A)计算矩阵A的两个特征值evalues和特征向量evectors,在evectors中,每一列是一个特征向量。示例代码:
>>> A = np.array([[1, -0.3], [-0.1, 0.9]])
>>> evalues, evectors = linalg.eig(A)
2.2 拟合与求解optimize模块
(1)拟合 curve_fit()函数
以下示例中,我们首先从已知函数中生成一些带有噪声的数据,然后使用curve_fit()函数拟合这些噪声数据。示例中的已知函数我们使用一个简单的线性方程式,即f(x)=ax+b。示例代码:
import numpy as np
from scipy.optimize import curve_fit
import matplotlib.pyplot as plt
#创建函数模型用来生成数据
def func(x, a, b):
return a*x + b
#生成干净数据
x = np.linspace(0, 10, 100)
y = func(x, 1, 2)
#对原始数据添加噪声
yn = y + 0.9 * np.random.normal(size=len(x))
#使用curve_fit函数拟合噪声数据
popt, pcov = curve_fit(func, x, yn)
#输出给定函数模型func的最优参数
print(popt)
结果为:
[ 0.99734363 1.96064258]
如果有一个很好的拟合效果,popt返回的是给定模型的最优参数。我们可以使用pcov的值检测拟合的质量,其对角线元素值代表着每个参数的方差。
>>>print(pcov)
[[ 0.00105056 -0.00525282]
[-0.00525282 0.03519569]]
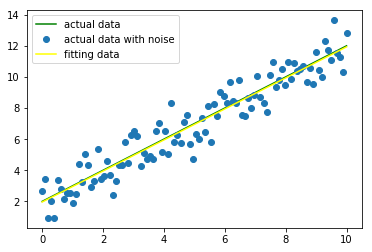
通过以下代码绘制出了拟合曲线与实际曲线的差异,示例代码:
yfit = func(x,popt[0],popt[1])
plt.plot(x, y, color="green",label = "actual data")
plt.plot(x, yn, "o", label = "actual data with noise")
plt.plot(x, yfit,color="yellow", label = "fitting data")
plt.legend(loc = "best")
plt.show()
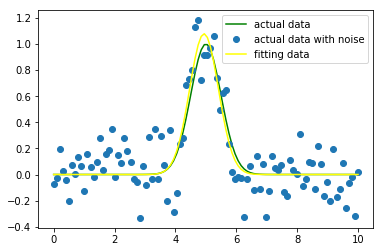
 下面做进一步研究,我们可以通过最小二乘拟合高斯分布(Gaussian profile),一种非线性函数:α*exp(-(x-μ)2/2σ2)
下面做进一步研究,我们可以通过最小二乘拟合高斯分布(Gaussian profile),一种非线性函数:α*exp(-(x-μ)2/2σ2)
在这里,α表示一个标量,μ是期望值,而σ是标准差。示例代码:
import numpy as np
from scipy.optimize import curve_fit
import matplotlib.pyplot as plt
#创建一个函数模型用来生成数据
def func(x, a, b, c):
return (a*np.exp(-(x-b)**2/2*c**2))
#生成原始数据
x = np.linspace(0, 10, 100)
y = func(x, 1, 5, 2)
#对原始数据增加噪声
yn = y + 0.2*np.random.normal(size=len(x))
#使用curve_fit函数拟合噪声数据
popt, pcov = curve_fit(func, x, yn)
#popt返回最拟合给定的函数模型func的参数值,如popt[0]=a,popt[1]=b,popt[2]=3
print(popt)
结果为:
[-0.49627942 2.78765808 28.76127826]
通过以下代码绘制出了拟合曲线与实际曲线的差异,示例代码:
p0=[1.2,4,3] #初步猜测参数,如果没有,默认全为1,即[1,1,1]
popt, pcov = curve_fit(func, x, yn,p0=p0)
#popt返回最拟合给定的函数模型func的参数值,如popt[0]=a,popt[1]=b,popt[2]=3
print(popt)
yfit = func(x,popt[0],popt[1],popt[2])
plt.plot(x, y, color="green",label = "actual data")
plt.plot(x, yn, "o", label = "actual data with noise")
plt.plot(x, yfit, color="yellow", label = "fitting data")
plt.legend(loc = "best")
plt.show()
结果如下图所示:
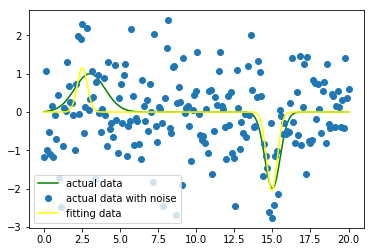
随着研究的深入,我们可以拟合一个多重高斯分布的一维数据集。现在将这个函数扩展为包含两个不同输入值的高斯分布函数。这是一个拟合线性光谱的经典实例,示例代码如下:
import numpy as np
from scipy.optimize import curve_fit
import matplotlib.pyplot as plt
def func(x, a0, b0, c0, a1, b1, c1):
return a0*np.exp(-(x - b0) ** 2/(2 * c0 ** 2)) + a1 * np.exp(-(x-b1) ** 2/(2 * c1 ** 2))
#生成原始数据
x = np.linspace(0, 20, 200)
y = func(x, 1, 3, 1, -2, 15, 0.5)
#对原始数据增加噪声
yn = y + 0.9 * np.random.normal(size=len(x))
#如果要拟合一个更加复杂的函数,提供一些估值假设对拟合效果更好
guesses = [1, 3, 1, 1, 15, 1]
#使用curve_fit函数拟合噪声数据
popt, pcov = curve_fit(func, x, yn, p0=guesses)
#popt返回最拟合给定的函数模型func的参数值,如popt[0]=a,popt[1]=b,popt[2]=3
print(popt)
yfit = func(x,popt[0],popt[1],popt[2],popt[3],popt[4],popt[5])
plt.plot(x, y, color="green",label = "actual data")
plt.plot(x, yn, "o", label = "actual data with noise")
plt.plot(x, yfit, color="yellow", label = "fitting data")
plt.legend(loc = "best")
plt.show()
结果如下图所示:
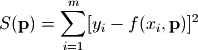 这种算法被称之为最小二乘拟合(Least-square fitting)。optimize模块提供了实现最小二乘拟合算法的函数leastsq(),leastsq是least square的简写,即最小二乘法。下面是用leastsq()对线性函数进行拟合的程序,示例代码:
这种算法被称之为最小二乘拟合(Least-square fitting)。optimize模块提供了实现最小二乘拟合算法的函数leastsq(),leastsq是least square的简写,即最小二乘法。下面是用leastsq()对线性函数进行拟合的程序,示例代码:import matplotlib.pylab as plt
import numpy as np
from scipy import optimize # 从scipy库引入optimize模块
X = np.array([ 8.19, 2.72, 6.39, 8.71, 4.7, 2.66, 3.78 ])
Y = np.array([ 7.01, 2.78, 6.47, 6.71, 4.1, 4.23, 4.05 ])
def residuals(p):
#计算以p为参数的直线和原始数据之间的误差
k, b = p
return Y-(k*X+b)
# leastsq()使得residuals()的输出数组的平方和最小,参数的初始值为[1, 0]
r = optimize.leastsq(residuals, [1,0])
k, b = r[0]
print("k=", k, "b=", b)
结果为:
k = 0.613495349193 b = 1.79409254326
可以通过通过绘图对比真实数据和拟合数据的误差,示例代码;
plt.plot(X, Y, "o", label = "actual data")
plt.plot(X, k*X+b, label = "fitting data")
plt.legend(loc = "best")
plt.show()
结果为:
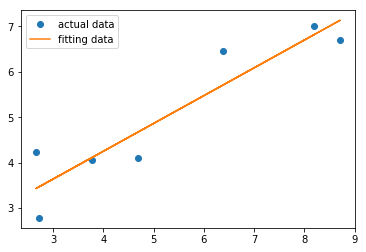 绘图中的圆点表示真实数据点,实线表示拟合曲线,由此看出拟合参数得到的函数和真实数据大体一致。接下来,用leastsq()对正弦波数据进行拟合,示例代码:
绘图中的圆点表示真实数据点,实线表示拟合曲线,由此看出拟合参数得到的函数和真实数据大体一致。接下来,用leastsq()对正弦波数据进行拟合,示例代码:import numpy as np
from scipy.optimize import leastsq # 从scipy库的optimize模块引入leastsq函数
import matplotlib.pyplot as plt # 引入绘图模块pylab,并重命名为pl
def func(x, p):
"""
数据拟合所用的函数: A*sin(2*pi*k*x + theta)
"""
A, k, theta = p
return A*np.sin(2*np.pi*k*x+theta)
def residuals(p, y, x):
"""
实验数据x, y和拟合函数之间的差,p为拟合需要找到的系数
"""
return y - func(x, p)
x = np.linspace(0, -2*np.pi, 100)
A, k, theta = 10, 0.34, np.pi/6 # 真实数据的函数参数
y0 = func(x, [A, k, theta]) # 真实数据
y1 = y0 + 2 * np.random.randn(len(x)) # 加入噪声之后的实验数据,噪声是服从标准正态分布的随机量
p0 = [7, 0.2, 0] # 第一次猜测的函数拟合参数
# 调用leastsq进行数据拟合
# residuals为计算误差的函数
# p0为拟合参数的初始值
# args为需要拟合的实验数据
plsq = leastsq(residuals, p0, args=(y1, x))
print ("actual parameter:", [A, k, theta]) # 真实参数
print ("fitting parameter", plsq[0]) # 实验数据拟合后的参数
plt.plot(x, y0, label="actual data") # 绘制真实数据
plt.plot(x, y1, label="experimental data with noise") # 带噪声的实验数据
plt.plot(x, func(x, plsq[0]), label="fitting data") # 拟合数据
plt.legend()
plt.show()
>>>actual parameter: [10, 0.34, 0.5235987755982988]
>>>fitting parameter [ 10.12646889 0.33767587 0.48944317]
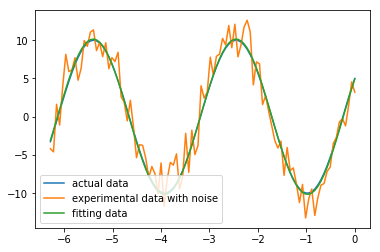
(3)标量函数极值求解fmin()函数
首先定义以下函数,然后绘制它,示例代码:
import numpy as np
from scipy import optimize
import matplotlib.pyplot as plt
def f(x):
return x**2 + 10*np.sin(x)
x = np.arange(-10, 10, 0.1)
plt.plot(x, f(x))
plt.show()
结果如下图所示:

>>> optimize.fmin_bfgs(f, 0)
Optimization terminated successfully.
Current function value: -7.945823
Iterations: 5
Function evaluations: 24
Gradient evaluations: 8
array([-1.30644003])
这个方法一个可能的问题在于,如果函数有局部最小值,算法会因初始点不同找到这些局部最小而不是全局最小,示例代码:
>>> optimize.fmin_bfgs(f, 3, disp=0)#disp是布尔型数据,如果为1,打印收敛消息
array([ 3.83746663])
如果我们不知道全局最小值的邻近值来选定初始点,我们需要借助于耗费资源些的全局优化。为了找到全局最小点,最简单的算法是蛮力算法,该算法求出给定格点的每个函数值。示例代码:
>>>grid = (-10, 10, 0.1)
>>>xmin_global = optimize.brute(f, (grid, ))
>>>xmin_global
array([-1.30641113])
>>> xmin_local = optimize.fminbound(f, 0, 10)
>>> xmin_local
3.8374671...
下面的程序通过求解卷积的逆运算演示fmin的功能。对于一个离散线性时不变系统h, 如果输入是x,那么其输出y可以用x和h的卷积表示:

import scipy.optimize as opt import numpy as np def test_fmin_convolve(fminfunc, x, h, y, yn, x0): """ x (*) h = y, (*)表示卷积 yn为在y的基础上添加一些干扰噪声的结果 x0为求解x的初始值 """ def convolve_func(h): """ 计算 yn - x (*) h 的power fmin将通过计算使得此power最小 """ return np.sum((yn - np.convolve(x, h))**2) # 调用fmin函数,以x0为初始值 h0 = fminfunc(convolve_func, x0) print fminfunc.__name__ print "---------------------" # 输出 x (*) h0 和 y 之间的相对误差 print "error of y:", np.sum((np.convolve(x, h0)-y)**2)/np.sum(y**2) # 输出 h0 和 h 之间的相对误差 print "error of h:", np.sum((h0-h)**2)/np.sum(h**2) print def test_n(m, n, nscale): """ 随机产生x, h, y, yn, x0等数列,调用各种fmin函数求解b m为x的长度, n为h的长度, nscale为干扰的强度 """ x = np.random.rand(m) h = np.random.rand(n) y = np.convolve(x, h) yn = y + np.random.rand(len(y)) * nscale x0 = np.random.rand(n) test_fmin_convolve(opt.fmin, x, h, y, yn, x0) test_fmin_convolve(opt.fmin_powell, x, h, y, yn, x0) test_fmin_convolve(opt.fmin_cg, x, h, y, yn, x0) test_fmin_convolve(opt.fmin_bfgs, x, h, y, yn, x0) test_n(200, 20, 0.1)
运行结果为:

fmin --------------------- error of y: 0.000360456186137 error of h: 0.0122264525455 Optimization terminated successfully. Current function value: 0.207509 Iterations: 96 Function evaluations: 17400 fmin_powell --------------------- error of y: 0.000129249083036 error of h: 0.000300953639205 Optimization terminated successfully. Current function value: 0.207291 Iterations: 20 Function evaluations: 880 Gradient evaluations: 40 fmin_cg --------------------- error of y: 0.000129697740414 error of h: 0.000292820536053 Optimization terminated successfully. Current function value: 0.207291 Iterations: 31 Function evaluations: 946 Gradient evaluations: 43 fmin_bfgs --------------------- error of y: 0.000129697643272 error of h: 0.000292817401206
(4)函数求解fsolve()
- func是用于定义需求解的非线性方程组的函数文件名
- x0为未知数矢量的初始值。
import matplotlib.pyplot as plt
from scipy.optimize import fsolve
import numpy as np
line = lambda x:x+3
solution = fsolve(line, -2)
print(solution)
结果为:
[-3,]
通过以下绘图函数可以看出当函数等于0时,x轴的坐标值为-3,示例代码:
x = np.linspace(-5.0, 0, 100)
plt.plot(x,line(x), color="green",label = "function")
plt.plot(solution,line(solution), "o", label = "root")
plt.legend(loc = "best")
plt.show()
结果为:
 下面我们通过一个简单的示例介绍一下两个方程交点的求解方法,示例代码:
下面我们通过一个简单的示例介绍一下两个方程交点的求解方法,示例代码:from scipy.optimize import fsolve
import numpy as np
import matplotlib.pyplot as plt
# 定义解函数
def findIntersection(func1, func2, x0):
return fsolve(lambda x: func1(x)-func2(x),x0)
# 定义两方程
funky = lambda x : np.cos(x / 5) * np.sin(x / 2)
line = lambda x : 0.01 * x - 0.5
# 定义两方程交点的取值范围
x = np.linspace(0, 45, 1000)
result = findIntersection(funky, line, [15, 20, 30, 35, 40, 45])
# 输出结果
print(result, line(result))
plt.plot(x,funky(x), color="green",label = "funky func")
plt.plot(x,line(x), color="yellow",label = "line func")
plt.plot(result,line(result), "o", label = "intersection")
plt.legend(loc = "best")
plt.show()
结果为:
 如果要对如下方程组进行求解的话:
如果要对如下方程组进行求解的话:- f1(u1,u2,u3) = 0
- f2(u1,u2,u3) = 0
- f3(u1,u2,u3) = 0
def func(x):
u1,u2,u3 = x
return [f1(u1,u2,u3), f2(u1,u2,u3), f3(u1,u2,u3)]
下面是一个实际的例子,求解如下方程组的解:
- 5*x1 + 3 = 0
- 4*x0*x0 - 2*sin(x1*x2) = 0
- x1*x2 - 1.5 = 0
from scipy.optimize import fsolve
from math import sin,cos
def f(x):
x0 = float(x[0])
x1 = float(x[1])
x2 = float(x[2])
return [
5*x1+3,
4*x0*x0 - 2*sin(x1*x2),
x1*x2 - 1.5
]
result = fsolve(f, [1,1,1])
print (result)
结果为:
[-0.70622057 -0.6 -2.5 ]
2.3 插值interpolate模块
计算插值有两种基本的方法,1、对一个完整的数据集去拟合一个函数;2、对数据集的不同部分拟合出不同的函数,而函数之间的曲线平滑对接。第二种方法又叫做仿样内插法,当数据拟合函数形式非常复杂时,这是一种非常强大的工具。我们首先介绍怎样对简单函数进行一维插值运算,然后进一步深入比较复杂的多维插值运算。
interp1d(x, y, kind='linear', ...)
其中,x和y参数是一系列已知的数据点,kind参数是插值类型,可以是字符串或整数,它给出插值的B样条曲线的阶数,候选值及作用下表所示:
| 候选值 | 作用 |
| ‘zero’ 、'nearest' | 阶梯插值,相当于0阶B样条曲线 |
| ‘slinear’ 、'linear' | 线性插值,用一条直线连接所有的取样点,相当于一阶B样条曲线 |
| ‘quadratic’ 、'cubic' | 二阶和三阶B样条曲线,更高阶的曲线可以直接使用整数值指定 |
import numpy as np
from scipy.interpolate import interp1d
import matplotlib.pyplot as plt
#创建待插值的数据
x = np.linspace(0, 10*np.pi, 20)
y = np.cos(x)
# 分别用linear和quadratic插值
fl = interp1d(x, y, kind='linear')
fq = interp1d(x, y, kind='quadratic')
#设置x的最大值和最小值以防止插值数据越界
xint = np.linspace(x.min(), x.max(), 1000)
yintl = fl(xint)
yintq = fq(xint)
plt.plot(xint,fl(xint), color="green", label = "Linear")
plt.plot(xint,fq(xint), color="yellow", label ="Quadratic")
plt.legend(loc = "best")
plt.show()
结果如下图所示:
 (2)噪声数据插值
(2)噪声数据插值import numpy as np
from scipy.interpolate import UnivariateSpline
import matplotlib.pyplot as plt
# 通过人工方式添加噪声数据
sample = 30
x = np.linspace(1, 10*np.pi, sample)
y = np.cos(x) + np.log10(x) + np.random.randn(sample)/10
# 插值,参数s为smoothing factor
f = UnivariateSpline(x, y, s=1)
xint = np.linspace(x.min(), x.max(), 1000)
yint = f(xint)
plt.plot(xint,f(xint), color="green", label = "Interpolation")
plt.plot(x, y, color="yellow", label ="Original")
plt.legend(loc = "best")
plt.show()
需要说明的是:在UnivariateSpline()函数中,参数s是平滑向量参数,被用来拟合还有噪声的数据。如果参数s=0,将忽略噪声对所有点进行插值运算。结果如下图所示: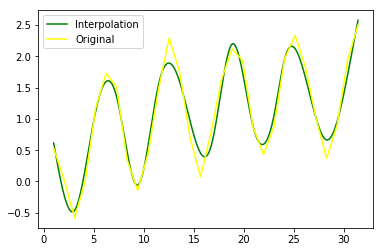
griddata(points, values, xi, method='linear', fill_value=nan)
import numpy as np
from scipy.interpolate import griddata#定义一个函数
def ripple(x,y):
return np.sqrt(x**2 + y**2) + np.sin(x**2 + y**2)
# 生成grid数据,复数定义了生成grid数据的step,若无该复数则step为5
grid_x, grid_y = np.mgrid[0:5:1000j, 0:5:1000j]
# 生成待插值的样本数据
points = np.random.rand(1000,2)
value = ripple(points[:,0]*5,points[:,1]*5)
# 用nearest方法插值
grid_z0 = griddata(points*5,value, (grid_x,grid_y),method='nearest')
我们还可以使用interpolate模块的SmoothBivariateSpline类进行多元仿样插值运算,对图片进行重构。示例代码:
import numpy as np
from scipy.interpolate import SmoothBivariateSpline as SBS
#定义一个函数
def ripple(x,y):
return np.sqrt(x**2 + y**2) + np.sin(x**2 + y**2)
# 生成grid数据,复数定义了生成grid数据的step,若无该复数则step为5
grid_x, grid_y = np.mgrid[0:5:1000j, 0:5:1000j]
# 生成待插值的样本数据
points = np.random.rand(1000,2)
value = ripple(points[:,0]*5,points[:,1]*5)
# 用nearest方法插值
fit = SBS(points[:,0]*5, points[:,1]*5, value, s=0.01, kx=4, ky=4)
interp = fit(np.linspace(0, 5, 1000), np.linspace(0, 5, 1000))
通过反复测试,尽管SmoothBivariateSpline表现略好,但其对给定的样本数据非常敏感,就可能导致忽略一些显著特征。而griddata函数有很强的鲁棒性,不管给定的数据样本,能够合理的进行插值运算。
NumPy库已经提供了一些基本的统计函数,如求期望、方差、中位数、最大值和最小值等。示例代码:
import numpy as np
#构建一个1000个随机变量的数组
x = np.random.randn(1000)
#对数组元素的值进行统计
mean = x.mean()
std = x.std()
var = x.var()
print(mean,std,var)
结果为:
(0.02877273942510088, 0.97623362287515114, 0.95303208643194282)
SciPy的stats模块提供了大约80种连续随机变量和10多种离散分布变量,这些分布都依赖于numpy.random函数。可以通过如下语句获得stats模块中所有的连续随机变量,示例代码:
from scipy import stats
[k for k, v in stats.__dict__.items() if isinstance(v, stats.rv_continuous)]
运行结果为:
['ksone', 'kstwobign', 'norm', 'alpha', 'anglit', 'arcsine', 'beta', 'betaprime', 'bradford', 'burr', 'burr12', 'fisk', 'cauchy', 'chi', 'chi2', 'cosine', 'dgamma', 'dweibull', 'expon', 'exponnorm', 'exponweib', 'exponpow', 'fatiguelife', 'foldcauchy', 'f', 'foldnorm', 'frechet_r', 'weibull_min', 'frechet_l', 'weibull_max', 'genlogistic', 'genpareto', 'genexpon', 'genextreme', 'gamma', 'erlang', 'gengamma', 'genhalflogistic', 'gompertz', 'gumbel_r', 'gumbel_l', 'halfcauchy', 'halflogistic', 'halfnorm', 'hypsecant', 'gausshyper', 'invgamma', 'invgauss', 'invweibull', 'johnsonsb', 'johnsonsu', 'laplace', 'levy', 'levy_l', 'levy_stable', 'logistic', 'loggamma', 'loglaplace', 'lognorm', 'gilbrat', 'maxwell', 'mielke', 'kappa4', 'kappa3', 'nakagami', 'ncx2', 'ncf', 't', 'nct', 'pareto', 'lomax', 'pearson3', 'powerlaw', 'powerlognorm', 'powernorm', 'rdist', 'rayleigh', 'reciprocal', 'rice', 'recipinvgauss', 'semicircular', 'skewnorm', 'trapz', 'triang', 'truncexpon', 'truncnorm', 'tukeylambda', 'uniform', 'vonmises', 'vonmises_line', 'wald', 'wrapcauchy', 'gennorm', 'halfgennorm']
连续随机变量对象主要使用如下方法,下表所示:
| 方法名 | 全称 | 功能 |
| rvs | Random Variates of given type | 对随机变量进行随机取值,通过size参数指定输出数组的大小 |
| Probability Density Function | 随机变量的概率密度函数 | |
| cdf | Cumulative Distribution Function | 随机变量的累积分布函数,它是概率密度函数的积分 |
| sf | Survival function | 随机变量的生存函数,它的值是1-cdf(t) |
| ppf | Percent point function | 累积分布函数的反函数 |
| stats | statistics | 计算随机变量的期望值和方差 |
| fit | fit | 对一组随机取样进行拟合,找出最适合取样数据的概率密度函数的系数 |
from scipy import stats
# 设置正态分布参数,其中loc是期望值参数,scale是标准差参数
X = stats.norm(loc=1.0, scale=2.0)
# 计算随机变量的期望值和方差
print(X.stats())
结果为:
(array(1.0), array(4.0))
此外,通过调用随机变量X的rvs()方法,可以得到包含一万次随机取样值的数组x,然后调用NumPy的mean()和var()计算此数组的均值和方差,其结果符合随机变量X的特性,示例代码:
#对随机变量取10000个值
x = X.rvs(size=10000)
print(np.mean(x), np.var(x))
结果为:
(1.0287787687588861, 3.9944276709242805)
使用fit()方法对随机取样序列x进行拟合,它返回的是与随机取样值最吻合的随机变量参数,示例代码:
#输出随机序列的期望值和标准差
print(stats.norm.fit(x))
结果为:
(1.0287787687588861, 1.998606432223283)
在下面的例子中,计算取样值x的直方图统计以及累计分布,并与随机变量的概率密度函数和累积分布函数进行比较。示例代码:
pdf, t = np.histogram(x, bins=100, normed=True)
t = (t[:-1]+t[1:])*0.5
cdf = np.cumsum(pdf) * (t[1] - t[0])
p_error = pdf - X.pdf(t)
c_error = cdf - X.cdf(t)
print("max pdf error: {}, max cdf error: {}".format(np.abs(p_error).max(), np.abs(c_error).max()))
运行结果如下所示:
max pdf error: 0.0208405611169, max cdf error: 0.0126874590568
通过绘图的方式查看概率密度函数求得的理论值(theory value)和直方图统计值(statistic value),可以看出二者是一致的,示例代码:
import pylab as pl
pl.plot(t, pdf, color="green", label = "statistic value")
pl.plot(t, X.pdf(t), color="yellow", label ="theory value")
pl.legend(loc = "best")
pl.show()
结果见下图所示:
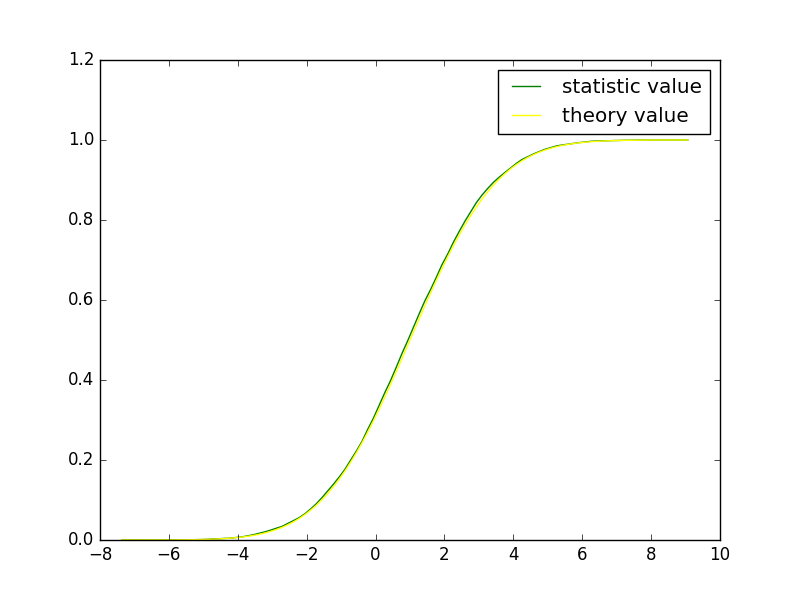
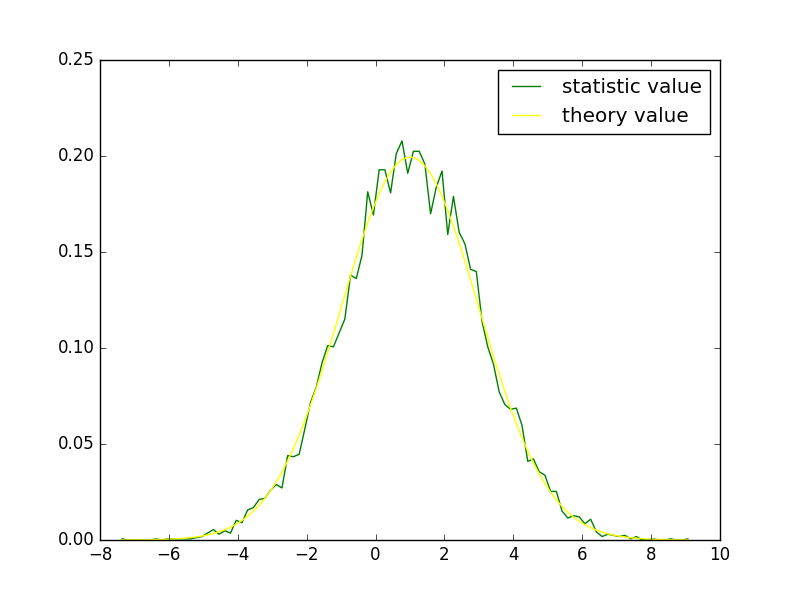 也可以用同样的方式显示随机变量X的累积分布和数组pdf的累加结果,示例代码:
也可以用同样的方式显示随机变量X的累积分布和数组pdf的累加结果,示例代码:import pylab as pl
pl.plot(t, cdf, color="green", label = "statistic value")
pl.plot(t, X.cdf(t), color="yellow", label ="theory value")
pl.legend(loc = "best")
pl.show()
结果为:
 (2)离散概率分布
(2)离散概率分布# 数组x保存骰子的所有可能值,数组p保存每个值出现的概率
x = range(1, 7)
p = (0.4, 0.2, 0.1, 0.1, 0.1, 0.1)
# 创建表示这个骰子的随机变量dice,调用其rvs()方法投掷此骰子20次,获得符合概率p的随机数
dice = stats.rv_discrete(values=(x, p))
print(dice.rvs(size=20))
运行结果:
array([3, 6, 4, 5, 5, 2, 1, 3, 3, 1, 1, 3, 1, 5, 1, 3, 4, 1, 2, 2])
除了自定义离散概率分布,我们也可以利用stats模块里的函数定义各种分布。下面以生成几何分布为例,其函数是geom(),示例代码:
import numpy as np
from scipy.stats import geom
# 设置几何分布的参数
p = 0.5
dist = geom(p)
# 设置样本区间
x = np.linspace(0, 5, 1000)
# 得到几何分布的 PMF 和CDF
pmf = dist.pmf(x)
cdf = dist.cdf(x)
# 生成500个随机数
sample = dist.rvs(500)
(3)描述与检验函数
import numpy as np
from scipy import stats
# 生成包括100个服从正态分布的随机数样本
sample = np.random.randn(100)
# 用normaltest检验原假设
out = stats.normaltest(sample)
print('normaltest output')
print('Z-score = ' + str(out[0]))
print('P-value = ' + str(out[1]))
# kstest 是检验拟合度的Kolmogorov-Smirnov检验,这里针对正态分布进行检验
# D是KS统计量的值,越接近0越好
out = stats.kstest(sample, 'norm')
print('
kstest output for the Normal distribution')
print('D = ' + str(out[0]))
print('P-value = ' + str(out[1]))
# 类似地可以针对其他分布进行检验,例如Wald分布
out = stats.kstest(sample, 'wald')
print('
kstest output for the Wald distribution')
print('D = ' + str(out[0]))
print('P-value = ' + str(out[1]))
SciPy的stats模块中还提供了一些描述函数,如几何平均(gmean)、偏度(skew)、样本频数(itemfreq)等。示例代码
import numpy as np
from scipy import stats
# 生成包括100个服从正态分布的随机数样本
sample = np.random.randn(100)
# 调和平均数,样本值须大于0
out = stats.hmean(sample[sample > 0])
print('Harmonic mean = ' + str(out))
# 计算-1到1之间样本的均值
out = stats.tmean(sample, limits=(-1, 1))
print('
Trimmed mean = ' + str(out))
# 计算样本偏度
out = stats.skew(sample)
print('
Skewness = ' + str(out))
# 函数describe可以一次给出样本的多种描述统计结果
out = stats.describe(sample)
print('
Size = ' + str(out[0]))
print('Min = ' + str(out[1][0]))
print('Max = ' + str(out[1][1]))
print('Mean = ' + str(out[2]))
print('Variance = ' + str(out[3]))
print('Skewness = ' + str(out[4]))
print('Kurtosis = ' + str(out[5]))
参考:简书
