需求
项目中需要求同一个患者在不同时间和不同设备下获取的MRI和CT的公共区域,由于目前项目中使用的配准代码(将MRI配准到CT上)不是开源的,所以只能考虑在配准前找到公共区域后,将MRI和CT的序列截断后重新保存成mhd文件,再看看配准效果是否有提升。
已知:(1)根据训练好的模型可以预测出CT序列和MRI序列上每一层的中线,由此可以求得该层中线上的中点
(2)根据预测出每一层的中线也能拟合出MRI和CT的序列(三维)的中平面
(3)根据配准的变换矩阵,可以求得源MRI与源CT的映射关系
(4)再由(3)中的映射关系,可求得对应的互信息
方案:(1)根据每一层的中点与中平面的相对位置,分别求得CT和MRI得相应位置序列
(2)根据每一层的宽度与上一层之比,分别求得在CT和MRI上的宽度比序列
(3)根据所求互信息的值,求互信息的阈值
-
根据前两种方案中求得的MRI和CT的两个序列,考虑通过计算两个序列的公共部分,求出MRI和CT公共部分。
-
后一种方案根据对应层的互信息,可以根据取互信息的阈值,分别求出MRI和CT截断的首尾端点。
实验
1. 根据以上方案(2)中的方法分别得到CT与MRI的宽度比序列
- 首先需要对二维的MRI和CT进行分割,CT中得到颅骨分割的结果,MRI中得到脑灰质区域的分割结果
- 从二维图像的中心分别向左向右遍历,找到边界
- 右边界 - 左边界即是当前层的宽度
- 分别得到MRI和CT中的相邻层宽度比序列
实现:
import os
import time
import cv2
import matplotlib.pyplot as plt
from glob import glob
import numpy as np
import SimpleITK as sitk
from skimage.measure import label
def readMhd(mhdPath):
'''
根据文件路径读取mhd文件,并返回其内容Array、origin、spacing
'''
itkImage = sitk.ReadImage(mhdPath)
imageArray = sitk.GetArrayFromImage(itkImage) # [Z, H, W]
origin = itkImage.GetOrigin()
spacing = itkImage.GetSpacing()
return imageArray, origin, spacing
def findCenterPoints(image):
'''
:param image: 二维矩阵
:return: 当前层的中点x,y坐标
'''
H = image.shape[0] // 2 # 向下取整
W = image.shape[1] // 2
centerHW = [H, W] # 取整
return centerHW
def otsuEnhance(img_gray, th_begin, th_end, th_step=1):
'''
根据类间方差最大求最适合的阈值
在th_begin、th_end中寻找合适的阈值
'''
assert img_gray.ndim == 2, "must input a gary_img"
max_g = 0
suitable_th = 0
for threshold in range(th_begin, th_end, th_step): # 前闭后开区间
bin_img = img_gray > threshold
bin_img_inv = img_gray <= threshold
fore_pix = np.sum(bin_img)
back_pix = np.sum(bin_img_inv)
if 0 == fore_pix:
break
if 0 == back_pix:
continue
w0 = float(fore_pix) / img_gray.size
u0 = float(np.sum(img_gray * bin_img)) / fore_pix
w1 = float(back_pix) / img_gray.size
u1 = float(np.sum(img_gray * bin_img_inv)) / back_pix
# 类间方差公式
g = w0 * w1 * (u0 - u1) * (u0 - u1)
# print('threshold, g:', threshold, g)
if g > max_g:
max_g = g
suitable_th = threshold
return suitable_th
# def getMeanValue(image):
# '''
# :param image: images中某一层的二维图像
# :return: 图像的平均灰度
# '''
# pixel_val_sum = 0.0
# for i in range(image.shape[0]):
# for j in range(image.shape[1]):
# pixel_val_sum += image[i][j]
# # print(pixel_val_sum)
# return abs(pixel_val_sum / (image.shape[0] * image.shape[1])) # 取均值的绝对值
def closeDemo(image, structure_size):
'''
闭操作:补全细小连接
'''
kenerl = cv2.getStructuringElement(cv2.MORPH_RECT, (structure_size, structure_size)) # 定义结构元素的形状和大小
# dst_1 = cv2.dilate(image, kenerl) # 先膨胀
# dst = cv2.erode(dst_1, kenerl) # 腐蚀操作
dst = cv2.morphologyEx(image, cv2.MORPH_CLOSE, kenerl) # 闭操作,与分开使用膨胀和腐蚀效果相同
return dst
def largestConnectComponent(bw_img):
'''
求bw_img中的最大联通区域
:param bw_img: 二值图
:return: 最大连通区域
'''
labeled_img, num = label(bw_img, connectivity=2, background=0, return_num=True) # 取连通区域,connectivity=2表示8领域
max_label = 0
max_num = 0
for i in range(1, num + 1): # lable的编号从1开始,防止将背景设置为最大连通域
if np.sum(labeled_img == i) > max_num:
max_num = np.sum(labeled_img == i)
max_label = i
lcc = (labeled_img == max_label) # 只留下最大连通区域,其他区域置0
return lcc
def segCTGaryMatter(image, threshold_min=300, threshold_max=1000, jpgSavePath=None, z=0, threshold_step=2, binary_threshold=None):
'''
:param image: 二维的image
:param threshold_min: OTSU的最小阈值300
:param threshold_max: OTSU的最大阈值1000
:param filter_size: 滤波器的大小
:return: 分割好的二维图像,otsu阈值
'''
if binary_threshold is None:
binary_threshold = otsuEnhance(image, threshold_min, threshold_max, threshold_step)
ret, binary = cv2.threshold(image, binary_threshold, 255, cv2.THRESH_BINARY)
image_max_region = largestConnectComponent(binary) # 求img[i]的最大联通区域
# 将bool类型先转为np.uint8(findContours函数中的输入类型)
image_max_region = image_max_region.astype(np.uint8)
# 求外轮廓,避免中间的孔洞被保留,这里只求外轮廓
_, contours, hierarchy = cv2.findContours(image_max_region, cv2.RETR_EXTERNAL, cv2.CHAIN_APPROX_NONE)
new_image = np.zeros(image.shape, np.uint8) * 255 # 创建与原图像等大的黑色幕布
cv2.drawContours(new_image, contours, -1, (255, 255, 255), 3) # contours是轮廓,-1表示全画,然后是轮廓线使用白色,厚度3
# if z == 0:
# # 显示第0层的contours
# print('第0层的contours: ', contours)
# print('第0层的contours的长度: ', len(contours[0]))
# print('第0层将边界画到画布上后的值: ', new_image)
new_image_fill = np.copy(new_image)
cv2.fillPoly(new_image_fill, contours, (255, 255, 255)) # 填充外轮廓孔洞
if jpgSavePath is not None:
# 保存分割结果
saveContoursImage = new_image.astype(np.uint8)
cv2.imwrite(jpgSavePath.replace('.png', '_contours_{}.png'.format(z)), saveContoursImage) # 保存的左右相减的结果
saveFillImage = new_image_fill.astype(np.uint8)
cv2.imwrite(jpgSavePath.replace('.png', '_fill_{}.png'.format(z)), saveFillImage)
# print('new_image_fill_shape: ', new_image_fill.shape) # 512*512
# plt.figure()
# plt.subplot(121)
# plt.title('outer_contours')
# plt.imshow(new_image, cmap='gray')
# plt.subplot(122)
# plt.title('contours_fill')
# plt.imshow(new_image_fill, cmap='gray')
# plt.show()
# plt.close()
# print(new_image_fill.dtype) # uint8
# H, W = np.where(new_image_fill > 0)
# print(len(H)) # 26万
# print(len(W))
return new_image_fill, binary_threshold
def FillHole(im_in):
# 复制 im_in 图像
im_floodfill = im_in.copy()
# Mask 用于 floodFill,官方要求长宽+2
h, w = im_in.shape[:2]
mask = np.zeros((h + 2, w + 2), np.uint8)
seedPoint = (0, 0)
# floodFill函数中的seedPoint对应像素必须是背景
isbreak = False
for i in range(im_floodfill.shape[0]):
for j in range(im_floodfill.shape[1]):
if (im_floodfill[i][j] == 0):
seedPoint = (i, j)
isbreak = True
break
if (isbreak):
break
# 得到im_floodfill 255填充非孔洞值
cv2.floodFill(im_floodfill, mask, seedPoint, 255)
# 得到im_floodfill的逆im_floodfill_inv
im_floodfill_inv = cv2.bitwise_not(im_floodfill)
# 把im_in、im_floodfill_inv这两幅图像结合起来得到前景
im_out = im_in | im_floodfill_inv
return im_out
# 分割MRI
def segMRIGaryMatter(img, threshold_min=0, threshold_max=300, z=0, jpgSavePath=None, structure_size=3, filter_size=3):
# if z <= 4:
# # 解决中文显示问题
# plt.rcParams['font.sans-serif'] = ['SimHei']
# plt.rcParams['axes.unicode_minus'] = False
# plt.title('')
# plt.imshow(img, cmap='gray')
# plt.show()
img[np.where((img > 30) & (img < 80))] = 100 # 填补脑组织暗区
# if z <= 4:
# # 解决中文显示问题
# plt.rcParams['font.sans-serif'] = ['SimHei']
# plt.rcParams['axes.unicode_minus'] = False
# plt.title('填补暗区')
# plt.imshow(img, cmap='gray')
# plt.show()
binary_threshold = otsuEnhance(img, threshold_min, threshold_max, 1) # 使用类间方差来计算合适的阈值
# print('MRI_binary_threshold: ', binary_threshold)
ret, binary = cv2.threshold(img, binary_threshold, 255, cv2.THRESH_BINARY) # 二值化到0-255
# if z <= 4:
# # 解决中文显示问题
# plt.rcParams['font.sans-serif'] = ['SimHei']
# plt.rcParams['axes.unicode_minus'] = False
# plt.title('二值图')
# plt.imshow(binary, cmap='gray')
# plt.show()
# print('二值图的type:', type(binary))
# blur = cv2.medianBlur(binary, filter_size) # 中值滤波去噪,设置滤波器大小为3
#
#
# # 在求最大连通区域前就对图像做一次闭操作,补全细小连接
# img = closeDemo(blur, structure_size)
img = largestConnectComponent(binary) # 求img[i]的最大联通区域
# if z <= 4:
# # 解决中文显示问题
# plt.rcParams['font.sans-serif'] = ['SimHei']
# plt.rcParams['axes.unicode_minus'] = False
# plt.title('最大连通区域')
# plt.imshow(img, cmap='gray')
# plt.show()
curSlice = (img * 255).astype(np.uint8)
oriCurSlice = curSlice.copy()
curSlice = FillHole(curSlice) # 填充孔洞,全部填充成0
curSlice = (curSlice / 255).astype(np.bool) # 先转成bool
curSlice = curSlice.astype(np.float32) # 再转成float32
if jpgSavePath is not None:
# 保存分割结果
saveOriImage = oriCurSlice.astype(np.uint8)
cv2.imwrite(jpgSavePath.replace('.png', '_ori_{}.png'.format(z)), saveOriImage) # 保存填充前的图像
saveFillImage = (curSlice * 255).astype(np.uint8) # 再一次类型转换
cv2.imwrite(jpgSavePath.replace('.png', '_fill_{}.png'.format(z)), saveFillImage)
return curSlice
def findSliceWidth(image, center_point, z):
'''
:param image: 分割脑部组织区域的图像(CT、MRI的处理相同)
:param centerW: 中点:取图像的中点
:param z: 用于测试
:return: 返回边界宽度
'''
H, W = np.where(image > 0) # 都是整数
if len(W) != 512 * 512: # 全黑的部分填充后变成全白,len(W) == 512 * 512
# 分割区域的边界值
w_left = min(W)
w_right = max(W)
# 左部分从中心点反向遍历
l = center_point[1] - 1 # 列号从256往后递减,行号固定到256中间
# print('center_point[0] - 1', center_point[0] - 1, ' l: ', l)
center_H = center_point[0] - 1
while l - 1 >= w_left:
left_cur_point = [center_H, l]
left_next_point = [center_H, l - 1] # pre_point
# 当前点的像素值大于0,下一个点的像素值等于0
if image[left_cur_point[0], left_cur_point[1]] > 0 and image[left_next_point[0], left_next_point[1]] == 0:
break
l -= 1
# left_width
# 右部分从中心点沿着右方向正向遍历
# right_cur_point = [right_w, right_w] # 中点下一个位置
r = center_point[1]
while r < w_right: # 防止下标越界
right_cur_point = [center_H, r]
right_next_point = [center_H, r + 1]
if image[right_cur_point[0], right_cur_point[1]] > 0 and image[
right_next_point[0], right_next_point[1]] == 0:
break # 上一个点是白,下一个是黑,停止查找
r += 1
# right_width
slice_width = r - l # 宽度等于右边界-左边界
else:
return 0 # 全黑的层宽度取0
return slice_width
if __name__ == '__main__':
CT_RotateMhdDir = # 旋转之后检查效果后保存的数据
MRI_RotateMhdDir = # MRI的images文件
CT_jpgSaveDir = # 用于保存,CT分割后的结果的目录
MRI_jpgSaveDir = # 用于保存,MRI分割后的结果的目录
# CT_lineDir = r'C:UsersVickyDesktopwx_03_30CT_line'
# MRI_lineDir = r'C:UsersVickyDesktopwx_03_30MRI_line'
trendChartDir = # 用于保存CT和MRI宽度比趋势图路径
CT_RotateMhdID = os.listdir(CT_RotateMhdDir)
MRI_RotateMhdID = glob(MRI_RotateMhdDir + '\*.mhd')
MRI_RotateMhdID2 = []
#将所有的病例号取出
for i in range(len(MRI_RotateMhdID)):
# print(MRI_RotateMhdID[i].split('.')[0].split('\')[7])
MRI_RotateMhdID2.append(str(MRI_RotateMhdID[i].split('.')[0].split('\')[7]))
# 经过脑部姿态调整通过自检的数据MRI和CT不是一一对应的所以需要取交集
interID = set(CT_RotateMhdID) & set(MRI_RotateMhdID2) # 先转成set再求两组数据取交集
for mhdID in interID:
print(mhdID)
CT_mhdDir = os.path.join(CT_RotateMhdDir, mhdID) # 拼接CT_mhd文件的绝对目录
print('CT_mhdDir: ', CT_mhdDir)
CT_mhdPath = glob(CT_mhdDir + '\*.mhd') # 取mhd文件的路径
MRI_mhdPath = os.path.join(MRI_RotateMhdDir, mhdID + '.mhd')
print('MRImhdPath: ', MRI_mhdPath)
CT_jpgSubDir = os.path.join(CT_jpgSaveDir, mhdID) # 拼接CT_jpg保存的目录
MRI_jpgSubDir = os.path.join(MRI_jpgSaveDir, mhdID)
trendChartSubDir = os.path.join(trendChartDir, mhdID)
if not os.path.exists(CT_jpgSubDir):
os.makedirs(CT_jpgSubDir) # 新建保存CT中间分割结果图片的子文件
if not os.path.exists(MRI_jpgSubDir):
os.makedirs(MRI_jpgSubDir) # 新建保存MRI中间分割结果图片的子文件
if not os.path.exists(trendChartSubDir):
os.makedirs(trendChartSubDir)
CT_jpgSavePath = os.path.join(CT_jpgSubDir, (mhdID + '.png')) # 拼接绝对路径
MRI_jpgSavePath = os.path.join(MRI_jpgSubDir, (mhdID + '.png'))
# print('CT_mhdPath: ', CT_mhdPath)
CT_imageArray, _, _ = readMhd(CT_mhdPath[0]) # 读取原图mhd文件,坐标顺序Z,H,W
MRI_imageArray, _, _ = readMhd(MRI_mhdPath)
CT_slice_width_Array = [] # 用于存放当前病例所有层的宽度
MRI_slice_width_Array = []
for z in range(CT_imageArray.shape[0]):
# 理想条件下,旋转之后的中心==图像的中心
CT_center_point = findCenterPoints(CT_imageArray[z])
seg_CT_image, binary_threshold = segCTGaryMatter(CT_imageArray[z], -500, -300, CT_jpgSavePath,z) # 阈值大致控制在-400
# 计算CT每一层的宽度
CT_cur_slice_width = findSliceWidth(seg_CT_image, CT_center_point, z)
if CT_cur_slice_width != 0: # 当前层有分割结果就存到slice_width_Array数组中
CT_slice_width_Array.append(CT_cur_slice_width)
CT_slice_width_Array = np.array(CT_slice_width_Array) # 类型转换
# 对MRI求每一层的宽度
for z in range(MRI_imageArray.shape[0]):
MRI_center_point = findCenterPoints(MRI_imageArray[z])
seg_MRI_image = segMRIGaryMatter(MRI_imageArray[z], 0, 300, z,
MRI_jpgSavePath) # 阈值大致控制在0-300
# 计算MRI每一层的宽度
MRI_cur_slice_width = findSliceWidth(seg_MRI_image, MRI_center_point, z)
# temp = cur_slice_width
if MRI_cur_slice_width != 0: # 当前层有分割结果就存到slice_width_Array数组中
MRI_slice_width_Array.append(MRI_cur_slice_width)
MRI_slice_width_Array = np.array(MRI_slice_width_Array) # 类型转换
# 对CT相邻层求宽度比
CT_slice_width_ratio_Array = []
CT_slice_num = [] # 横坐标是索引,作图用
for i in range(1, len(CT_slice_width_Array)): # 从第一层开始求比值
# 当前层和上一层的比值
CT_slice_width_ratio_Array.append(
np.float32(CT_slice_width_Array[i]) / CT_slice_width_Array[i - 1])
CT_slice_num.append(i) # 记录索引
#类型转换
CT_slice_width_ratio_Array = np.array(CT_slice_width_ratio_Array)
CT_slice_num = np.array(CT_slice_num)
# CT相邻层宽度比绘图
markes = ['-o', '-^']
plt.figure()
plt.rcParams['font.sans-serif'] = ['SimHei'] # 用来显示中文标签
plt.title('{}'.format(mhdID)) # 标题设置为当前病例号
plt.xlabel("层数", fontsize=15)
plt.ylabel("宽度比", fontsize=15)
plt.plot(CT_slice_num, CT_slice_width_ratio_Array, markes[0], label='CT')
plt.legend()
# 求MRI相邻层宽度比
MRI_slice_width_ratio_Array = []
MRI_slice_num = [] # 横坐标是索引,作图用
for i in range(1, len(MRI_slice_width_Array)): # 从第一层开始求比值
# 当前层和上一层的比值
MRI_slice_width_ratio_Array.append(
np.float32(MRI_slice_width_Array[i]) / MRI_slice_width_Array[i - 1])
MRI_slice_num.append(i) # 记录索引
MRI_slice_width_ratio_Array = np.array(MRI_slice_width_ratio_Array)
MRI_slice_num = np.array(MRI_slice_num)
# 绘制MRI相邻层宽度比绘图
plt.rcParams['font.sans-serif'] = ['SimHei'] # 用来显示中文标签
plt.title('{}'.format(mhdID)) # 标题设置为当前病例号
plt.xlabel("层数", fontsize=15)
plt.ylabel("宽度比", fontsize=15)
plt.plot(MRI_slice_num, MRI_slice_width_ratio_Array, markes[1], label='MRI')
plt.legend() # 用于显示上面的label
plt.savefig('{}/{}.jpg'.format(trendChartSubDir, mhdID)) # 两条曲线画到一张图上
plt.close()
```
效果:

0008数据MRI与CT的相邻层宽度比
上图将MRI和CT的相邻层宽度比绘制到一幅图像上,可以看出CT的相邻层宽度比值一直接近于1,MRI的宽度比值的跨度很大,我们想利用这两个序列找到其中的公共区域,根据当前结果来看不存在公共区域
存在的问题:MRI使用传统方法分割是灰度值较低且MRI拍摄时没有颅骨边界,得到的区域并不准确,导致宽度信息不准确,而CT由于颅骨的灰度值较高使用传统方法能够分割的较好

0008数据CT的分割结果
其中contours表示颅骨外边界,fill表示对外边界的填充,对于部分层数据比如其中的第0层没有求出外边界,导致填充时对整幅图像都进行了填充,所以对于这些曾的宽度取零,避免除0操作,检查了大部分数据的分割结果一般都是在第一层会出现此情况(前面层拍摄信息比较少), 在求宽度比使用当前层宽度比上下一层宽度

0008数据MRI分割结果
其中的ori是直接求连通区域的结果,fill是对连通区域孔洞进行填充后的结果,对于MRI中脑灰质的灰度值较低,考虑先将暗区值变大后再做最大连通区域提取,最后对连通区域中的孔洞进行填充,以上结果表明此方案并不能准确地分割出MRI,考虑原因还是在对于MRI中中间几层效果还好,前后的层脑灰质组织并不连通,直接求连通区域就去掉了大部分的区域
对于此问题后续没有想到更好分割MRI的方法,所以该方案实验没有继续进行
2. 根据以上方案(3)中的方法根据互信息求CT和MRI的公共区域
- 另外的小组在进行配准时会得到相应的变换矩阵,根据变换矩阵反映射可以得到从MRI配准到CT之后(配准后MRI和CT的层数一致),对应的配准后的MRI对应在源MRI上是哪一层
- 将配准小组求得的源MRI 与 源CT的对应层,求他们的互信息
- 求互信息的阈值,将CT和MRI的数据中前面层的互信息小于这个阈值和后面的层互信息大于这个阈值的都去掉,重新保存成mhd文件
- 将以上截断后的MRI和CT数据用于配准,看能否提升配准的效果(由于现在配准的代码并不开源,所以暂时想到此方法用于提升配准效果)
测试一:刚开始使用配准好的MRI和源CT求互信息,截断CT再去配准。考虑是将MRI配准到CT,最后MRI的信息应该跟CT保持一致,所以将互信息大的CT的那些层才用于配准,能改善配准的效果
实现:
import os
import time
import cv2
import matplotlib.pyplot as plt
from glob import glob
import numpy as np
import SimpleITK as sitk
from skimage.measure import label
import sklearn.metrics as skm
def readMhd(mhdPath):
'''
根据文件路径读取mhd文件,并返回其内容Array、origin、spacing
'''
itkImage = sitk.ReadImage(mhdPath)
imageArray = sitk.GetArrayFromImage(itkImage) # [Z, H, W]
origin = itkImage.GetOrigin()
spacing = itkImage.GetSpacing()
return imageArray, origin, spacing
def saveMhd(imageArray, origin, spacing, savePath):
'''
根据图像Array、origin、spacing构造itk image对象,并将其保存到savePath
'''
itkImage = sitk.GetImageFromArray(imageArray, isVector=False)
itkImage.SetSpacing(spacing)
itkImage.SetOrigin(origin)
sitk.WriteImage(itkImage, savePath)
def mutInfo(x, y):
'''
直接根据sklearn库中的函数计算互信息
'''
return skm.mutual_info_score(x, y)
if __name__ == '__main__':
# 尝试方案:利用每一层的互信息
CT_RotateMhdDir = r'C:UsersVickyDesktopwx_03_30�405step2_CT_rotate_result' # 旋转之后检查效果后保存的CT数据
MRI_RegistMhdDir = r'C:UsersVickyDesktopwx_03_30MRIMRI2CT_check_result' # 从MRI到CT之后的配准图像
saveNewCTMhdDir = r'C:UsersVickyDesktopwx_03_30
ewCT' # 取完公共区域重新保存的mhd文件
# CT_jpgSaveDir = r'C:UsersVickyDesktopwx_03_30CT_rotate_seg_png' # 用于保存,CT分割后的结果的目录
# MRI_jpgSaveDir = r'C:UsersVickyDesktopwx_03_30MRI_rotate_seg_png'
# CT_lineDir = r'C:UsersVickyDesktopwx_03_30CT_line'
# MRI_lineDir = r'C:UsersVickyDesktopwx_03_30MRI_line'
# trendChartDir = r'C:UsersVickyDesktopwx_03_30 rendChart_0408' # 用于保存CT和MRI趋势图的路径
testChartDir = r'C:UsersVickyDesktopwx_03_30 est_Chart'
CT_RotateMhdID = os.listdir(CT_RotateMhdDir)
MRI_RotateMhdID = os.listdir(MRI_RegistMhdDir) # 文件结果中有子目录
# print('CT_RotateMhdID: ', CT_RotateMhdID)
# print()
# print('MRI_RotateMhdID: ', MRI_RotateMhdID)
# print()
interID = set(CT_RotateMhdID) & set(MRI_RotateMhdID) # 先转成set再求两组数据取交集
# print('ID交集:', interID, ' 交集长度: ', len(interID)) # 交集有36个文件,0008病例原图没有info信息,配准之后就只有35套
for mhdID in interID:
print(mhdID)
CT_mhdPath = os.path.join(CT_RotateMhdDir, mhdID, mhdID + '.mhd') # 拼接CT_mhd文件的绝对目录
CT_saveSubDir = os.path.join(saveNewCTMhdDir, mhdID)
if not os.path.exists(CT_saveSubDir):
os.makedirs(CT_saveSubDir)
# print('CT_mhdDir: ', CT_mhdPath)
MRI_mhdPath = os.path.join(MRI_RegistMhdDir, mhdID, mhdID + '.mhd') # 拼接MRI_mhd文件的绝对目录
# print('MRI_mhdDir: ', MRI_mhdPath)
trendChartSubDir = os.path.join(testChartDir, mhdID)
# print('trendChartSubDir: ', trendChartSubDir)
if not os.path.exists(trendChartSubDir):
os.makedirs(trendChartSubDir) # 新建图表子目录
CT_imageArray, origin, spacingXYZ = readMhd(CT_mhdPath) # 读取原图mhd文件,坐标顺序Z,H,W
print('a test 46')
MRI_imageArray, _, _ = readMhd(MRI_mhdPath)
mutInfoArray = [] # 用于存放CT和MRI当前病例的互信息
curSliceNums = [] # 用于存放当前层数——绘图
delSliceNums = [] # 用于存放需要截断的层
saveSliceNums = []
print('CT层数: ', CT_imageArray.shape[0])
# 取CT和MRI的最小层数,配准后依然保持的CT的信息
for z in range(CT_imageArray.shape[0]):
x = np.reshape(CT_imageArray[z], -1) # 先将当前层的图像矩阵转成一维向量
y = np.reshape(MRI_imageArray[z], -1)
# 计算当前层的互信息
curSliceMutInfo = mutInfo(x, y)
mutInfoArray.append(curSliceMutInfo)
curSliceNums.append(z)
if curSliceMutInfo < 0.8: # 互信息小于0.8的层删掉
delSliceNums.append(z)
else:
saveSliceNums.append(z)
print('删除的层: ', delSliceNums, 'length: ', len(delSliceNums))
print('保留的层: ', saveSliceNums, 'length: ', len(saveSliceNums))
minSliceNum = min(saveSliceNums)
maxSliceNum = max(saveSliceNums)
print('最小层: ', minSliceNum)
print('最大层: ', maxSliceNum)
newCTImageArray = CT_imageArray[minSliceNum:maxSliceNum + 1, :, :] # 前闭后开区间,少保留了一层
CT_savePath = os.path.join(CT_saveSubDir, mhdID + '.mhd')
# 保存新的mhd文件
saveMhd(newCTImageArray, origin, spacingXYZ, CT_savePath)
# 循环结束求出当前两个病例所有层的互信息
mutInfoArray = np.array(mutInfoArray) # 类型转换
curSliceNums = np.array(curSliceNums)
# print('互信息数组长度: ', len(mutInfoArray)) # 应该跟当前病例层数差不多
# 互信息绘图
markes = ['-^']
plt.figure()
plt.rcParams['font.sans-serif'] = ['SimHei'] # 用来显示中文标签
plt.title('{}'.format(mhdID)) # 标题设置为当前病例号
plt.xlabel("当前层数", fontsize=20)
plt.ylabel("互信息", fontsize=20)
plt.plot(curSliceNums, mutInfoArray, markes[0], label='CT&MRI_mutInfo')
plt.legend()
plt.savefig('{}/{}.jpg'.format(testChartDir, mhdID))
# if mhdID == '0002' or mhdID == '0007':
# plt.show()
plt.show()
plt.close()
效果:根据测试一的想法实现的结果可视化出来,大部分都如下图,从图中可以看到在中间层配准之后的互信息较大,在数据的前侧和后侧互信息都比较低,根据这个特点取互信息阈值为0.8,用于截断CT,将新数据给另外的小组进行配准,主要观察对于之前配准效果不好的几个数据,并没有改善
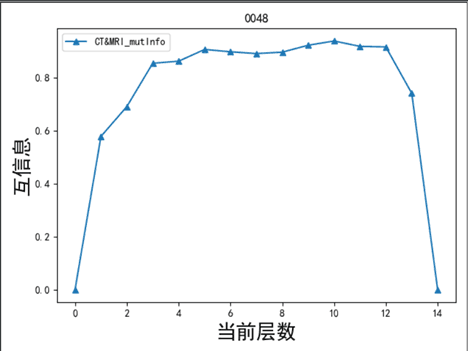
计算配准后的MRI和源CT之间的互信息
考虑原因:主要考虑在配准后,MRI的信息都与CT的保持一致,MRI有些层是根据配准时相邻层间的插值得到,因此根据配准之后再计算互信息得到的结果不具有说服力
测试二:考虑将配准之后的MRI与没配准之前的源MRI找到对应层的映射关系,求源MRI和源CT的互信息
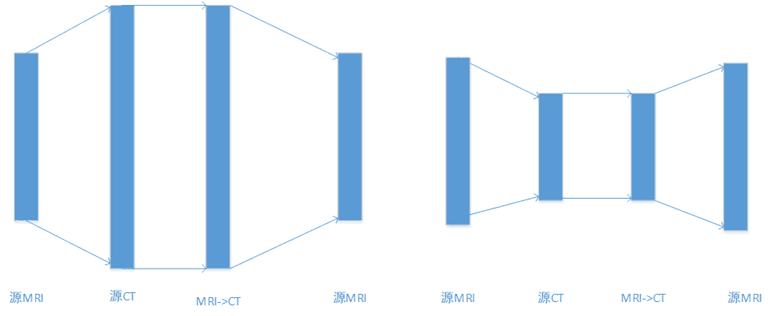
MRI配准到CT上之后层间映射关系
由于CT数据和MRI数据的层数每个数据都不是固定的,所以求源MRI与源CT对应关系时,存在以上两种情况,源MRI与源CT可能多对一、也可能一对多(对于数据中多层对应一层的情况,跳过当前层,取下一层)
实现:
import SimpleITK as sitk
import numpy as np
import matplotlib.pyplot as plt
from glob import glob
import os
import shutil
import json
import sklearn.metrics as skm
import skimage.transform as transform
def readMhd(mhdPath):
'''
根据文件路径读取mhd文件,并返回其内容Array、origin、spacing
'''
itkImage = sitk.ReadImage(mhdPath)
imageArray = sitk.GetArrayFromImage(itkImage) # [Z, H, W]
origin = itkImage.GetOrigin()
spacing = itkImage.GetSpacing()
return imageArray, origin, spacing
def saveMhd(imageArray, origin, spacing, savePath):
'''
根据图像Array、origin、spacing构造itk image对象,并将其保存到savePath
'''
itkImage = sitk.GetImageFromArray(imageArray, isVector=False)
itkImage.SetSpacing(spacing)
itkImage.SetOrigin(origin)
sitk.WriteImage(itkImage, savePath)
def read(file, origin=None, type=sitk.sitkFloat32):
img = sitk.ReadImage(file, type)
if not origin == None:
# print(origin)
img.SetOrigin(origin)
# img.SetSpacing((1,1,1))
array = sitk.GetArrayFromImage(img)
spacing = img.GetSpacing()
size = img.GetSize()
origin = img.GetOrigin()
# size = np.flipud(size)
return array, spacing, size, origin
################
def readMatrix(matrixFile):
lines = []
with open(matrixFile, 'r') as f:
line = f.readline()
while line:
line = line.strip()
line = line.split(" ")
line = list(map(eval, line))
lines.append(line)
# print(line)
line = f.readline()
matrix = np.array(lines, dtype=np.float32)
# print(matrix)
return matrix
def sliceTransfer(matrix, slice, x=256, y=256, mri_spacing=(1, 1, 1), ct_spacing=(1, 1, 1)):
# 层数转换对应公式
# print(matrix[2, 2], matrix[2, 3])
# 这里matrix[2, 2]参数影响不大,四舍五入、向上取整、向下取整有影响
return round(
(matrix[2, 0] * ct_spacing[0] * x + matrix[2, 1] * ct_spacing[1] * y + matrix[2, 2] * slice * ct_spacing[2] + matrix[2, 3]) /
mri_spacing[2])
def json_data(json_path, file):
with open(json_path, 'rt') as f:
jsondata = json.load(f)
return jsondata[file]["centerZYX"]
###########################################
### matrix_path resultMatrix.txt 对应路径
### ct_path CT 对应路径
### mri_path MRI_对应路径
def calSlice(matrix_path, ct_path, mri_path, json_path=None):
'''
:param matrix_path: 对应变换矩阵参数文件的路径
:param ct_path: 源CT(姿态调整之后)的路径
:param mri_path: 源MRI(姿态调整之后)的路径
:param json_path:
:return:
'''
matrix = readMatrix(matrix_path)
mri_array, mri_spacing, mri_sizes, mri_origin = read(mri_path)
ct_array, ct_spacing, ct_sizes, ct_origin = read(ct_path)
# matrix = np.linalg.inv(matrix)
ct_slices = [slice for slice in range(ct_sizes[2])]
if json_path is None:
mri_slices = [sliceTransfer(matrix, slice, ct_sizes[0], ct_sizes[1], mri_spacing, ct_spacing)-1 for slice in
ct_slices]
else:
file = mri_path.split("\")[-1]
xy = json_data(json_path, file)
x = xy[1]
y = xy[2]
print(xy)
mri_slices = [sliceTransfer(matrix, slice, x, y) for slice in ct_slices]
start = -1
end = -1
for i in range(len(ct_slices)):
if mri_slices[i] >= 0 and start == -1:
start = i
if mri_slices[i] <= mri_array.shape[0] - 1:
end = i
print('CT_slices: ', ct_slices[start:end + 1], 'MRI_slices: ', mri_slices[start:end + 1])
return ct_slices[start:end + 1], mri_slices[start:end + 1]
def segMRICTBySlices(CTArray, MRIArray, CTSlices, MRISlices, testChartDir):
'''
:param CTArray: CT三维矩阵
:param MRIArray: MRI三维矩阵
:param CTSlices: CT对应层
:param MRISlices: MRI对应层
:return: 根据互信息,截断之后的MRI和CT
'''
# MRI和CT的shape可能都不同
print('MRI_shape: ', MRIArray.shape)
print('CT_shape: ', CTArray.shape)
# MRIArray = transform.resize(MRIArray, (MRIArray.shape[0], CTArray.shape[1], CTArray.shape[2]), order=0,
# mode='constant', cval=MRIArray.min(),
# anti_aliasing=True, preserve_range=True)
# print('MRI_shape2: ', MRIArray.shape)
sliceLength = len(CTSlices) # 两个的长度是相同的
newCTImageArray = np.zeros(CTArray.shape)
newMRIImageArray = np.zeros(MRIArray.shape)
for z in range(sliceLength):
curCTSlice = CTSlices[z]
curMRISlice = MRISlices[z]
imageCT = CTArray[curCTSlice]
imageMRI = MRIArray[curMRISlice]
x = np.array(np.reshape(imageCT, -1)) # 先将当前层的图像矩阵转成一维向量
y = np.array(np.reshape(imageMRI, -1))
# print('shape: ', x.shape, ' ', y.shape)
# print('length: ', len(x), ' ', len(y))
# 计算互信息两个向量必须是相同大小
tempY = np.zeros(x.shape) # 不够长度的补0
tempY[:len(y)] = y
curSliceMutInfo = skm.mutual_info_score(x, tempY)
# curSliceMutInfo = myMutInfo(x, tempY, scale)
mutInfoArray.append(curSliceMutInfo)
curCTSliceNums.append(curCTSlice)
mutInfoArray = np.array(mutInfoArray)
print('sliceLength: ', sliceLength)
maxMutualInfo = max(mutInfoArray)
# 初始化
CTMinSliceNum = CTSlices[0]
MRIMinSliceNum = MRISlices[0]
if mutInfoArray[0] < 0.2: # 前端结点刚开始的互信息大于0.2,直接取第一个结点截断
for i in range(sliceLength - 1):
if mutInfoArray[i] > maxMutualInfo * 0.7:
# 判断是否CT->MRI是一对多的情况
if MRISlices[i] == MRISlices[i + 1]:
i = i - 1 # 不取当前这一层
break
else:
# 虽然没有大于最大值*0.7 但是当前互信息大于0.125 针对0036数据处理
if mutInfoArray[i] >= 0.125:
break
CTMinSliceNum = CTSlices[i]
MRIMinSliceNum = MRISlices[i]
# 初始化后部分端点索引
CTMaxSliceNum = CTSlices[sliceLength - 1]
MRIMaxSliceNum = MRISlices[sliceLength - 1]
if mutInfoArray[sliceLength - 1] < 0.1: # 如果结尾值小于0.1才使用0.7*max判断
j = sliceLength - 1
while j > 0:
if mutInfoArray[j] > maxMutualInfo * 0.7:
if MRISlices[j] == MRISlices[j - 1]: # 一对三的情况只会出现在CT 36 层 -> MRI 12层的情况
j = j + 1
break
else:
if mutInfoArray[j] > 0.2: # 针对0047这种病例处理
break
j = j - 1
CTMaxSliceNum = CTSlices[j]
MRIMaxSliceNum = MRISlices[j]
else:
# 截断CT
print('CT最小层: ', CTMinSliceNum, 'CT最大层: ', CTMaxSliceNum)
newCTImageArray = CTArray[CTMinSliceNum:CTMaxSliceNum + 1, :, :] # 前闭后开区间
# 截断MRI
print('MRI最小层: ', MRIMinSliceNum, 'MRI最大层: ', MRIMaxSliceNum)
newMRIImageArray = MRIArray[MRIMinSliceNum:MRIMaxSliceNum + 1, :, :]
constValue = []
constValue1 = []
constValue2 = []
for p in range(sliceLength):
constValue.append(np.mean(mutInfoArray))
constValue1.append(maxMutualInfo * 0.8)
constValue2.append(maxMutualInfo * 0.7)
# 互信息绘图
markes = ['-^', '-x', '-o', '-*']
plt.figure()
# plt.rcParams['font.sans-serif'] = ['SimHei'] # 用来显示中文标签
plt.title('{}'.format(mhdID)) # 标题设置为当前病例号
plt.xlabel("CTSlices", fontsize=20)
plt.ylabel("mutualInfo", fontsize=20)
plt.plot(curCTSliceNums, mutInfoArray, markes[0], label='CT&MRI_mutualInfo')
plt.xticks(curCTSliceNums)
plt.plot(curCTSliceNums, constValue1, markes[3], color='red', label='MaxMutualValue*0.8')
plt.plot(curCTSliceNums, constValue, markes[2], color='green', label='MeanMutualValue')
plt.plot(curCTSliceNums, constValue2, markes[1], color='black', label='MaxMutualValue*0.7')
plt.legend()
plt.savefig('{}/{}.jpg'.format(testChartDir, mhdID))
# if mhdID == '0002' or mhdID == '0007':
# plt.show()
plt.show()
plt.close()
return newCTImageArray, newMRIImageArray
if __name__ == "__main__":
CTDir = # CT旋转通过检查的数据
MRIDir = # MRI通过检查的数据——目录未按照单个病例放一个目录
transMatrixDir = # 变换矩阵参数保存到txt文件中
CTSegResultDir = # 存放截断后的CT目录
MRISegResultDir = # 存放截断后的MRI目录
testChartDir = # 保存互信息绘图
# 求MRI数据和CT数据交集
CTMhdID = os.listdir(CTDir)
# MRIMhdID = os.listdir(MRIDir)
MRIMhdPath = glob(MRIDir + '/*.mhd') # mask->seg_mhd,会包含mask的mhd,可以通过交集去掉
MRIMhdID = []
for mriPath in MRIMhdPath:
MRIMhdID.append(os.path.basename(mriPath).split('.')[0])
interMhdID = list(set(CTMhdID) & set(MRIMhdID))
# print('交集的长度: ', len(interMhdID), '交集的序号: ', interMhdID)
count = 0
for mhdID in interMhdID:
print('ID: ', mhdID)
# 拼接单个MRI和CT的路径
mhdName = mhdID + '.mhd'
ctMhdDir = os.path.join(CTDir, mhdID)
ctMhdPath = os.path.join(ctMhdDir, mhdName)
# print('CT: ', ctMhdPath)
mriMhdPath = os.path.join(MRIDir, mhdName)
# print('MRI: ', mriMhdPath)
transMatrixfile = 'resultMatrix.txt'
transMatrixPath = os.path.join(transMatrixDir, mhdID, transMatrixfile)
# print('transMatrix: ', transMatrixPath)
CTArray, CTorigin, CTspacing = readMhd(ctMhdPath)
MRIArray, MRIorigin, MRIspacing = readMhd(mriMhdPath)
# 返回源MRI和源CT的对应层
CTSlices, MRISlices = calSlice(transMatrixPath, ctMhdPath, mriMhdPath)
newCTImage, newMRIImage = segMRICTBySlices(CTArray, MRIArray, CTSlices, MRISlices, testChartDir)
CTSegResultSubDir = os.path.join(CTSegResultDir, mhdID)
if not os.path.exists(CTSegResultSubDir):
os.makedirs(CTSegResultSubDir)
MRISegResultSubDir = os.path.join(MRISegResultDir, mhdID)
if not os.path.exists(MRISegResultSubDir):
os.makedirs(MRISegResultSubDir)
newCTPath = os.path.join(CTSegResultSubDir, mhdName)
newMRIPath = os.path.join(MRISegResultSubDir, mhdName)
# 保存截断后的CT MRI的mhd文件
saveMhd(newCTImage, CTorigin, CTspacing, newCTPath)
saveMhd(newMRIImage, MRIorigin, MRIspacing, newMRIPath)
count = count + 1
print('处理数据的个数 ', count)
效果:使用以上方案截断CT和MRI后再用于配准,通过手工检查配准的数据,相较之前的配准效果来看,对于位置的细节信息,此方案有所改善。
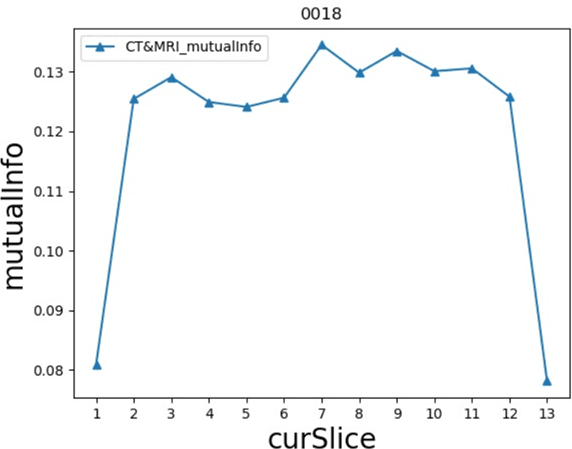
0018数据根据源CT和源MRI对应层求得的互信息

根据求得的互信息,手工检查将配准后的MRI叠加到CT上的数据,主要还是考虑去掉前后互信息较小的层,保留中间层(病灶也大都处于该区域)
根据以上方案求得的互信息,主要像以下两幅图所示,根据下图求互信息阈值,首先简单考虑能更多地保留中间层区域,将阈值取为互信息最大值 * 0.8、互信息最大值 * 0.7 、互信息均值绘制到互信息的图像上,比较分析后取互信息最大值 * 0.7作为阈值用于求取截断CT和MRI的端点,边界部分对于不同数据特殊处理。
实验至此,对于互信息阈值的选取方法考虑可以使用梯度等信息进行改善,但考虑其效果还需和别的方案效果做对比才能最终决定是否选取该方案,所以暂时没有优化。
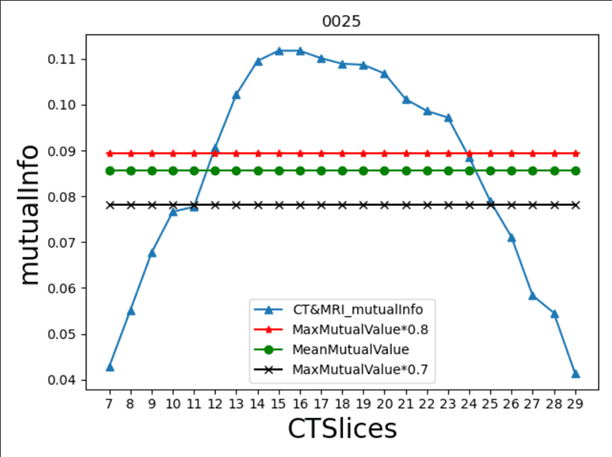
0025数据源MRI和源CT对应层互信息

0021数据源MRI和源CT对应层互信息
3.根据以上方案(1)求MRI和CT的公共区域
- 求MRI和CT数据中,每一层中点与中平面的相对位置(左为a右为b)得到MRI和CT上的字符串
- 根据最长公共子串算法(LCS)求出两序列的公共子串,得到相应的MRI和CT的端点
- 根据以上所求端点用于截断MRI和CT
测试一:考虑我们已经通过模型预测得到MRI和CT每一层的中线,我们可以通过这个中线求每一层的中点,通过我们对MRI和CT旋转之前拟合的中平面,可以得到每一层中点与中平面的位置序列
将中平面可视化到三维的MRI和CT上,在二维数据上来看每一层上就是一条直线,可以通过计算当前层的中点与直线对应的位置关系(将中点的坐标带入直线的方程,大于0、小于0、等于0看作三种位置关系,用不同字符表示)
实现:
import os
import time
import cv2
import json
import matplotlib.pyplot as plt
from glob import glob
import numpy as np
import SimpleITK as sitk
import scipy
import scipy.ndimage
import skimage.transform as transform
from sklearn.cluster import KMeans
from sklearn import linear_model
from skimage.measure import label
import sklearn.metrics as skm
def readMhd(mhdPath):
'''
根据文件路径读取mhd文件,并返回其内容Array、origin、spacing
'''
itkImage = sitk.ReadImage(mhdPath)
imageArray = sitk.GetArrayFromImage(itkImage) # [Z, H, W]
origin = itkImage.GetOrigin()
# spacing = np.flipud(itkImage.GetSpacing()) # [X,Y,Z] -> [Z,Y,X]
spacing = itkImage.GetSpacing()
return imageArray, origin, spacing
def saveMhd(imageArray, origin, spacing, savePath):
'''
根据图像Array、origin、spacing构造itk image对象,并将其保存到savePath
'''
itkImage = sitk.GetImageFromArray(imageArray, isVector=False)
itkImage.SetSpacing(spacing)
itkImage.SetOrigin(origin)
sitk.WriteImage(itkImage, savePath)
def getCenterPoints(mask2DArray):
'''
根据预测的中线计算当前层中点
:param imageArray:二维的Mask数组
:return:每一层二维mask的中点
'''
totalYX = np.where(mask2DArray == 1)
if len(totalYX[0]) == 0 or len(totalYX[1]) == 0: # 当前层没有预测结果的mask, 返回None
return None
else:
centerPoint = [int(np.mean(totalYX[0])), int(np.mean(totalYX[1]))]
return centerPoint
def drawPlane(image, w, b, value, scale=1):
'''
将平面:w[0] * z + w[1] * y + w[2] * x + b = 0画到三维图像中
image: 图像数组, 会被修改, 坐标顺序是ZYX
w、b: w为平面的法向量,b为平面方程的截距
value: 用来表达平面的像素值
scale:采样倍率,越大,所画出来的点越密集,防止由于离散点从而在平面中出现空洞
返回画上了中线的image
从三种轴面画中线(避免除0操作,取该轴向系数最大的)
'''
imageShape = image.shape # imageShape
absW = np.abs(w)
maxAxis = np.argmax(absW) # 取最大值的索引
if maxAxis == 0: # 如果最大值在索引0处取得
coordY, coordX = np.mgrid[
range(int(np.round(imageShape[1] * scale))), range(int(np.round(imageShape[2] * scale)))] # 生成网格
coordY = coordY.astype(np.float32)
coordX = coordX.astype(np.float32)
coordY /= scale
coordX /= scale
coordZ = -(w[1] * coordY + w[2] * coordX + b) / w[0]
elif maxAxis == 1: # 如果最大值在索引1取得
coordZ, coordX = np.mgrid[
range(int(np.round(imageShape[0] * scale))), range(int(np.round(imageShape[2] * scale)))]
coordZ = coordZ.astype(np.float32)
coordX = coordX.astype(np.float32)
coordZ /= scale
coordX /= scale
coordY = -(w[0] * coordZ + w[2] * coordX + b) / w[1]
else: # 如果最大值在索引2取得
coordZ, coordY = np.mgrid[
range(int(np.round(imageShape[0] * scale))), range(int(np.round(imageShape[1] * scale)))]
coordZ = coordZ.astype(np.float32)
coordY = coordY.astype(np.float32)
coordZ /= scale
coordY /= scale
coordX = -(w[0] * coordZ + w[1] * coordY + b) / w[2]
coordZ = np.round(coordZ).astype(np.int)
coordY = np.round(coordY).astype(np.int)
coordX = np.round(coordX).astype(np.int)
# 将list变为一维向量
coordZ = coordZ.reshape(-1)
coordY = coordY.reshape(-1)
coordX = coordX.reshape(-1)
# 筛选合法的坐标位置
validIndicesZ = np.logical_and(coordZ >= 0, coordZ < imageShape[0])
validIndicesY = np.logical_and(coordY >= 0, coordY < imageShape[1])
validIndicesX = np.logical_and(coordX >= 0, coordX < imageShape[2])
validIndices = np.logical_and(validIndicesZ, np.logical_and(validIndicesY, validIndicesX))
coordZ = coordZ[validIndices]
coordY = coordY[validIndices]
coordX = coordX[validIndices]
image[coordZ, coordY, coordX] = value
# def neiborExpand(imageArray, row, col, pixelValue):
# '''
# 对中点进行8邻域扩充,一个像素点在图像上并不明显,考虑扩充其8邻域再画到原图上
# :param imageArray: 当前层
# :param row:
# :param col:
# :param pixelValue: 设置该点及邻域的像素值
# :return: 无返回值,直接在原图上修改
# '''
# Neighbors = [(1, 1), (1, -1), (1, 0), (-1, 0), (-1, 1), (-1, -1), (0, 1), (0, -1)]
# imageArray[row][col] = pixelValue
# for neighbor in Neighbors:
# dr, dc = neighbor
# try:
# imageArray[row + dr][col + dc] = pixelValue # 将其8邻域都设置为9000
# except IndexError:
# pass
def drawCenterPoints(imageArray, maskArray, pixelValue):
sliceNum = imageArray.shape[0]
# newImageArray = imageArray.copy() # 避免修改原图
for z in range(sliceNum):
centerPoints = getCenterPoints(maskArray[z])
if centerPoints is not None: # 如果中点为none不处理
# 在原图上将中点画上,直接修改的是原图,无返回值
imageArray[z, centerPoints[0], centerPoints[1]] = pixelValue
if __name__ == '__main__':
# 画中线与中点应该画在未进行脑部字条调整的数据上
CTDataDir = # 该目录下包含未旋转的CT源数据以及CT逐层预测的mask
MRIImageDataDir = # MRI源数据
MRImaskDataDir = # MRI逐层预测的mask
CTplaneParamsDictPath = # CT平面参数json文件
MRIplaneParamsDictPath = # MRI平面参数json文件
newCTMhd = # CT画上中线和中点后的保存的目录
newMRIMhd = # MRI画上中线和中点后保存的目录
# 读取CT平面参数json文件
with open(MRIplaneParamsDictPath, 'r') as fCT:
CTplaneParamsDict = json.load(fCT)
# 读取的MRI的平面参数json文件
with open(MRIplaneParamsDictPath, 'r') as fMRI:
MRIplaneParamsDict = json.load(fMRI)
CTMhdID = os.listdir(CTDataDir)
# CT 和 MRI 数据病例号一一对应
for mhdID in CTMhdID:
print('mhdID: ', mhdID)
mhdName = mhdID + '.mhd'
# 拼接CTImageMhdPath 和 CTMaskMhdPath
CTMaskMhdName = mhdID + '_mp.mhd'
CTImageMhdPath = os.path.join(CTDataDir, mhdID, mhdName)
CTMaskMhdPath = os.path.join(CTDataDir, mhdID, CTMaskMhdName)
# 拼接MRIImageMhdPath 和 MRIMaskMhdPath
MRIImageMhdPath = os.path.join(MRIImageDataDir, mhdName)
MRIMaskMhdPath = os.path.join(MRImaskDataDir, mhdName)
wCT = CTplaneParamsDict[mhdName]['w']
bCT = CTplaneParamsDict[mhdName]['b']
wMRI = MRIplaneParamsDict[mhdName]['w']
bMRI = MRIplaneParamsDict[mhdName]['b']
# 读取的CT MRI 原图像数组
CTImageArray, CTOrigin, CTSpacing = readMhd(CTImageMhdPath)
MRIImageArray, MRIOrigin, MRISpacing = readMhd(MRIImageMhdPath)
# 读取的CT MRI mask数组
CTMaskArray, _, _ = readMhd(CTMaskMhdPath)
MRIMaskArray, _, _ = readMhd(MRIMaskMhdPath)
drawPlane(CTImageArray, wCT, bCT, 9999)
drawCenterPoints(CTImageArray, CTMaskArray, 9999)
drawPlane(MRIImageArray, wMRI, bMRI, 1000)
drawCenterPoints(MRIImageArray, MRIMaskArray, 1000)
# 将经过修改的图像数据重新保存
#拼接MRI CT的保存mhdPath
CTSaveMhdDir = os.path.join(newCTMhd, mhdID)
MRISaveMhdDir = os.path.join(newMRIMhd, mhdID)
if not os.path.exists(CTSaveMhdDir):
os.makedirs(CTSaveMhdDir)
if not os.path.exists(MRISaveMhdDir):
os.makedirs(MRISaveMhdDir)
CTSaveMhdPath = os.path.join(CTSaveMhdDir, mhdName)
MRISaveMhdPath = os.path.join(MRISaveMhdDir, mhdName)
saveMhd(CTImageArray, CTOrigin, CTSpacing, CTSaveMhdPath)
saveMhd(MRIImageArray, MRIOrigin, MRISpacing, MRISaveMhdPath)
根据中线拟合的中平面的参数都保存到json文件中,利用中平面的参数将中平面画到每一层上,根据预测得到的mask求出的中点也一并画上,通过可视化的方法检查和最终求得的序列是否一致
得到每一层上的中线和中点,求相应的序列就比较简单了,可以根据中线上任意两点得到中线的方程,再将中点坐标带入得到位置关系,以下是求MRI和CT上相应位置序列的核心代码
def generalEquation(first_x, first_y, second_x, second_y):
# 一般式 Ax+By+C=0
A = second_y - first_y
B = first_x - second_x
C = second_x * first_y - first_x * second_y
return A, B, C
def getCTSequence(midLineArray, maskArray):
sliceNum = midLineArray.shape[0]
sequence = []
for z in range(sliceNum):
centerPoints = getCenterPoints(maskArray[z])
if centerPoints is not None: # 如果中点为none不处理
row, col = np.where(midLineArray[z] == 9999)
# 取中线的起始两点确定中线的方程
rowMin = min(row) # 行最小
rowMax = max(row) # 行最大
# 找到行标对应的索引
rowMinIndex = row.tolist().index(rowMin)
rowMaxIndex = row.tolist().index(rowMax)
# 根据行标求列号
colMin = col[rowMinIndex]
colMax = col[rowMaxIndex]
A, B, C = generalEquation(rowMin, colMin, rowMax, colMax)
relativeVal = A * centerPoints[0] + B * centerPoints[1] + C
if relativeVal > 0:
sequence.append('a')
if relativeVal < 0:
sequence.append('b')
if relativeVal == 0:
sequence.append('c')
sequence = np.array(sequence) # 将其转化为numpy的数组形式
return sequence
效果:根据可视化平面与中点的位置关系,MRI中的位置序列大多是在同一侧,比较得到的序列和画上中线与中点的mhd是一致的,因此无法根据此方案得到序列的公共区域

上图是CT的0003数据画上中平面和中点后的每一层图像

上图是MRI的0003数据将中平面和中点画上的每一层图像
看以看出在MRI中,位置关系可能出现,连续几层都在一个方向,导致出现以下的结果
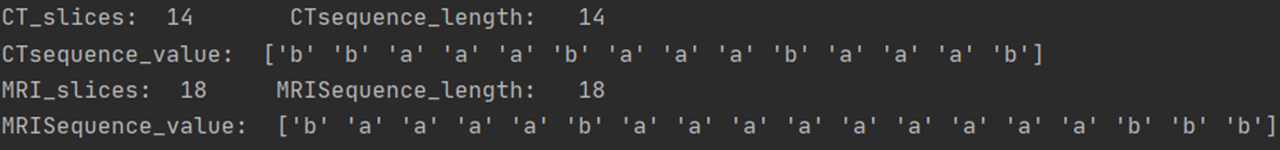
以上数据的序列前面几层至少还能对应上,但后面MRI的序列全为a
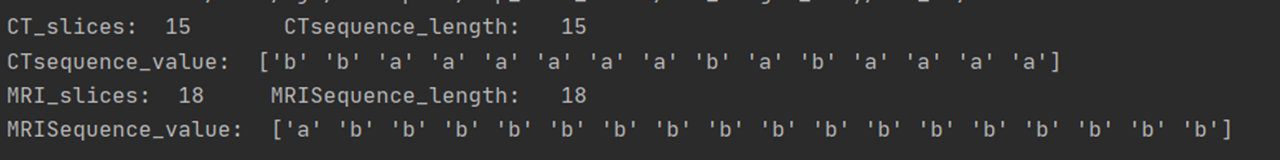
甚至出现上面这个数据这种情况,MRI序列全为b无法求出公共区域
问题可能存在的原因:
在拟合过程中可能会由于其中一条中线偏离得比较远,整个平面都被牵制得很远(虽然在拟合时选择的随机拟合方程理论上由于随机选取的关系可能不会取到上述说的比较极端的中线),但目前的推断大致是这个原因
测试二:考虑以上的出现问题的原因,可能由于拟合的中平面导致求出的位置信息有误,所以我们考虑取的平面不是根据中线拟合的平面而是取当前层的前面三层的中点形成的平面与当前层的中点的位置关系得到一个序列,这样得到的序列比数据的总层数少三层,由于我们考虑前几层一般不在CT和MRI公区域内且前面几层下巴的位置不存在病灶
首先可视化平面与点的位置关系,将当前层前三个中点组成的平面画到第4层上,以便检验后续求得的位置序列
实现:
import os
import numpy as np
import json
from glob import glob
import matplotlib.pyplot as plt
import SimpleITK as sitk
def readMhd(mhdPath):
'''
根据文件路径读取mhd文件,并返回其内容Array、origin、spacing
'''
itkImage = sitk.ReadImage(mhdPath)
imageArray = sitk.GetArrayFromImage(itkImage) # [Z, H, W]
origin = itkImage.GetOrigin()
# spacing = np.flipud(itkImage.GetSpacing()) # [X,Y,Z] -> [Z,Y,X]
spacing = itkImage.GetSpacing()
return imageArray, origin, spacing
def saveMhd(imageArray, origin, spacing, savePath):
'''
根据图像Array、origin、spacing构造itk image对象,并将其保存到savePath
'''
itkImage = sitk.GetImageFromArray(imageArray, isVector=False)
itkImage.SetSpacing(spacing)
itkImage.SetOrigin(origin)
sitk.WriteImage(itkImage, savePath)
def drawPlanePoints(imageType, MhdID, curCenterDict, imageArray, lineValue, pointValue):
center0 = curCenterDict[str(0)]['center']
center1 = curCenterDict[str(1)]['center']
center2 = curCenterDict[str(2)]['center']
shockSequence = {}
for z in range(3, imageArray.shape[0]):
print(imageType, ' ', mhdID, ' ', z)
if str(z) not in curCenterDict:
continue
center3 = curCenterDict[str(z)]['center']
# 向量表示: 终点减去起点,坐标顺序ZYX
vector1 = list(map(lambda x: x[0] - x[1], zip(center0, center1))) # 第一个点和第二个点表示的向量
vector2 = list(map(lambda x: x[0] - x[1], zip(center2, center1))) # 第二个点和第三个点表示的向量
# vector3 = list(map(lambda x: x[0] - x[1], zip(center3, center1))) # 第四个点和第二个点表示的向量
# 叉乘求vector1和vector2的法向量,其坐标顺序为ZYX
nomalVector = [vector2[1] * vector1[2] - vector2[2] * vector1[1],
vector2[2] * vector1[0] - vector2[0] * vector1[2],
vector2[0] * vector1[1] - vector2[1] * vector1[0]]
print('法向量: ', nomalVector)
b = -(nomalVector[0] * center0[0] + nomalVector[1] * center0[1] + nomalVector[2] * center0[2])
imageArray[z, int(center3[1]), int(center3[2])] = pointValue # 将中点画上
# 中点一个像素点不容易观察所以扩充它的8邻域
Neighbors = [(1, 1), (1, -1), (1, 0), (-1, 0), (-1, 1), (-1, -1), (0, 1), (0, -1)]
for neighbor in Neighbors:
dr, dc = neighbor
try:
imageArray[z, int(center3[1]) + dr, int(center3[2]) + dc] = pointValue # 将其8邻域都设置为9000
except IndexError:
pass
drawPlane(imageArray[z], nomalVector, b, lineValue, z) # z前面三层中点形成的平面画到当前层
# # 取法向量中与X轴正向的法向量
# if nomalVector[2] < 0: # 与X轴做内积为负时,取反所有的系数
# nomalVector[0] = - nomalVector[0]
# nomalVector[1] = - nomalVector[1]
# nomalVector[2] = - nomalVector[2]
#
# wave = nomalVector[0] * z + nomalVector[1] * center3[1] + nomalVector[2] * center3[2] + b # 将中点带入直线方程计算相对位置
#
# # 将序列保存成字典格式
# shockSequence[str(z)] = {
# "wave": wave
# }
center0, center1, center2 = center1, center2, center3 # 更新中点值
# print(imageType, 'shockSequence: ', shockSequence)
# return shockSequence
def drawPlane(image, w, b, value, z, scale=1):
'''
将前三层的中点组成的平面:w[0] * z + w[1] * y + w[2] * x + b = 0画到二维图像中
image: 二维的图像数组
w、b: 平面参数
value: 用来表达平面的像素值
z:当前需要加上的中线的层
scale:采样倍率,越大,所画出来的点越密集,防止由于离散点从而在平面中出现空洞
返回画上了中线的image
使用两种方法画中线(从三个平面去画)主要是为了避免除0操作
'''
# imageCopy = image.copy() # 避免修改原图
imageShape = image.shape
absW = np.abs(w)
coordX = np.arange(int(np.round(imageShape[1] * scale)))
coordX = coordX.astype(np.float32) # 确定X坐标
coordX /= scale
coordY = -(w[0] * z + w[2] * coordX + b) / w[1]
# 将坐标点转为int类型
coordY = np.round(coordY).astype(np.int)
coordX = np.round(coordX).astype(np.int)
# 将坐标list转成一维向量
coordY = coordY.reshape(-1)
coordX = coordX.reshape(-1)
# 筛选合法的坐标位置
validIndicesY = np.logical_and(coordY >= 0, coordY < imageShape[0]) # 保证Y没越界
validIndicesX = np.logical_and(coordX >= 0, coordX < imageShape[1]) # 保证X没越界
validIndices = np.logical_and(validIndicesY, validIndicesX)
coordY = coordY[validIndices]
coordX = coordX[validIndices]
image[coordY, coordX] = value
if __name__ == '__main__':
CTDir = # CT旋转通过检查的数据
MRIDir = # MRI通过检查的数据——目录未按照单个病例放一个目录
MRICPJsonPath = # MRI中点json文件(根据预测的mask所求)
CTCPJsonPath = # CT中点json文件
CTMidLineDir = # 用于保存将平面和中点画上去之后的CT图像
MRIMidLineDir = # 用于保存将平面和中点画上去之后的MRI图像
# 位置序列保存成对应的json文件的目录
CTshockSquenceDir =
MRIshockSquenceDir =
# 两部分数据可能不会对应到相同的病例,求交集
CTMhdID = os.listdir(CTDir) # 得到CT中所有病例ID
# MRIMhdID = os.listdir(MRIDir)
MRIMhdPath = glob(MRIDir + '/*.mhd') # mask->_seg.mhd,会包含mask的mhd,可以通过交集去掉
MRIMhdID = []
for mriPath in MRIMhdPath:
MRIMhdID.append(os.path.basename(mriPath).split('.')[0])
interMhdID = list(set(CTMhdID) & set(MRIMhdID)) # MRI对应38个文件,CT对应46个文件,交集37个文件
# print('交集的长度: ', len(interMhdID), '交集的序号: ', interMhdID)
count = 0
for mhdID in interMhdID:
print('mhdID: ', mhdID)
# 拼接单个MRI和CT的路径
mhdName = mhdID + '.mhd'
# ctMhdDir = os.path.join(CTDir, )
ctMhdPath = os.path.join(CTDir, mhdID, mhdName)
# print('ctMhdPath: ', ctMhdPath)
mriMhdPath = os.path.join(MRIDir, mhdName)
# print('mriMhdPath: ', mriMhdPath)
CTArray, CTorigin, CTspacing = readMhd(ctMhdPath)
MRIArray, MRIorigin, MRIspacing = readMhd(mriMhdPath)
# MRI的中点Json文件
with open(MRICPJsonPath + mhdID + 'Center.json', 'r') as fMRI:
curMRICenterDict = json.load(fMRI)
# CT的中点Json文件
with open(CTCPJsonPath + mhdID + 'Center.json', 'r') as fCT:
curCTCenterDict = json.load(fCT)
# MRI设置直线画上去的像素值为1000, CT设置的值为8000,中点CT9999,中点MRI设置为2000
drawPlanePoints('CT', mhdID, curCTCenterDict, CTArray, 8000, 9999) #
# # 将求得的CT MRI 序列保存成json --一个文件保存成一个json文件
# with open(CTshockSquenceDir + mhdID + '.json', 'w') as fp:
# json.dump(CTshockSquence, fp)
drawPlanePoints('MRI', mhdID, curMRICenterDict, MRIArray, 1000, 2000) # 画MRI的中线和中点
# with open(MRIshockSquenceDir + mhdID + '.json', 'w') as fp:
# json.dump(MRIshockSquence, fp)
CTMidLineSubDir = os.path.join(CTMidLineDir, mhdID)
if not os.path.exists(CTMidLineSubDir):
os.makedirs(CTMidLineSubDir)
MRIMidLineSubDir = os.path.join(MRIMidLineDir, mhdID)
if not os.path.exists(MRIMidLineSubDir):
os.makedirs(MRIMidLineSubDir)
newCTPath = os.path.join(CTMidLineSubDir, mhdName)
newMRIPath = os.path.join(MRIMidLineSubDir, mhdName)
print('newCTPath: ', newCTPath)
print('newMRIPath: ', newMRIPath)
# 保存截断后的CT MRI的mhd文件
saveMhd(CTArray, CTorigin, CTspacing, newCTPath)
saveMhd(MRIArray, MRIorigin, MRIspacing, newMRIPath)
count = count + 1
print('处理完毕: ', count)
效果:
上图CT的0003数据按照上述方案将前三层的中点组成的平面画到第4层上,可以看出从第4层才开始有直线,并且可以看出中点相对直线是左右摇摆的

上图是MRI0003数据的可视化效果
然后根据当前层的中点与第二层中点组成的向量在前三层中点组成的平面的法向量的投影的方向得到相对位置序列,按照方向转为字符串
实现:
def getSequence(CenterPointDict, sliceNum):
'''
:param CenterPointDict: 每一层对应的中点json文件
:param sliceNum : 该数据的层数
:return: 该序列在
'''
positions = {} # 用于存放序列的字典
center0 = CenterPointDict[str(0)]['center']
center1 = CenterPointDict[str(1)]['center']
center2 = CenterPointDict[str(2)]['center']
for i in range(3, sliceNum): # 有些层可能没有预测的mask即无法求中点
if str(i) not in CenterPointDict:
continue
center3 = CenterPointDict[str(i)]['center']
w1 = list(map(lambda x: x[0] - x[1], zip(center0, center1)))
w2 = list(map(lambda x: x[0] - x[1], zip(center2, center1)))
w3 = list(map(lambda x: x[0] - x[1], zip(center3, center1)))
# 利用叉乘求法向量,坐标顺序Z Y X
v = [w2[2] * w1[1] - w2[1] * w1[2],
w2[0] * w1[2] - w2[2] * w1[0],
w2[1] * w1[0] - w2[0] * w1[1]
]
# 取与X轴夹角为锐角的法向量
if v[0] < 0:
v = [-1 * v[0], -1 * v[1], -1 * v[2]]
v1 = [vv / (((v[0]) ** 2 + (v[1]) ** 2 + (v[2]) ** 2) ** 0.5) for vv in v] # 取单位法向量
position = v1[0] * w3[2] + v1[1] * w3[1] + v1[2] * w3[0] # 计算w3在单位法向量上的投影
if position > 0:
positions[str(i)] = {
'position': 'a' # 投影为正记为a
}
else:
positions[str(i)] = {
'position': 'b' # 投影为负记为b
}
center0, center1, center2 = center1, center2, center3
return positions
最后得到的是MRI和CT上对应的相对位置字符串,问题转化为求两个字符串的最长公共子串(LCS),根据我们的项目所求的特点,实际是求最长公共子序列问题与leetcode上的1143类似,不过这里我们的问题不是求最长公共子序列的长度而是求公共子序列的内容并对应回原串中找到子序列两端点的位置,后续根据这两个端点截断MRI和CT
实现:
def lcs(s1, s2, matrix):
'''
:param s1: MRI序列字符串
:param s2: CT序列字符串
:param matrix: 记录求公共序列长度时方向的来源
:return: 方向记录在matrix中无返回值
'''
l1, l2 = len(s1), len(s2)
f1 = [0 for i in range(l2 + 1)]
for i in range(1, l1 + 1):
f = [0 for i in range(l2 + 1)]
for j in range(1, l2 + 1):
if s1[i - 1] == s2[j - 1]:
f[j] = f1[j - 1] + 1
matrix[i][j] = "↖"
elif f1[j] == f[j - 1]:
f[j] = f1[j]
matrix[i][j] = "+"
elif f1[j] > f[j - 1]:
f[j] = f1[j]
matrix[i][j] = "↑"
else:
f[j] = f[j - 1]
matrix[i][j] = "←"
f1 = f
def printlcs(matrix, s, i, j, result="", s1begin=-1, s1end=-1, s2begin=-1, s2end=-1):
'''
递归找最长公共子序列对应的序列
'''
if i == 0 or j == 0:
# results.append([result,s1begin,s1end,s2begin,s2end])
# results[str(result)+str(s1begin)+str(s1end)+str(s2begin)+str(s2end)] = [result,s1begin,s1end,s2begin,s2end]
results.add((result, s1begin, s1end, s2begin, s2end))
return
if matrix[i][j] == "↖":
s1begin = i - 1
s2begin = j - 1
if s1end == -1:
s1end = i - 1
if s2end == -1:
s2end = j - 1
result = s1[i - 1] + result
printlcs(matrix, s, i - 1, j - 1, result, s1begin, s1end, s2begin, s2end)
elif matrix[i][j] == "+": # 当这个方向有两种情况,两个方向都递归回去找
printlcs(matrix, s, i - 1, j, result, s1begin, s1end, s2begin, s2end)
printlcs(matrix, s, i, j - 1, result, s1begin, s1end, s2begin, s2end)
elif matrix[i][j] == "↑":
printlcs(matrix, s, i - 1, j, result, s1begin, s1end, s2begin, s2end)
else:
printlcs(matrix, s, i, j - 1, result, s1begin, s1end, s2begin, s2end)
lcsindexs = {}
for imagename in imagenames:
name = imagename[-4:]
# with open('/home/ljl/workspace/exp_code_final/MRI_plane_params_result_crossljl256_01/plane_params.json', 'r') as f1:
# params = json.load(f1)
# centerZYX = params[name+'.mhd']['centerZYX']
# v = params[name+'.mhd']['w']
# v1 = [vv/(((v[0])**2 + (v[1])**2 + (v[2])**2)**0.5) for vv in v]
with open('/home/ljl/workspace/exp_code_final/center/256_01Waves4to1/' + name + '.json', 'r') as f1:
mri = json.load(f1)
with open('/home/ljl/workspace/exp_code_final/center/ctWaves4to1/' + name + '.json', 'r') as f2:
ct = json.load(f2)
mriindexs = []
mriwaves = []
ctindexs = []
ctwaves = []
for i in range(3, len(mri) + 10):
if str(i) not in mri:
continue
mriindexs.append(i)
mriwaves.append(mri[str(i)]['wave'])
for i in range(3, len(ct) + 10):
if str(i) not in ct:
continue
ctindexs.append(i)
ctwaves.append(ct[str(i)]['wave'])
s1, s2 = "".join(mriwaves), "".join(ctwaves)
l1, l2 = len(mri), len(ct)
matrix = matrix = [["" for i in range(l2 + 1)] for j in range(l1 + 1)]
results = set()
lcs(s1, s2, matrix)
result = ""
# 使用set,对于不同方向求得相同子串时去重,
results = set() # result中存放5个值,公共子序列的内容,s1的起始 s1的终止,s2的起始 s2的终止
s1begin, s1end, s2begin, s2end = -1, -1, -1, -1
# result,s1begin,s1end,s2begin,s2end = printlcs(matrix,s1,l1,l2)
printlcs(matrix, s1, l1, l2)
# print('病例ID:', name)
# print('MRI相对平面的位置序列: ', s1)
# print('CT相对平面的位置序列: ', s2)
best = ["", -1, -1, -1, -1]
for res in results: # 对于有多种解的情况我们根据CT和MRI截断后长度之和最大取得最优解
if (res[2] - res[1]) + (res[4] - res[3]) > (best[2] - best[1]) + (best[4] - best[3]):
best = [res[0], res[1], res[2], res[3], res[4]]
# print('公共子序列解的个数: ', len(results))
print('最优解: ', best[0])
# print('MRI相应的层索引: ', mriindexs[best[1]:best[2] + 1])
# print('MRI相应的序列: ')
# print("".join(mriwaves[best[1]:best[2] + 1]))
#
# print('CT相应的层索引: ', ctindexs[best[3]:best[4] + 1])
# print('CT相应的序列: ')
# print("".join(ctwaves[best[3]:best[4] + 1]), '
')
# 将求得的CT MRI 序列ID保存成字典
lcsindexs[name] = {
"mriindexs": mriindexs[best[1]:best[2] + 1],
"ctindexs": ctindexs[best[3]:best[4] + 1]
}
以上求得的MRI、CT相应的序列,可选择其首尾用于截断MRI、CT
将以上处理的数据再用于配准的效果:相较于上周根据互信息截断之后的配准效果来看,位置信息略差,但是保留的层数比使用互信息截断的层数多,病灶也能保留的更多