最近有一大堆质谱数据,但好多蛋白都是已经研究过得,为了寻找和bait蛋白相关,但又特异的假定蛋白,决定写个Python程序过滤掉不需要的信息,保留想要的信息。
方案:
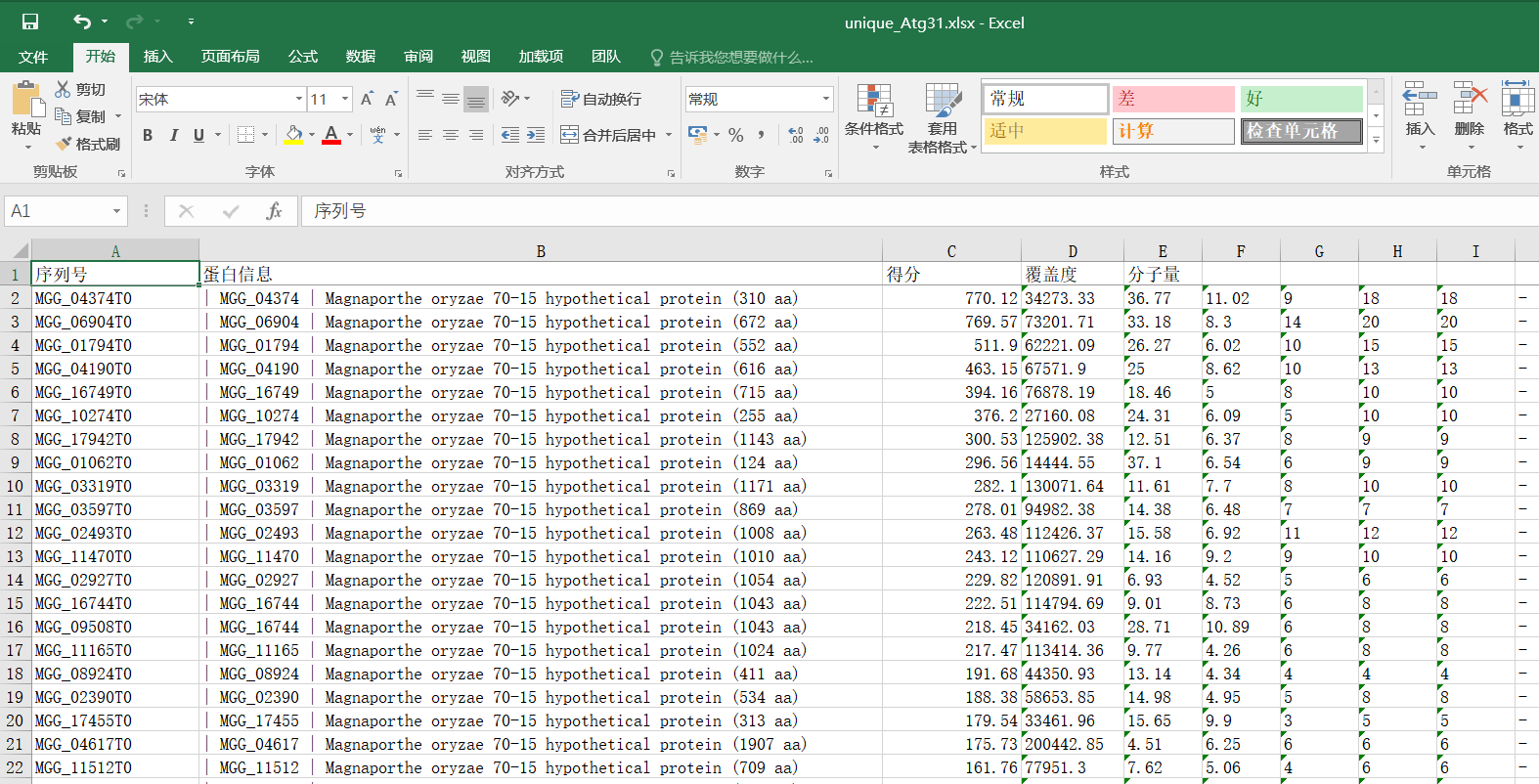
1,找出所有质谱数据中特异蛋白中的假定蛋白并按得分高低排序。
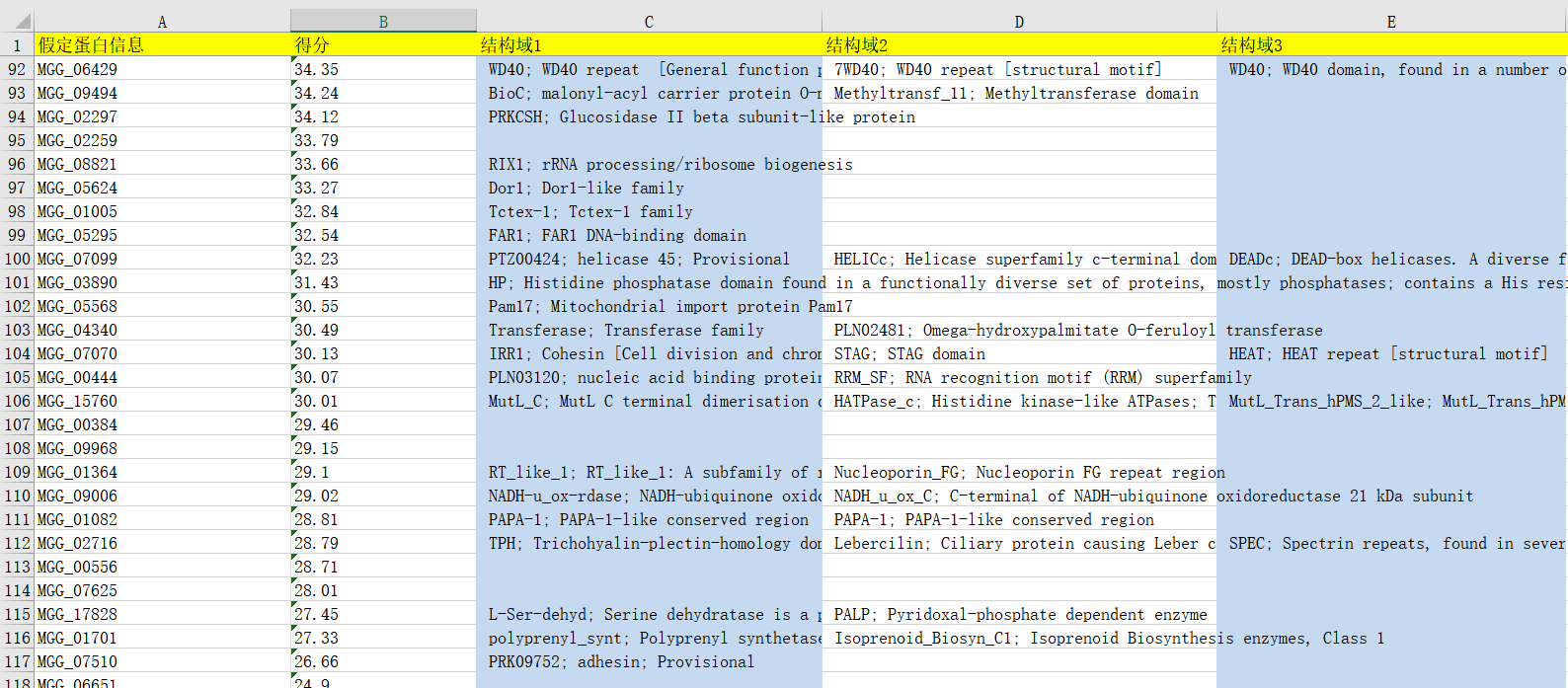
2,根据蛋白序列号找出假定蛋白可能含有的结构域,写入excel文件。
3,说干就干
第一步主要用集合的性质去重,用re正则表达式找出序列号,用openpyxl写入excel,根据得分排序。
#质谱蛋白去重 import re import openpyxl reg = re.compile(r'MGG_d{5}') def read_csv(name): with open(name,'r') as f: csv_data = f.read() csv_num = re.findall(reg, csv_data) return set(csv_num) def write_excel(file,filename): wb = openpyxl.Workbook() ws = wb.active ws['A1'],ws['B1'],ws['C1'],ws['D1'],ws['E1'] = '序列号','蛋白信息','得分','覆盖度','分子量' ws.freeze_panes = 'A2' for num in unique: for line in open(file): #print(line+'****') if num in line and 'hypothetical protein' in line: ws.append((line.split(',')[1:])) wb.save(filename) if __name__ == '__main__': ATG3 = read_csv(r'C:UserszhuxuemingDesktopATG3.csv')#添加所需比对的文件,以及绝对路径。需要.csv格式的excel Vps9 = read_csv(r'C:UserszhuxuemingDesktopvps9.csv')#添加所需比对的文件,以及绝对路径。需要.csv格式的excel K3G4 = read_csv(r'C:UserszhuxuemingDesktopK3G4.csv')#添加所需比对的文件,以及绝对路径。需要.csv格式的excel unique = ATG3-(Vps9|K3G4)#进行数据筛选,ATG3-(Vps9|K3G4)代表ATG3中的数据有,而vps9和K3G4中都没有的集合。-号为差集,|为并集 #unique_Vps9 = Vps9-(K3G4|ATG3) write_excel(r'C:UserszhuxuemingDesktopATG3.csv', r'C:UserszhuxuemingDesktopunique_Atg31.xlsx')#第一个为需要比对的文件,第二个为所输出的excel和路径
>>>
第二步,根据第一步excel中的序列号在NCBI上找出结构域信息获取并写入新的excel中。
import requests import re import openpyxl from bs4 import BeautifulSoup head = {'User-Agent':'Mozilla/5.0 (Windows NT 10.0; WOW64) AppleWebKit/537.36 (KHTML, like Gecko) Chrome/63.0.3239.132 Safari/537.36'} url = r'https://www.ncbi.nlm.nih.gov/gene/?term=' wb = openpyxl.Workbook() ws = wb.active ws['A1'],ws['B1'],ws['C1'],ws['D1'],ws['E1'] = '假定蛋白信息','得分','结构域1','结构域2','结构域3' ws.freeze_panes = 'A2' def seq_data(file, filename): for line in open(file): if 'MGG_' in line: score = line.split(',')[2] MGG = re.search(r'MGG_d{5}',line).group(0) full_url = url + MGG l = [] l.append(MGG) l.append(score) try: res = requests.get(full_url,headers = head) res.encoding = 'utf-8' soup = BeautifulSoup(res.text,'lxml') domain = soup.find_all("dd",class_='clearfix')#获取标签内容 for each in domain: l.append(each.text) except BaseException: pass ws.append(l)#写入excel wb.save(filename)#保存 if __name__ == '__main__': seq_data(r'C:UserszhuxuemingDesktopunique_Atg31.csv', r'C:UserszhuxuemingDesktopATG3_special_hyp_protein_domain.xlsx')