题目描述:
http://acm.hdu.edu.cn/showproblem.php?pid=1560
中文大意:
用户会给出 N 条 DNA 序列,要求我们计算出一条尽可能短的 DNA 序列,使其包含所有给定序列。
给定序列的嘌呤、嘧啶可以分开,但前后关系不能变。
思路:
从第 0 位开始,逐位设置最短 DNA 序列。
而每个位置,有且仅有 4 种选择:A C G T。
选择依据是:该嘌呤或嘧啶是否是某条基因片段剩余部分的首个字母。
例如下图中,最短 DNA 序列的第 3 位,可设置为 G 或 T。
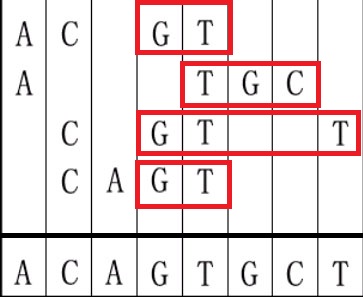
假设当前位置设置为了 G,则:
第一条基因片段的剩余部分变成了“T”,
第二条的剩余部分不变,仍然为“TGC”,
第三条的剩余部分变成了“TT”,
第四条的剩余部分变成了“T”。
接着,我们去设置最短序列的第 4 位,以此类推,直至处理完所有基因片段。
回溯回来后,再尝试将第三位设置成 T,展开新的深搜。
代码:
#include<bits/stdc++.h>
using namespace std;
int n;
string str[8];//n 条 DNA 序列
char DNA[4] = {'A', 'C', 'G', 'T'};
int pos[8];//各 DNA 序列当前的处理位置
int depth;//深搜深度
bool have_result;//是否有解
//index:填写最短序列的第 index 位
void dfs(int index){
//剪枝:
//1.超过深搜限制深度
if(index > depth){
return;
}
//2.找到正确答案
if(have_result){
return;
}
//n 条DNA序列剩余部分的最大长度
int max_surplus = 0;
for(int i=0;i<n;i++){
int surplus = str[i].length() - pos[i];
if(surplus > max_surplus){
max_surplus = surplus;
}
}
//3.估价函数:已搜层数 + 预搜层数 > 限制深度
if(index+max_surplus > depth){
return;
}
//4.没有剩余基因片段了,即找到了一组解
if(max_surplus == 0){
have_result = true;
return;
}
//pos为公共数据,每次深搜回溯后,需要保持其值不变,故提前复制一份
int pre_pos[8];
memcpy(pre_pos, pos, sizeof(pos));
//最短序列的当前位置可以为:A 或 C 或 G 或 T
for(int k=0;k<4;k++){
//选择依据:DNA[k] 是当前某条基因片段剩余部分的首个字母
bool appear = false;
for(int i=0;i<n;i++){
int j = pos[i];
if(j < str[i].length() && str[i][j] == DNA[k]){
appear = true;
pos[i]++;
}
}
if(appear){
dfs(index+1);
}
//恢复 pos
memcpy(pos, pre_pos, sizeof(pre_pos));
}
}
int main(){
int t;
cin>>t;
while(t--){
cin>>n;
depth = -1;
for(int i=0;i<n;i++){
cin>>str[i];
//最长基因片段肯定 ≤最短序列的长度,故可以直接将其作为起始深度
if(depth < str[i].length()){
depth = str[i].length();
}
}
memset(pos, 0, sizeof(pos));
have_result = false;
while(true){
dfs(0);
if(have_result){
break;
}
//深度不断增加
depth++;
}
cout<<depth<<endl;
}
}