一、对基因型进行PCA分析;
命令如下所示:
QTLtools pca --vcf genotypes.chr22.vcf.gz --scale --center --maf 0.05 --distance 50000 --out test
genotypes.chr22.vcf.gz输入文件如下所示:
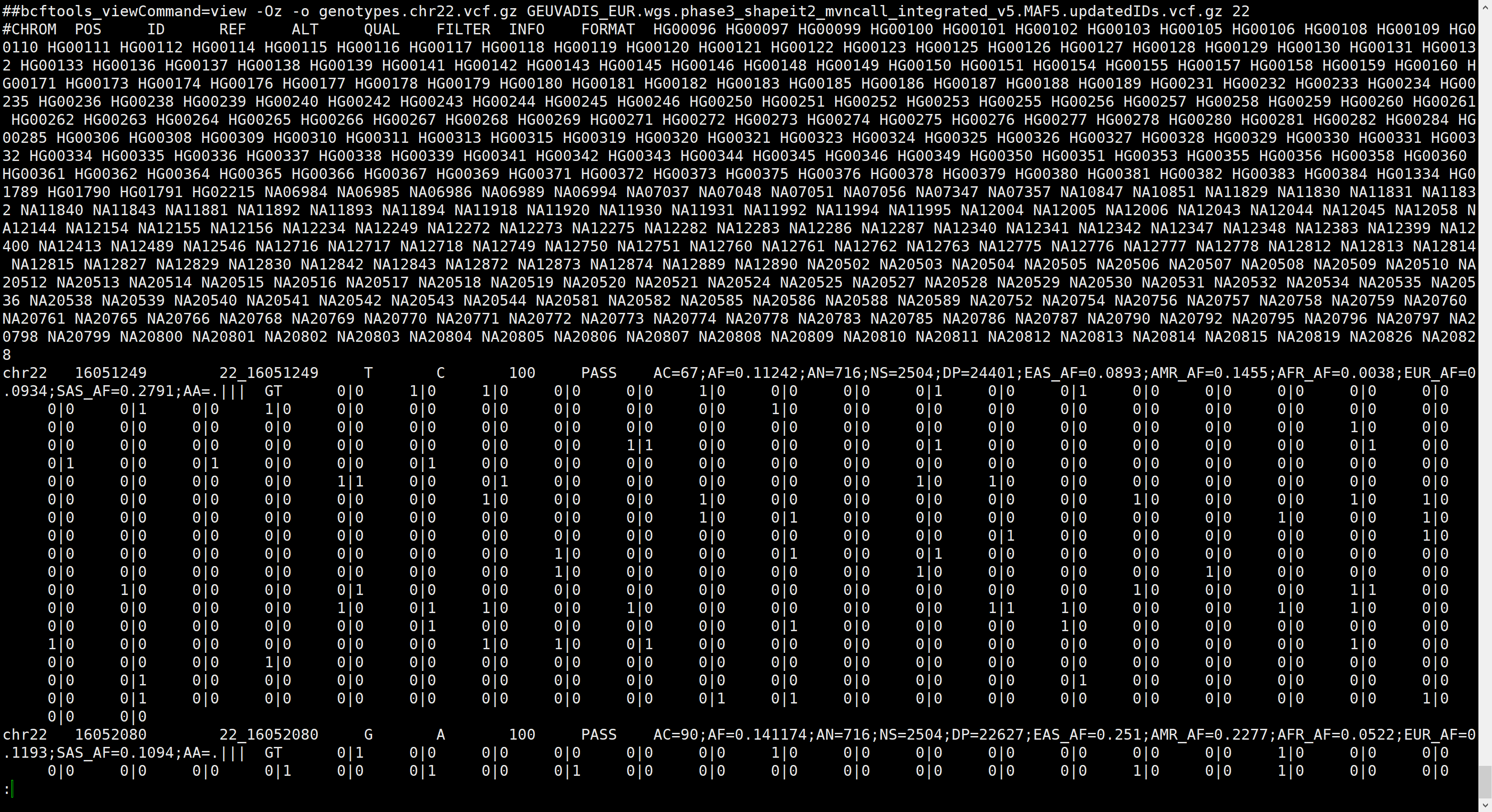
- --maf 0.05 指的是只考虑此等位基因频率小于0.05的变异位点;
- --distance 50000 指的是只考虑间隔至少50kb的变异位点;
运行后,会生成两个输出文件test.pca和test.pca_stats
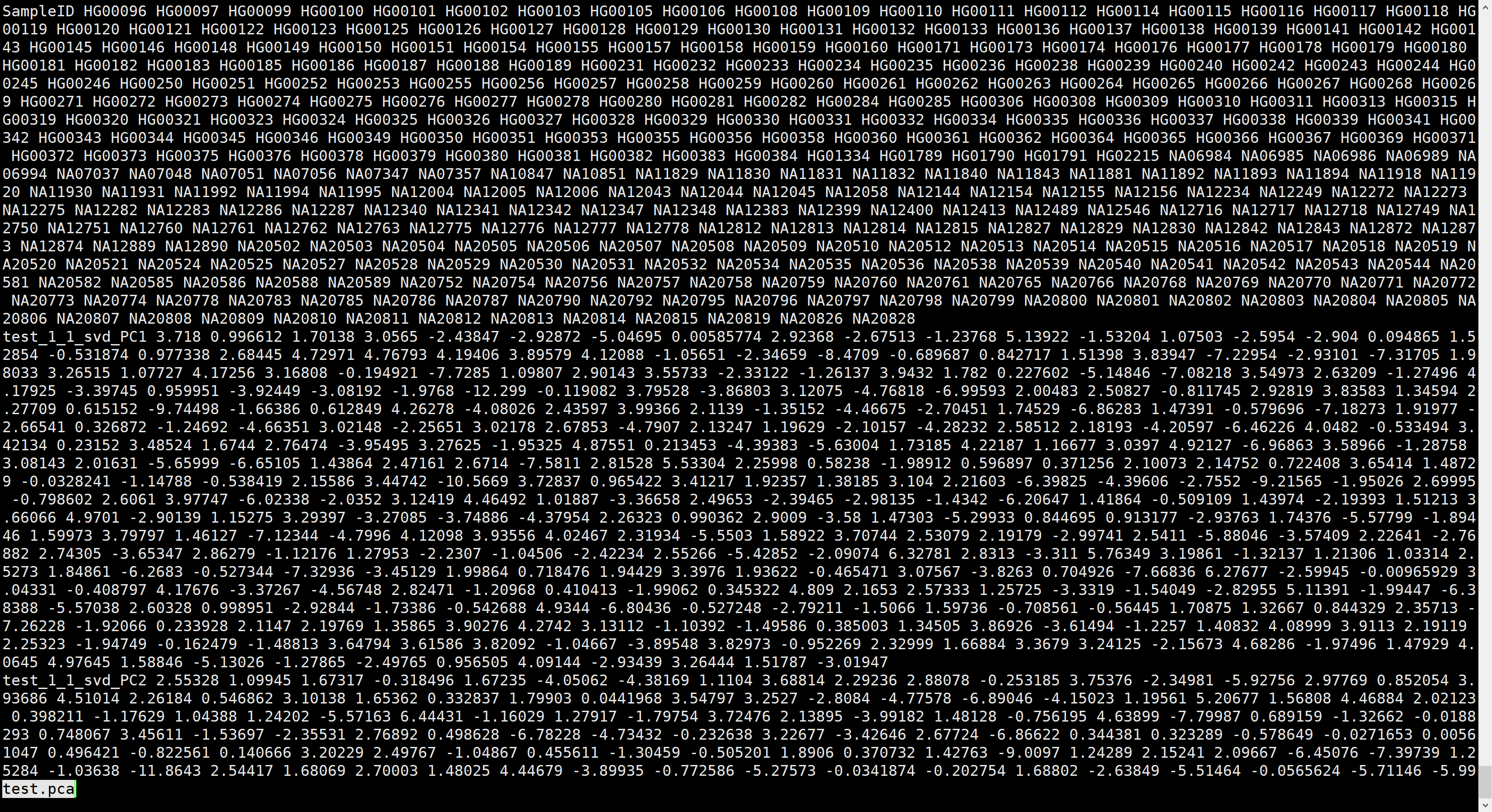
test.pca表示基因型的PCA结果,如下所示:
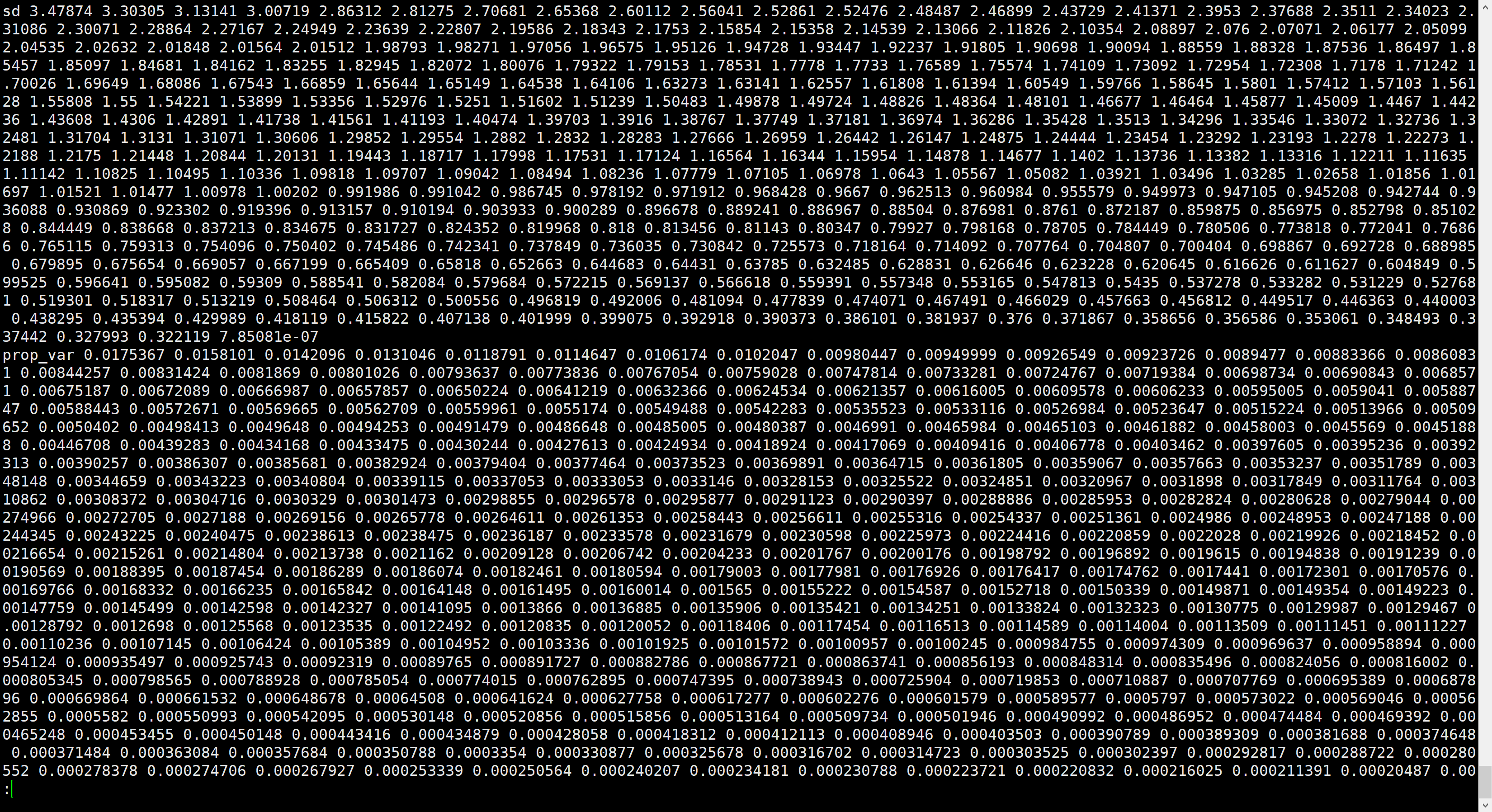
test.pca_stats指的是每个PC所解释 的百分比,如下所示:
二、对表型进行PCA分析;
命令如下所示:
QTLtools pca --bed genes.50percent.chr22.bed.gz --scale --center --out genes.50percent.chr22
输入文件genes.50percent.chr22.bed.gz如下所示:

- 表型文件总共有6+N列,N指的是样本数量,比如示例文件有4个样本,那么表型文件就有10列;
- 第一列是染色体;
- 第二列是起始位置;
- 第三列是终止位置;
- 第四列是ID,比如exon的ID;
- 第五列是group ID, 比如gene ID;
- 第六列是方向;
- 从第七列开始,则是每个样本对应的表型值;
- 文件分隔符为 TAB;
- 缺失值用NA表示,对于缺失值,QTLtools 会自动impute;
同样的,会生成genes.50percent.chr22.pca和genes.50percent.chr22.pca_stats两个文件。
genes.50percent.chr22.pca如下所示:

genes.50percent.chr22.pca_stats如下所示:
