马氏距离(Mahalanobis Distence)
是度量学习(metric learning)中一种常用的测度,所谓测度/距离函数/度量(metric)也就是定义一个空间中元素间距离的函数,所谓度量学习也叫做相似度学习。
什么是马氏距离
似乎是一种更好度量相似度的方法。
马氏距离是基于样本分布的一种距离。
物理意义就是在规范化的主成分空间中的欧氏距离。
所谓规范化的主成分空间就是利用主成分分析对一些数据进行主成分分解。
再对所有主成分分解轴做归一化,形成新的坐标轴。由这些坐标轴张成的空间就是规范化的主成分空间。
解释第一句话: 马氏距考虑到了样本在不同类别的不同分布情况。第二句: 是基于欧氏距离的一种改进。后两句针对高维线性分布的数据中各维度间非独立同分布分布的问题,后文继续探讨。
距离公式:
-
单点长度:
(D_M(x) = sqrt[]{(x-mu)^{T} Sigma^{-1}(x-mu)})
-
两点间长度:
(D_M(x, y) = sqrt[]{(x-y)^{T} Sigma_{x,y}^{-1}(x-y)})
- 其中(Sigma)是多维随机变量的协方差矩阵,若其是单位矩阵,则马氏距离退化为欧式距,即各维度间独立且同分布(因为只有同分布才会方差相同,对角线是相同的方差)
- (单点长度中的这个(mu)可以看成是样本点的中心,欧氏距离的一般定义是把原点看成中心点)
马氏距离的作用(基于欧式距离)
欧氏距离对于不同的量纲一视同仁
举个例子:在模式识别课上有个数据是学生的身高、鞋码、体重来分类性别。三个数据的单位都不一样,如果x1样本和x2的鞋码之间相差10(其他一样),x1和x3的体重相差10(其他一样),在欧式空间中就认为x1,x2的距离和x1,x3的距离是相同的,明显x1和x2的性别差异就很大。
下面随便举一个例子,随机生成一个维度间相关的分布
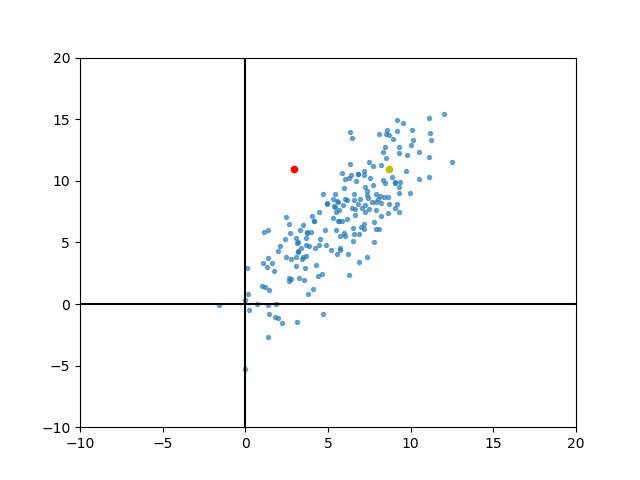
归一化可以解决这个问题。归一化之后维度的中心,就在坐标原点的地方了。
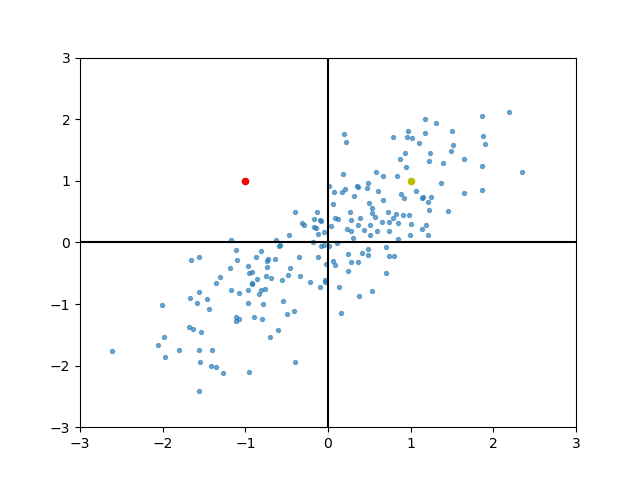
归一化后的欧氏距离考虑方差的影响
-
举个例子:一个样本点分别到两个类分布中心的距离相同(欧式),可以通过观察方差分布来分类。
-
判断两个点哪一个是该类的例子:欧氏距离对于方差视而不见,即使中心化之后两个点到中心的相对距离的互相关系(谁近谁远)是不会改变的。
如上图(模拟某一个类别的两个属性值的分布),两个点到其维度中心的距离都相同,但是明显左侧的红点不属于这一类。
但是仅计算测试样本(红,黄两点)与样本中心的欧式距离来判断,这两个点会被分为同一类。
所以要本质上解决这个问题,就要针对主成分分析中的主成分来进行标准化。
马氏距离——向量空间按照主成分旋转后的欧氏距离
主成分分析:找到主成分方向(方差大的维度),将整个样本空间按照主成分方向旋转,让主成分方向作为新的轴,让维度之间尽可能互相独立。
马氏距离:在主成分空间中,样本到原点的欧式距离(因为PCA的时候已经中心化数据了,所以该类别样本的中心就是原点)
计算步骤:
-
找出主成分方向,中心化后的数据求协方差矩阵,经过特征值分解找出排序后的特征值矩阵U
-
U矩阵作用于数据矩阵,旋转空间
-
新数据空间标准化,减去均值除以标准差,标准化后,让维度同分布,独立
-
计算新坐标的欧氏距离
注意: 此时原点是类中心
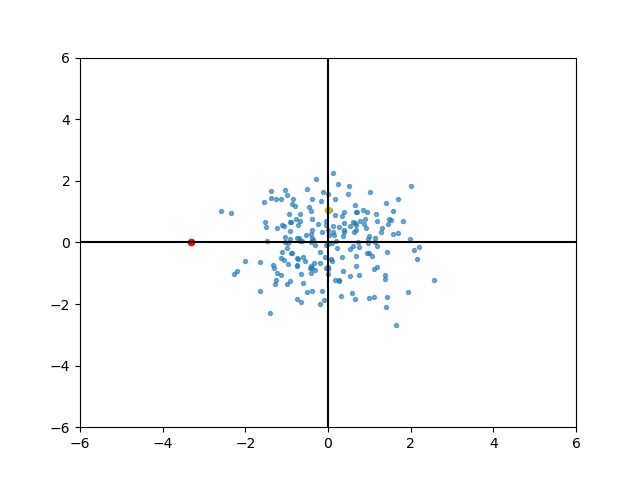
可见现在黄色的点距离中心的距离比红色的点更近了,红点即为离群点。
可见在主成分空间能更好的看相似度。
数学推导
根据步骤:
假设数据没有中心化后的(X)矩阵每一列是一个样本,旋转矩阵为(U),新的坐标为:
变换后每个维度都线性无关,各维度方差为协方差矩阵的特征值,满足如下(最后要用到):
(Cov_F = (F-mu_F)(F-mu_F)^T = U^T(X-mu_X)(X-mu_X)^TU)
(=U^TCov_XU)
马氏距离是旋转变换加上缩放之后的欧氏距离(这里缩放其实就是对F在做一次标准化,使得维度同方差):
每一个维度的方差:(sigma_{Fi} = sqrt{lambda_{Fi}},Cov_F=diag(lambda_{F1},..., lambda_{Fn}))
标准化之后(f是一个样本,f_i是第i个维度):(fi' = (frac{f_i-mu_{Fi}}{sigma_{Fi}}))
坐标到原点的欧氏距离(未开方):(D_M=f_1'^2 + f_2'^2 + f_3'^2 + ... + f_n'^2)
(=(f_1-mu_{F1}, f_2-mu_{F2}, ...,f_n-mu_{Fn})diag(frac{1}{lambda_1}, frac{1}{lambda_2}, ..., frac{1}{lambda_n})(f_1-mu_{F1}, f_2-mu_{F2}, ...,f_n-mu_{Fn})^T)
(=(f-mu_F)^T(U^TCov_XU)^{-1}(f-mu_F),中间的对角阵是Cov_F^{-1})
(= (x-mu_X)^TUU^TCov_X^{-1}UU^T(x-mu_X), 由前面x旋转得到的f)
(= (x-mu_X)^TCov_X^{-1}(x-mu_X), U)是单位正交的
最后在开平方就是马氏距离
推导式中的U可以是一个非方阵,也就是用了PCA的方法,但是不影响结果,因为在中间步骤U会被消去。
马氏距离的问题
- 协方差矩阵必须是满秩,需要求逆,对称矩阵不一定可逆哦,原因是其特征值会出现0,如果出现则可以考虑先PCA,这里PCA不会损失信息,原因上面提到了。
- 不能处理非线性流行(manifold),流行问题还没有了解。。
- 马氏距离其实也是基于样本的,自学习到一种空间变化,在主成分空间计算距离
附python作图&实现代码
class MahalanobisDistance():
# 没有考虑的很周全的代码
def __init__(self, X):
"""
构造函数
:param
X:m * n维的np数据矩阵 每一行是一个sample 列是特征
"""
self._PCA = None
self._mean_x = np.mean(X, axis=0)
mean_removed = X # - self._mean_x
# cov = np.dot(mean_removed.T, mean_removed) / X.shape[0] # 计算协方差矩阵
cov = np.cov(mean_removed, rowvar=False)
# 判断协方差矩阵是否可逆
if np.linalg.det(cov) == 0.0:
# 对数据做PCA 去掉特征值0的维度
eig_val, eig_vec = np.linalg.eig(cov)
e_val_index = np.argsort(-eig_val) # 逆序排
e_val_index = e_val_index[e_val_index > 1e-3] # 需要特征值大于0的维度
self._PCA = eig_vec[:, e_val_index] # 降维矩阵 Z = XU
PCA_X = np.dot(X, self._PCA) # 降维
self._mean_x = PCA_X.mean(axis=0) # 重新计算均值 去中心
mean_removed = PCA_X # - self._mean_x
# cov = np.dot(mean_removed.T, mean_removed) / PCA_X.shape[0] # 重新计算协方差矩阵
cov = np.cov(mean_removed, rowvar=False)
self._cov_i = np.linalg.inv(cov) # 协方差矩阵求逆
def __call__(self, X, y=None):
"""
计算x与y的马氏距离 如果不传入y则计算x到样本中心的距离
:param
X:行向量/矩阵 样本点特征维数必须和原始数据一样
y:行向量 样本点特征维数必须和原始数据一样
:return
distance 马氏距离 如果出错则返回-1
"""
# 不考虑出错的情况 维度不符合
if y is None:
# 计算到样本中心的距离
y = self._mean_x
X_data = X.copy()
if self._PCA is not None:
X_data = np.dot(X_data, self._PCA) # 对数据降维
X_data = X_data - y
distance = np.dot(np.dot(X_data, self._cov_i), X_data.T)
if len(X.shape) != 1:
# X是一个矩阵
distance = distance.diagonal()
return np.sqrt(distance)
# m_distance = MahalanobisDistance(train_data)
# d = m_distance(x, y)
下面的作图可看可不看,作为记录
def plot_distri(x1, x2, rang, p1, p2):
# 画分布图 p1 p2是测试点每一个维度的方差
plt.figure()
plt.scatter(x1, x2, s=10, marker='H', alpha=0.6)
plt.scatter(p1[0], p2[0], s=20, color='r')
plt.scatter(p1[1], p2[1], s=20, color='y')
plt.xlim(rang)
plt.ylim(rang)
plt.axvline(x=0, color='k')
plt.axhline(y=0, color='k')
def normalization(X):
mean = X.mean(axis=1)
std = X.std(axis=1)
# print("mean", mean)
return (X - np.tile(mean.reshape(-1, 1), X.shape[1])) / np.tile(std.reshape(-1, 1), X.shape[1]), std
x1 = np.random.normal(6, 3, 200)
x2 = x1 * 1.2 + 2.5 * np.random.randn(200)
rang =(-10, 20)
# 画一个有维度相关性的图
# 去中心化
x1_m = x1.mean()
x1_std = x1.std()
x2_m = x2.mean()
x2_std = x2.std()
x1s = (x1 - x1_m) / x1_std
x2s = (x2 - x2_m) / x2_std
px1 = np.array([-1, 1]) # 第二个点是 正例
px2 = np.array([1, 1])
pxx1 = px1 * x1_std + x1_m
pxx2 = px2 * x2_std + x2_m
plot_distri(x1, x2, rang, pxx1, pxx2)
plot_distri(x1s, x2s, (-3, 3), px1, px2)
X = np.vstack([x1s, x2s])
covX = np.cov(X, rowvar=True)
eigval, eigvec = np.linalg.eig(covX)
eig_arg = np.argsort(-eigval)
U = eigvec[:, eig_arg]
# 按照主成分方向旋转
M = np.dot(U, X)
# 按照特征大小缩放
M, std = normalization(M)
M_p = np.dot(U, np.array([[-1, 1], [1, 1]]))
M_p[0, :] = M_p[0, :] / std[0]
M_p[1, :] = M_p[1, :] / std[1]
plot_distri(M[0, :], M[1, :], (-6, 6), M_p[:, 0], M_p[:, 1])
plt.show()