REF https://www.plob.org/article/5987.html
解压后,查看html格式的结果报告。结果分为如下几项:
结果分为绿色的"PASS",黄色的"WARN"和红色的"FAIL"。
1 Basic statistics
如下面例子所示: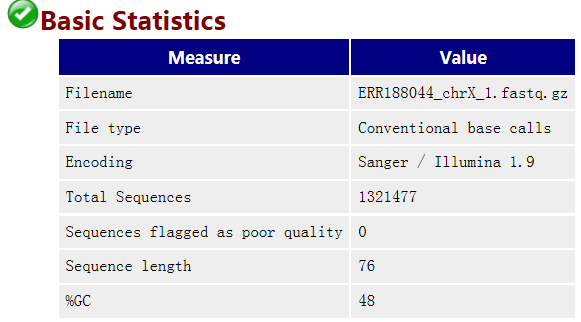
2 Per base sequence quality
quality就是Fred值,-10*log10(p),p为测错的概率。所以一条reads某位置出错概率为0.01时,其quality就是20。图像如下面例子:
横轴代表位置,纵轴代表quality。红色表示中位数,黄色是25%-75%区间,触须是10%-90%区间,蓝线是平均数。
若任一位置的下四分位数低于10或中位数低于25,报"WARN";若任一位置的下四分位数低于5或中位数低于20,报"FAIL".
3 Per Sequence Quality Scores
每条reads的quality的均值的分布: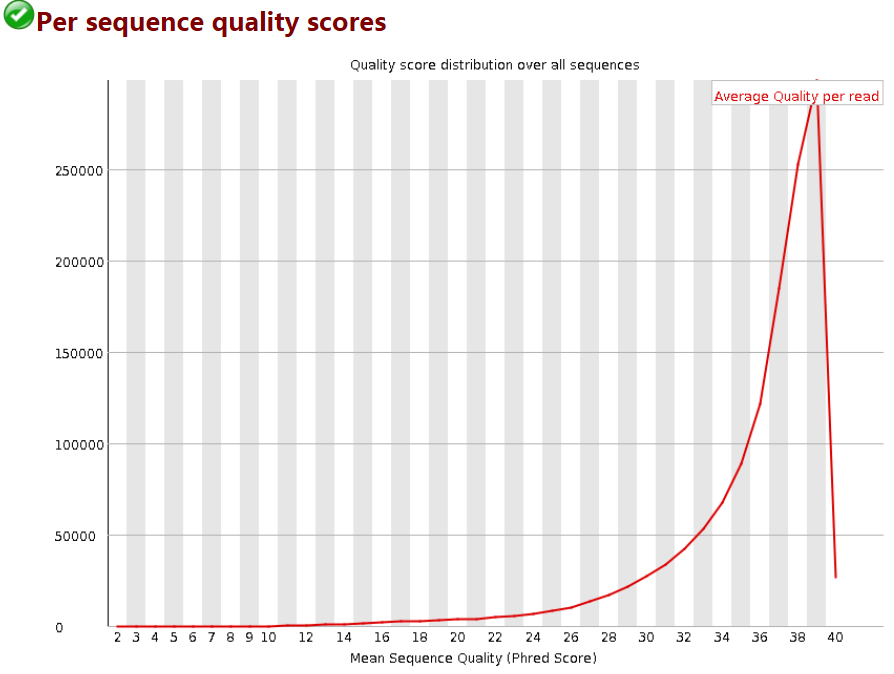
横轴为quality,纵轴是reads数目。当出现上图的情况时,我们就会知道有一部分reads具有比较差的质量。
当峰值小于27(错误率0.2%)时报"WARN",当峰值小于20(错误率1%)时报"FAIL"。
4 Per Base Sequence Content
对所有reads的每一个位置,统计ATCG四种碱基(正常情况)的分布: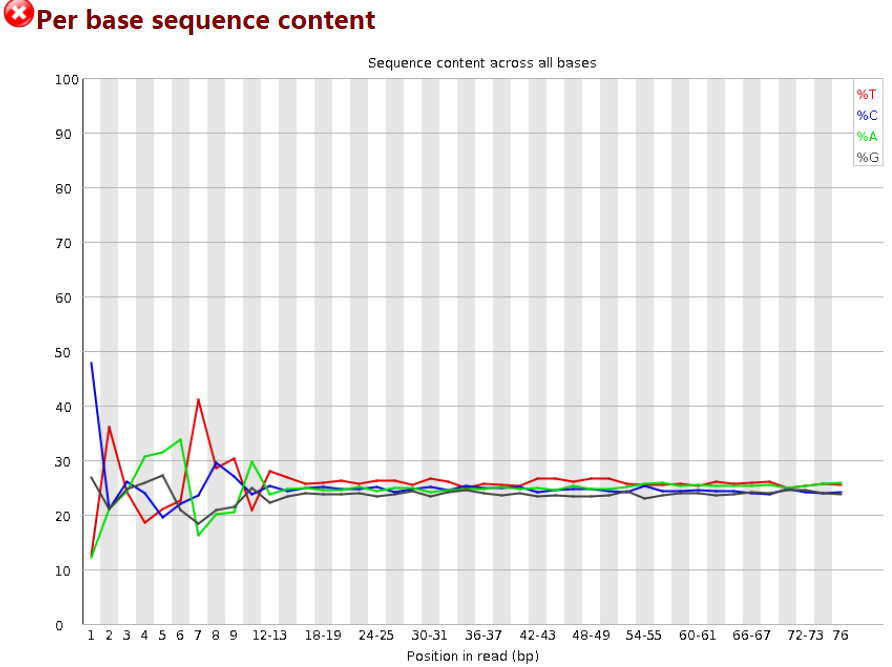
横轴为位置,纵轴为百分比。 正常情况下四种碱基的出现频率应该是接近的,而且没有位置差异。因此好的样本中四条线应该平行且接近。当部分位置碱基的比例出现bias时,即四条线在某些位置纷乱交织,往往提示我们有overrepresented sequence的污染。当所有位置的碱基比例一致的表现出bias时,即四条线平行但分开,往往代表文库有bias (建库过程或本身特点),或者是测序中的系统误差。
当任一位置的A/T比例与G/C比例相差超过10%,报"WARN";当任一位置的A/T比例与G/C比例相差超过20%,报"FAIL"。
5 Per Base GC Content
对所有reads的每个位置,统计GC含量。
如果建库足够均匀,reads的每个位置应当是没有差异的,所以GC含量的线应当平行于X轴,反映样品(基因组、转录组等)的GC含量。当部分位置GC含量出现bias时,往往提示我们有overrepresented sequence的污染。当所有位置的GC含量一致的表现出bias时,往往代表文库有bias (建库过程或本身特点),或者是测序中的系统误差。
当任一位置的GC含量偏离均值的5%时,报"WARN";当任一位置的GC含量偏离均值的10%时,报"FAIL"。
6 Per Sequence GC Content
统计reads的平均GC含量的分布。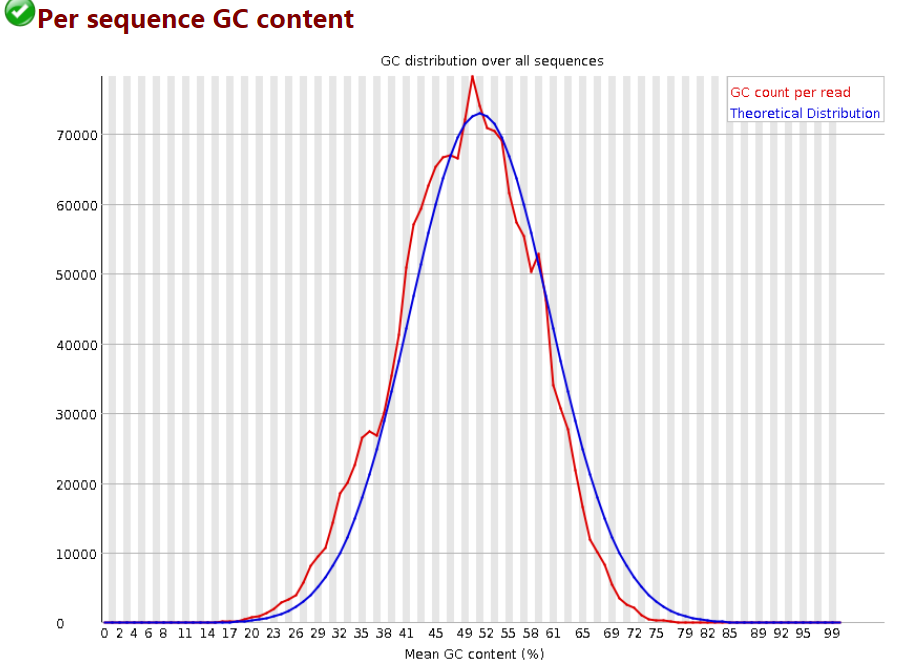
红线是实际情况,蓝线是理论分布(正态分布,均值不一定在50%,而是由平均GC含量推断的)。 曲线形状的偏差往往是由于文库的污染或是部分reads构成的子集有偏差(overrepresented reads)。形状接近正态但偏离理论分布的情况提示我们可能有系统偏差。
偏离理论分布的reads超过15%时,报"WARN";偏离理论分布的reads超过30%时,报"FAIL"。
7 Per Base N Content
当测序仪器不能辨别某条reads的某个位置到底是什么碱基时,就会产生“N”。对所有reads的每个位置,统计N的比率:

正常情况下N的比例是很小的,所以图上常常看到一条直线,但放大Y轴之后会发现还是有N的存在,这不算问题。当Y轴在0%-100%的范围内也能看到“鼓包”时,说明测序系统出了问题。当任意位置的N的比例超过5%,报"WARN";当任意位置的N的比例超过20%,报"FAIL"。
8 Sequence Length Distribution
reads长度的分布。
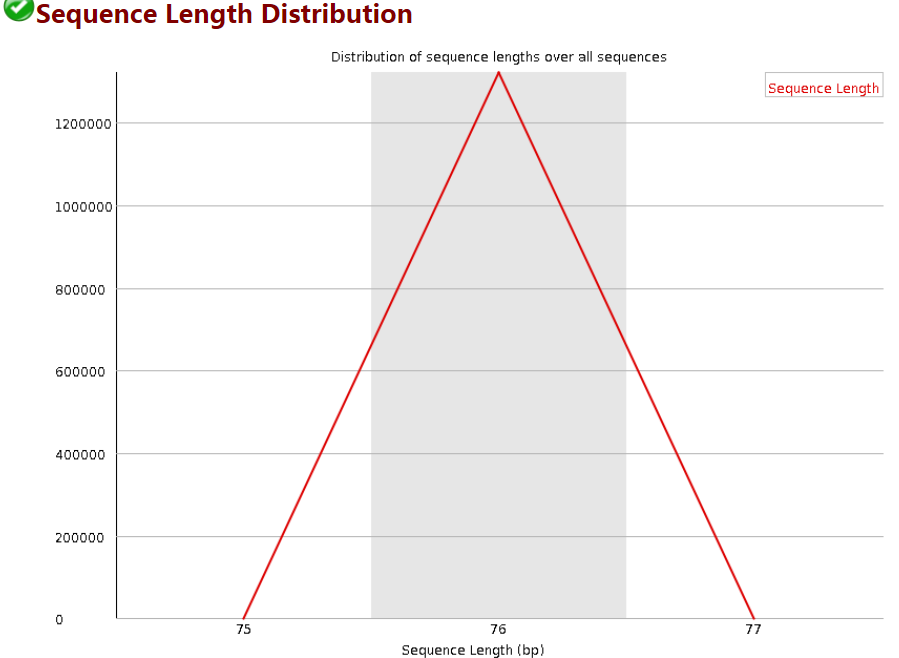
当reads长度不一致时报"WARN";当有长度为0的read时报“FAIL”。
9 Duplicate Sequences
统计序列完全一样的reads的频率。测序深度越高,越容易产生一定程度的duplication,这是正常的现象,但如果duplication的程度很高,就提示我们可能有bias的存在(如建库过程中的PCR duplication)。
duplication_levels
横坐标是duplication的次数,纵坐标是duplicated reads的数目,以unique reads的总数作为100%。 上图的情况中,相当于unique reads数目~20%的reads是观察到两个重复的,~7%是观察到三次重复的,依此类推。
可以想象,如果原始数据很大(事实往往如此),做这样的统计将非常慢,所以fastqc中用fq数据的前200,000条reads统计其在全部数据中的重复情况。重复数目大于等于10的reads被合并统计,这也是为什么我们看到上图的最右侧略有上扬。大于75bp的reads只取50bp(不知道怎么选的)进行比较。但由于reads越长越不容易完全相同(由测序错误导致),所以其重复程度仍有可能被低估。
当非unique的reads占总数的比例大于20%时,报"WARN";当非unique的reads占总数的比例大于50%时,报"FAIL“。
10 Overrepresented Sequences
如果有某个序列大量出现,就叫做over-represented。fastqc的标准是占全部reads的0.1%以上。和上面的duplicate analysis一样,为了计算方便,只取了fq数据的前200,000条reads进行统计,所以有可能over-represented reads不在里面。而且大于75bp的reads也是只取50bp。如果命令行中加入了-c contaminant file,出现的over-represented sequence会从contaminant_file里面找匹配的hit(至少20bp且最多一个mismatch),可以给我们一些线索。
当发现超过总reads数0.1%的reads时报”WARN“,当发现超过总reads数1%的reads时报”FAIL“。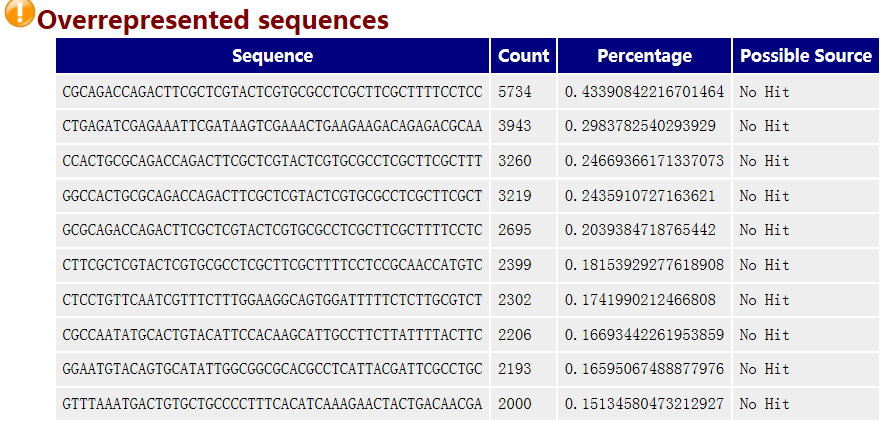
11 Overrepresented Kmers
如果某k个bp的短序列在reads中大量出现,其频率高于统计期望的话,fastqc将其记为over-represented k-mer。默认的k = 5,可以用-k --kmers选项来调节,范围是2-10。出现频率总体上3倍于期望或是在某位置上5倍于期望的k-mer被认为是over-represented。fastqc除了列出所有over-represented k-mers,还会把前6个的per base distribution画出来。
kmer_profiles
当有出现频率总体上3倍于期望或是在某位置上5倍于期望的k-mer时,报”WARN“;当有出现频率在某位置上10倍于期望的k-mer时报"FAIL"。