From:
https://mp.weixin.qq.com/s/4dtXwK1KTLXndGvO5m2BEQ
单细胞测序主要涉及单细胞基因组测序和转录组测序两个方面,分别针对单细胞DNA及RNA进行序列分析和比较。单细胞测序一直是科学家关注的一个热点。单细胞基因组测序目前主要有两种扩增方法:MALBAC方法及MDA方法,而单细胞转录组目前主要有三种方法:SMART扩增技术、10×genomics技术及Andeplete技术。
要实现单细胞转录组测序,需要解决2个难题:
1.PCR偏差:单个细胞含有约10pg total RNA,而约80%以上信息为rRNA,从单细胞RNA到文库意味着核酸的扩增量要达到百万倍以上。而在这个高的扩增量不引入PCR偏差一直是个较大的问题。
我们可以想一下,如果两个样本基因表达量是相同的,但扩增效率A是99%,B是97%,在扩增30个循环后,两者在扩增后的表达就有了1.84(0.99^30/0.97^30)倍的差异。而当我们分析差异基因的时候如果选1.5倍作为差异基因的标准,那么本来没有差异的基因也会出现差异。
2.去除rRNA:rRNA(核糖体RNA)在total RNA占比一般在80%以上,如果不加区分地进行逆转录,再扩增、建库很可能测序得到的绝大部分序列都是rRNA的序列,但是一般情况下,如果你更关心mRNA等编码基因的序列,rRNA序列不能给我们带来有效的信息,可以说它是无用的。
下面我们来分别介绍单细胞转录组的三个扩增技术:
-
SMART扩增技术:
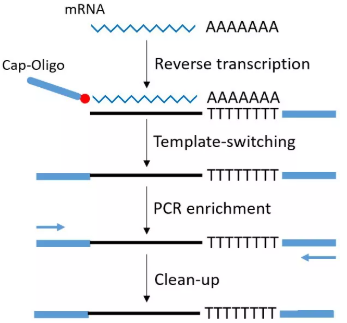
SMART扩增技术最核心的技术,就是设计了2个特殊的引物。再配合用MMLV逆转录酶进行逆转录。
特殊引物1由中间PolyT序列加上一段通用序列及3’末端两个简并碱基构成,但在PolyT的3’端倒数第二个碱基是A、C、G而非T的简并碱基,而倒数第一个为简并碱基,这样做的好处是让它正好结合在mRNA的3’端连到Poly(A)尾巴的这个连接处,而不会结合到mRNA的别的地方。这样就保证了逆转录的起始位置正好是mRNA的3’端的序列终止位置。MMLV逆转录酶,这个酶有个特点,就是它在转录到mRNA的5’端末端的时侯,会在新合成的cDNA的3’末端,多加出几个C碱基来。
特殊引物2由一段通用序列及它的3’端是3个非脱氧的G碱基构成,也就是核糖核酸的、RNA的G碱基,而不是DNA的G碱基,这个引物可以与刚才新合成的cDNA的3’端的那几个C碱基发生互补杂交,然后引导这个MMLV酶再次发挥聚合作用,以刚才那条新合成的cDNA为模板,复制的结果,就是得到双链的cDNA。
这个双链cDNA,两端都已经接好了我们人工设计的PCR引物序列,然后,就加入常规的PCR引物,进行常规的PCR扩增,常规PCR扩增,得到大量DNA。然后可以象常规的DNA建库那样,超声打断、建库、上机测序了。
通过SMART技术得到的主要是mRNA信息,LncRNA信息大部分会丢失,SMART技术对于RNA的质量要求较高,如果RNA出现降解会导致mRNA 5’端信息丢失。通用引物技术能保证扩增的均一性,但PCR引入的突变不能够分析出来。
-
10×genomics技术:
首先在凝胶微珠上种上特定的DNA片段,DNA片段由三部分组成:Barcode、UMI、PolyT组成。Barcode是16个碱基的长度。一共有400万种Barcode,一个微珠是对应于一种Barcode,通过这400万种Barcode,可以把凝胶微珠给区分开(通过barcode可以把cell分开)。UMI是一段随机序列,也就是说每一个DNA分子,都有自己的UMI序列(一个微珠上有很多种UMI,通过UMI可以把RNA分子分开,并可以标记RNA分子的数量)。10个碱基长的UMI,有100万种序列的变化(4^10 = 1,048,576),UMI的作用是为了区分哪些reads是来自于一个原始cDNA分子(???),区分基因片段重复还是duplication及区分是真实的SNP位点还是PCR产生的突变(???)。通过10×genomics仪器将单个细胞与单个凝胶微珠通过油相混在一起,形成油包水的小微滴,接下来把细胞膜破掉,让细胞当中的mRNA游离出来。游离出来的mRNA与小液滴中的水相混合,也就是和逆转录酶、结合在凝胶微珠上的核酸引物、以及dNTP底物相接触。
接着,发生逆转录反应。mRNA与凝胶微珠上带标签的DNA分子相结合,在逆转录酶的作用下,逆转录出cDNA来。把这个乳浊液当中所有的水相抽出来,也就是把所有带了标签的cDNA分子都抽出来,再把这些cDNA分子都加上接头,经过PCR扩增,做成illumina的测序文库,放到Illumina的测序仪上进行测序。测序完成之后,进行数据分析。
10×genomics技术一次可以同时得到大量细胞数据,但只能得到mRNA信息,LncRNA大部分信息丢失,UMI技术能很好去除认为分析引入duplication及PCR引入SNP位点。同样对RNA质量要求高,降解同样会引起5’端信息丢失。
-
Anydeplete 技术

Anydeplete技术首先通过随机引物进行一链合成,一链合成引入核苷酸类似物,用于酶切打断,二链合成同样引入核苷酸类似物用于保证链特异性。然后两端加上接头,接头一条链也带有核苷酸类似物,用于酶切降解。当形成单链文库后,设计特异性引物与rRNA形成文库结合,一轮退火延伸,rRNA文库形成双链结构。Reverse adaptor上带有特异的酶切位点,当形成双链结构酶切位点被识别,切去接头,这样rRNA形成的文库不带有完整的接头,而其他文库带有完整接头,通过PCR扩增富积既能得到想要的信息,包含mRNA及LncRNA信息。同样Anydeplete技术与10×genomics技术一样,包含分子标签,可分析duplication及PCR产生突变位点。
Anydeplete技术能够用于降解性样本,保证5’端及3’端信息的完整,能同时得到mRNA及LncRNA信息,如果只希望得到mRNA信息,Anydeplete技术则会引起一部分数据浪费。
总结:
-
SMART技术可用于单细胞mRNA测序,对RNA质量要求高,RNA降解会引起5’端信息丢失,没有分子标签功能。
-
10×genomics技术可用于单细胞mRNA测序,对RNA质量要求高,RNA降解会引起5’端信息丢失,有分子标签功能。
-
Anydeplete技术可用于单细胞mRNA及LncRNA测序,对RNA质量要求不高,可用于降解性样本测序。