Gao Y, Yang Z, Yang W, et al. Plant-ImputeDB: an integrated multiple plant reference panel database for genotype imputation[J]. Nucleic Acids Research, 2020.
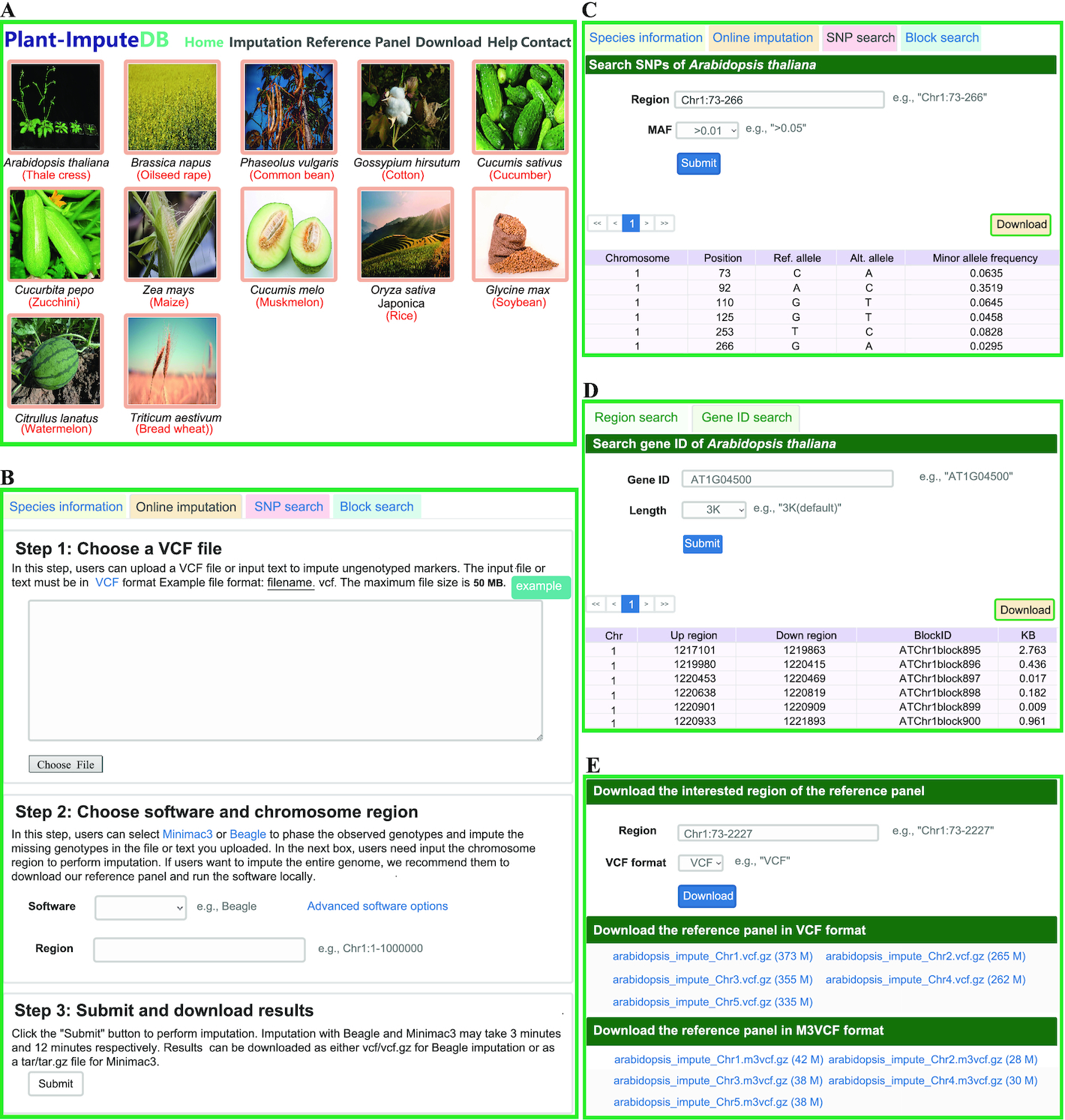
本文作者首先构建了一个包含了不同作物的reference panel,随后根据该panel搭建了Plant-ImputeDB数据库。该panel包含12种植物。数据库支持在线基因型估计及SNP、block的搜索与下载。同时,支持在两种知名的imputation软件间进行选择。
下表展示了panel中包含的物种。对于12种中的10种,从数据库或研究中下载了原始基因型文件(VCF格式)。5种(拟南芥,菜豆,玉米,西瓜)的样品使用WGS进行基因分型3; 三个物种(黄瓜,甜瓜和西葫芦)的样品与高通量GBS进行基因分型; 对于面包小麦,使用外显子组捕获测序技术对样品进行基因分型;对于大豆,样品采用SoySNP50K Illumina Infinium II BeadChip进行基因分型。对于其他两种物种,油菜,棉花,原始数据集的测序从NCBI数据库以登录号SRP155312和SRP115740提供下载。
利用原始的测序数据,使用Sentieon pipeline鉴定出了高质量的SNP。

下图展示了该数据库的工作流程。其大致分为数据收集(A)、数据处理(B)、数据库内容和网页前端(C-F)。
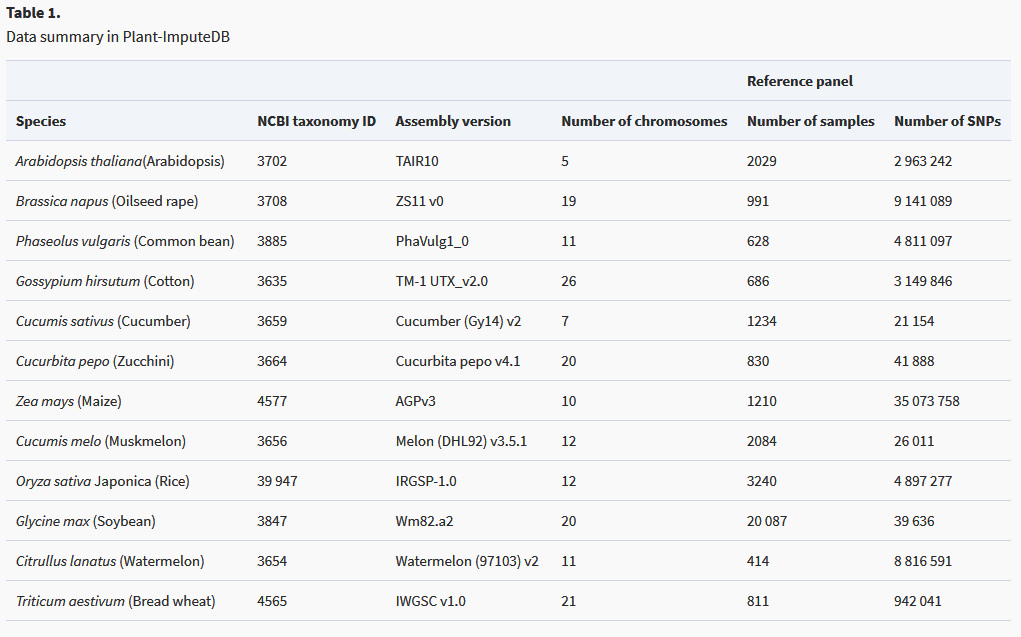
在基因型插补软件Beagle、Minimac3和Impute2之间,作者选择了Beagle和Minimac3进行reference panel的搭建。Beagle (v5.1)和Minimac3都基于通用隐马尔可夫模型框架,而Impute2基于Markov链蒙特卡洛框架。
首先,Beagle对于筛选后的SNP(MAF > 0.01, call rate > 0.5)采用默认参数进行reference panel的生成,然后采用Minimac3将VCF格式转换为M3VCF格式。
随后,作者对插补结果进行了测试。首先,作者进行了5倍交叉验证策略计算了所有物种的估算准确性:对于每个物种,参考面板中的所有样本均随机分为5份(fold),其中一份被选为研究种群,其余样本均用于构建panel。考虑到大多数商业化array的特点,作者从研究群体的整个基因组中随机选择了100000个SNP,并掩盖了其他SNP。然后,使用Beagle和Minimac3插入具有默认参数的基因型,进而得到array的模拟数据和对模拟数据进行插补的结果。插补的SNP中, MAF ≥ 0.01 and estimated squared correlation ≥ 0.3的得到了保留
对于模拟数据与真实数据,作者进行了对比,并计算了concordance rate (CR)(通过将正确估算的基因型的数量除以每个物种估算的基因型的总数来计算CR)和(R^2)(真实基因型和估算的基因型之间的平方相关性),以此评价插补性能。估算后,研究群体SNP的数量平均增加了34.47倍。所有测试物种的平均CR均大于0.88。Beagle的平均R 2从甜瓜的0.76到棉花的0.96,而Minimac3的平均R 2从甜瓜的0.76到普通豆的0.97。
此外,作者还使用不同密度的模拟数据集和独立的数据集来评估参考panel的估算准确性。首先,对于我们数据库中的12个物种,作者按照 Friedrich, J. et al.[1]的模拟方法从50%到95%中随机选择了10种不同百分比的被掩盖SNP的100个样品,通过比较插补结果和原始基因型来计算插补准确性。对于两个插补工具Beagle和Minimac3,所有模拟数据集的平均准确度范围为0.83至0.99(补充图3,4)。其次,作者还对数据库中相应物种的九个独立验证集,包括水稻,拟南芥,玉米,油菜,棉花,大豆,黄瓜,甜瓜和面包小麦进行了测序、插补,以评估插补精度(补充图5, 6)。所有这些验证结果表明,参考面板和插补工具可以相对较高的精度用于不同群体的基因型插补。
数据库开发采用了Flask (version 1.1.1) 和AngularJS (version 1.6.1),运行在Apache 2 web server (version 2.4.18) 上,并采用了 MongoDB (version 3.4.2)作为数据库引擎
其他参考文献
Friedrich J., Antolin R., Edwards S.M., Sanchez-Molano E., Haskell M.J., Hickey J.M., Wiener P. Accuracy of genotype imputation in Labrador Retrievers. Anim. Genet. 2018;49:303–311. ↩︎