核化这个概念在很多机器学习方法中都有应用,如SVM,PCA等。在此结合sklearn中的KPCA说说核函数具体怎么来用。
KPCA和PCA都是用来做无监督数据处理的,但是有一点不一样。PCA是降维,把m维的数据降至k维。KPCA恰恰相反,它是把m维的数据升至k维。但是他们共同的目标都是让数据在目标维度中(线性)可分,即PCA的最大可分性。
在sklearn中,kpca和pca的使用基本一致,接口都是一样的。kpca需要指定核函数,不然默认线性核。
首先我们用下面的代码生成一组数据。
import numpy as np from sklearn.decomposition import PCA, KernelPCA import matplotlib.pyplot as plt import math x=[] y=[] N = 500 for i in range(N): deg = np.random.randint(0,360) if np.random.randint(0,2)%2==0: x.append([6*math.sin(deg), 6*math.cos(deg)]) y.append(1) else: x.append([15*math.sin(deg), 15*math.cos(deg)]) y.append(0) y = np.array(y) x = np.array(x) print('ok')
这些数据可以用下图来表示
显然,我们选的正样本(蓝)都落在一个半径为6的圆上,负样本(红)全选在一个半径为15的圆上。
这样的数据显然是线性不可分的。如果我们强行要用线性分类器来做,可以对原始数据做一个kpca处理。
如下代码:
kpca = KernelPCA(kernel="rbf", n_components=14) x_kpca = kpca.fit_transform(x)
我们用rbf核,指定维度为14(为啥是14,后面说),也就是说吧X(2维)中的数据映射到14维的空间中去。
然后我们用一个线性的SVM分类器来对映射后的数据做一个分类,随机取80%做训练集,20%做测试集。
from sklearn import svm clf = svm.SVC(kernel='linear') clf.fit(x_kpca[:0.8*N],y[:0.8*N]) y0 = y[0.8*N:] y1 = clf.predict(x_kpca[0.8*N:]) print(np.linalg.norm(y0-y1, 1))
此时,输出的是0,也就是说我们预测出的分类与正确的分类完全吻合,没有一个错误。此时分类器在这个数据集上是完全有效的。
综上所述,通过核化,可以把原本线性不可分的数据映射到高维空间后实现线性可分。
不过这个过程也不完全是那么简单,我们取不同的维度得到的结果可能相差甚远,也就是经典的调参问题。
在此,我测试了几组不同的k取值,得到不同k值条件下分类准确率值。如下图。
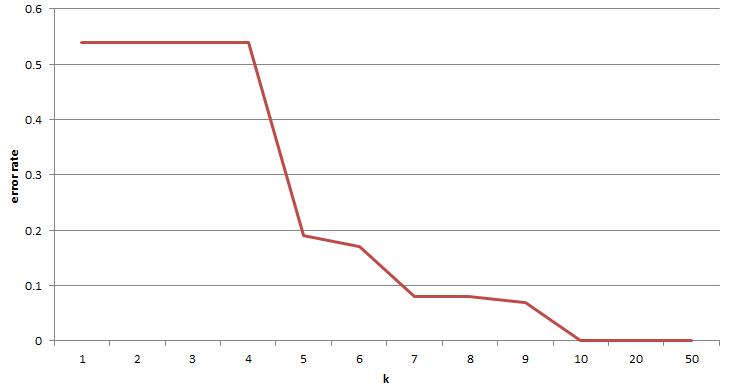
可以看出,不同的k值对结果是有影响的。但不同的数据集可能不一样,所以需要进行调参。我这个取14就全ok了。