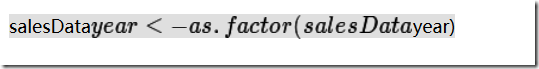一些函数不知道什么意思要查,看数值例子,做笔记,知道函数的功能,函数和返回值。
网页上查找关键词,巧用查找(ctrl+F)
数据读取处理,有read.table read
R—读取数据(导入csv,txt,excel文件)
read.table函数:read.table函数以数据框的格式读入数据,所以适合读取混合模式的数据,但是要求每列的数据数据类型相同。
read.table读取数据非常方便,通常只需要文件路径、URL或连接对象就可以了,也接受非常丰富的参数设置:
- file参数:这是必须的,可以是相对路径或者绝对路径(注意:Windows下路径要用斜杠'/'或者双反斜杠'\')。
- header参数:默认为FALSE即数据框的列名为V1,V2...,设置为TRUE时第一行作为列名。
data1<-read.table('item.csv')#默认header=FALSE
data2<-read.table('item.csv',header=TRUE)
- read.csv、read.csv2、read.delim是read.table函数的包装,分隔符分别对应逗号,分号,制表符,同样接受read.table所有参数。
- read.csv函数header参数默认为TRUE,不同于read.table。
data3<-read.csv('item.csv',sep=',',header=TRUE)
data4<-read.table('item.csv')
#下文示例采用read.csv函数,两种写法效果相同
# head(gc.data)
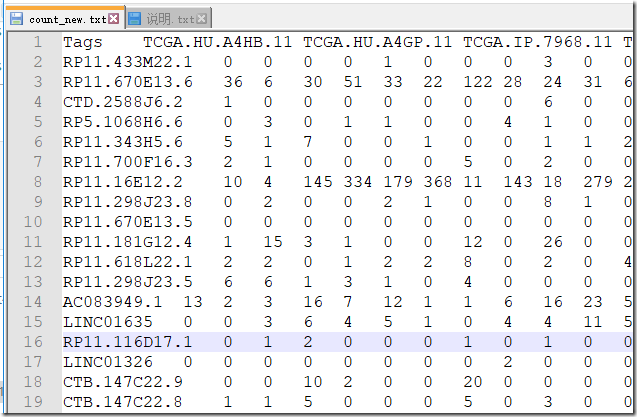
count数据
原数据csv转成txt后再做
library("DESeq2")
coldata<-data.frame(c(rep("normal",30),rep("tumor",377)))
"condition"->names(coldata)
names(coldata)
rownames(coldata) = colnames(exprSet)
rownames() 行名 colnames() 列名
gctestnew=gc.data[seq(1,10),seq(1,4)]
exprSet = as.matrix(gc.data[,-1])
矩阵提取相应的行,现在还在越界,已解决:
id=up_diff_result$Row.names
name=gc.data[id,1]
代码的形式更改镜像下载包
source("http://bioconductor.org/biocLite.R") options(BioC_mirror="http://mirrors.ustc.edu.cn/bioc/") biocLite('WGCNA')
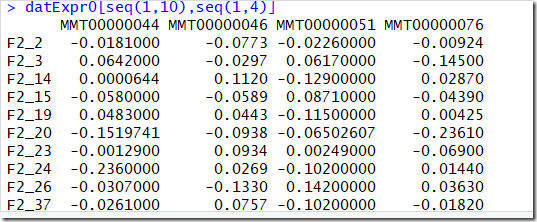
转置 as.data.frame datExpr0 = as.data.frame(t(femData[, -c(1:8)])); #去除数据 1 到 8 列 转置后生成数据框。 names(datExpr0) = femData$substanceBXH;
R语言的一些基础补充
sampleTree = hclust(dist(datExpr0), method = "average");
sampleTree = hclust(dist(datExpr0), method = "average");
https://blog.csdn.net/sherrymi/article/details/38341185
dim(femData); #查看数组(矩阵)的大小
plot(sampleTree,….)
https://www.cnblogs.com/wutongyuhou/p/5873056.html
sizeGrWindow(width, height)
width desired width of the window, in inches.
height desired heigh of the window, in inches.
datExpr0[seq(1,10),seq(1,4)] #显示1-10行,1-4列
datExpr0 = as.data.frame(t(femData[, -c(1:8)])); #去除数据 1 到 8 列 转置后生成数据框。
nGenes = ncol(datExpr) # 3600L? 多少列
#郭:例子:matrix(1:20,nrow=4,ncol=5,byrow=FALSE) 分析: 矩阵A的数据是1-20,在R语言中1-20就是1:20,4*5的意思就是4行5列。
nSamples = nrow(datExpr) # 多少列
F2_2 行 样本134, MMT0000044 列 是基因3600 134*3600 排序了
R语言 sub与gsub函数的区别
> text <- c("we are the world", "we are the children")
> sub("w", "W", text)
[1] "We are the world" "We are the children"
> sub("W","w",text)
[1] "we are the world" "we are the children"
> gsub("W","w",text)
[1] "we are the world" "we are the children"
> gsub("w","W",text)
[1] "We are the World" "We are the children"
> sub(" ", "", "abc def ghi")
[1] "abcdef ghi"
> ## [1] "abcdef ghi"
> gsub(" ", "", "abc def ghi")
[1] "abcdefghi"
> ## [1] "abcdefghi"
从上面的输出结果可以看出,sub()和gsub()的区别在于,前者只替换第一次匹配的字符串,而后者会替换掉所有匹配的字符串。
R子集subset
> x<-c(6,1,2,3,NA,12)
> x[x>5] #x[5]是未知的,因此其值是否大于5也是未知的
[1] 6 NA 12
> subset(x,x>5) #subset直接会把NA移除
[1] 6 12
> subset(airquality, Temp > 80, select = c(Ozone, Temp))
Ozone Temp
29 45 81
35 NA 84
36 NA 85
38 29 82
39 NA 87
40 71 90
...
> subset(airquality, Day == 1, select = -Temp)
Ozone Solar.R Wind Month Day
1 41 190 7.4 5 1
32 NA 286 8.6 6 1
62 135 269 4.1 7 1
93 39 83 6.9 8 1
124 96 167 6.9 9 1
...
> subset(airquality, select = Ozone:Wind)
Ozone Solar.R Wind
1 41 190 7.4
2 36 118 8.0
3 12 149 12.6
4 18 313 11.5
5 NA NA 14.3
就是把x中所有不是NA的值赋予x,比如说x=c(1,2,NA,4),那么运行这个程序
x<-x[!is.na(x)]
以后,x=c(1,2,4)
x<-“123”,x为character类型,而as.numeric(x)则为numeric类型的123。但是因子(factor)类型却不一样。
a<-factor(c(100,200,300,301,302,400,10)),它们的值分别为100 200 300 301 302 400 10,然而
as.numeric(a)对应的值并非100 200 300 301 302 400 10,而是2 3 4 5 6 7 1。因子(factor)转换成数值型(numeric)
的规则是这样的:
一共有n个数,那么转换后的数字就会在1——n中取值,数字最小的取一,次小的取二,以此类推。
那么如何让因子(factor)类型里的数值转换对应的数值型呢?
mean(as.numeric(as.character(factorname)))
mean(as.numeric(levels(factorname)[factorname]))
---------------------
作者:记录本
来源:CSDN
原文:https://blog.csdn.net/jiluben/article/details/40222229
版权声明:本文为博主原创文章,转载请附上博文链接!
> c(1:5) [1] 1 2 3 4 5
> list(a=1,b=2,c=3) $`a` [1] 1 $b [1] 2 $c [1] 3
> 5^5 [1] 3125
R语言常用命令
https://www.cnblogs.com/mohuishou-love/p/10232347.html
- mapply
mapply(FUN, ..., MoreArgs=NULL, SIMPLIFY=TRUE, USE.NAMES=TRUE)
mapply是多变量版的sapply,参数(...)部分可以接收多个数据,mapply将FUN应用于这些数据的第一个元素组成的数组,然后是第二个元素组成的数组,以此类推。要求多个数据的长度相同,或者是整数倍关系。返回值是vector或matrix,取决于FUN返回值是一个还是多个。
> mapply(sum, list(a=1,b=2,c=3), list(a=10,b=20,d=30))
a b c
11 22 33
> mapply(function(x,y) x^y, c(1:5), c(1:5))
[1] 1 4 27 256 3125
> mapply(function(x,y) c(x+y, x^y), c(1:5), c(1:5))
[,1] [,2] [,3] [,4] [,5]
[1,] 2 4 6 8 10
[2,] 1 4 27 256 3125作者:Stone_Stan4d
链接:https://www.jianshu.com/p/5529887f4447
來源:简书
简书著作权归作者所有,任何形式的转载都请联系作者获得授权并注明出处。
函数(和上面的没关系):
days <- function(x, y){
if(is.na(x))return(y)
else return(x)
}
year <- c(2010,2012,2013,12) c 表示列
sales <-c(1203,1220,2230,45)
salesData <- data.frame(year,sales)
4行2列
用str(salesData)观察数据框结构时,year的类型是num.
year在这里的语义是:每年的销售额,显然year是个类别,因此转化为因子:
R里面的数字向量都默认为数值型的,类别型和顺序型向量都叫因子,但是得自己设定。日期数据其实也是数值型的,但是也得设定,设定成日期型后可以进行运算。
如何把factor因子取消 as.character
关于R语言中的”因子”变量类型(一)
https://blog.csdn.net/zhangxiaojiakele/article/details/80117602
因子变量实际上是由数值型变量构成的!
这一标题有没有令你感到惊讶呢!下面给你展示一个比较sao的操作,你先自行体会以下(运气好的人可能体会过,因为我看见有人在QQ群问过这种问题):
x <- c("10", "20", "30", "40")
as.numeric(x)
## [1] 10 20 30 40
y <- as.factor(x)
y
## [1] 10 20 30 40
## Levels: 10 20 30 40
as.numeric(y)
## [1] 1 2 3 4
看到了吗?我们对于x这一数值构成的字符型向量进行数值转化操作,得到了我们想要的结果.然而,当将x转化为因子型变量时,景观看起来外观没有什么大的差别,但我们进行数值转化操作的结果却并非我们想要的,而是一堆奇怪的1,2,3,4.事实上1,2,3,4恰恰表示的对应元素在因子型变量中的排序先后,即 levels属性的先后.对照levels属性或许让你能够更至关的进行观察:
order(levels(y))
## [1] 1 2 3 4
as.numeric(y)
## [1] 1 2 3 4
这也是为什么很多人看着眼前的数据似乎是一组字符型变量,想将其转化为数值型变量,但却发现为得到想要的结果,究其原因就在于这些变量并不是字符型变量,而是因子型变量.这一情况最容易出现在以下情形中:你的数据中某一列原本应当是数值型变量,但某几个取值缺失了,而缺失值的表示并不是简单的空值,而是其他如”-“等符号,此时在数据读取的过程中,这一列变量将会被自动转化为字符型向量,而如果你恰恰又不幸地忘记设置stringAsFactors = FALSE, 那么,便会产生如上情形了.任何情况下,都要仔细检查变量类型.上述问题的解决方法是先使用as.characer()将因子型变量转化为字符型变量,继而使用as.numeric()函数将其转化为数值型变量.
---------------------
作者:zhangxiaojiakele
来源:CSDN
原文:https://blog.csdn.net/zhangxiaojiakele/article/details/80117602
版权声明:本文为博主原创文章,转载请附上博文链接!
help('lapply')R语言中ifelse函数可以完成类似的if...else的分支功能,可以认为是紧凑的if...else结构。其基本语法格式如下:
if(con, statement1, statement2)
con是逻辑条件,当逻辑条件的值为TRUE时,则输出statement1的值,否则输出statement2的值。
第一个例子:
x<-3
y<-ifelse(x>0, 2*x+1, 2*x-1)
其执行的过程是,若x是个大于0的值,则输出2*x+1的值,否则为2*x - 1的值。
问题![]() 待解决)
待解决)
source('http://bioconductor.org/biocLite.R')
Error in file(filename, "r", encoding = encoding) :
cannot open the connection
In addition: Warning message:
In file(filename, "r", encoding = encoding) : InternetOpenUrl failed: ''
source("https://bioconductor.org/biocLite.R") InternetOpenUrl failed: '无法与服务器建立连接'
(1)看看IE能不能打开网址 http://www.bioconductor.org/
(2) https://blog.csdn.net/truffle528/article/details/77200710 不行options(repos=structure(c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/")))
source("https://bioconductor.org/biocLite.R")
R连接函数paste和paste0
paste0函数,默认是sep=""(两个函数的唯一区别)
#要生成12个月的fitbit文件名 paste("fitbit", 1:12, ".jpg", sep = "") [1] "fitbit1.jpg" "fitbit2.jpg" "fitbit3.jpg" "fitbit4.jpg" "fitbit5.jpg" "fitbit6.jpg" "fitbit7.jpg" [8] "fitbit8.jpg" "fitbit9.jpg" "fitbit10.jpg" "fitbit11.jpg" "fitbit12.jpg" #可以看出参数里面有向量时的捉对拼接的效果,如果某个向量较短,就自动补齐 a <- c("甲","乙","丙","丁","戊","己","庚","辛","壬","癸") b <- c("子","丑","寅","卯","辰","巳","午","未","申","酉","戌","亥") paste0(a, b) [1] "甲子" "乙丑" "丙寅" "丁卯" "戊辰" "己巳" "庚午" "辛未" "壬申" "癸酉" "甲戌" "乙亥"
paste与paste0还有一个collapse参数,可以把这些字符串拼成一个长字符串,而不是放在一个向量中
#collapse是合并成一个字符串时的分隔符 paste("fitbit", 1:3, ".jpg", sep = "", collapse = "; ") [1] "fitbit1.jpg; fitbit2.jpg; fitbit3.jpg"
R——文件与工作空间的设置
- getwd() 获取当前工作空间
- ls() 列出当前工作空间所有的对象
- dir() 列出与当前工作空间平行的文件
- args(list.files) 获取list.files()函数的参数
- old.dir<-getwd() 将当前工作空间保存在old.dir变量
- dir.create('testdir') 创建一个新的工作空间
- setwd('testdir') 将上面创建的工作空间设置为当前工作空间
- file.create('mytest.R') 在当前工作空间创建一个文件
- list.files() 列出所有文件
- file.exists('mytest.R') 建议该文件是否存在
- file.rename('mytest.R','mytest2.R') 重命名
- file.copy('mytest2.R','mytest3.R') 复制
- file.path('mytest3.R') 获取路径
- dir.create(file.path('testdir2','testdir3'),recursive=TRUE) 以不覆盖方式创建工作空间
- unlink('testdir2',recursive=TRUE) 删除工作空间
- setwd(old.dir) 还原工作空间
- unlink('testdir',recursive=TRUE) 继续删除工作空间
R中的路径设置
1.使用getwd()函数来显示当前工作目录。
> getwd()
[1] "C:/Users/Administrator/Documents"
2.使用setwd()函数更改当前目录。
> dir.create("E:/R_Files")
> setwd("E:/R_Files")
> getwd()
[1] "E:/R_Files"
注意:
(1) 函数setwd()不会自动创建一个不存在的目录。如果必要的话,可以使用函数dir.create()来创建新目录,然后使用setwd()将工作目录指向这个新目录。
(2)R里的dir.create()是不级联的,就是说一次只能创建有一个“/”的路径。如果需要两个“/”的路径(”E:/R_Files/R1”),就需要用dir.create()创建两次,然后文件才创建成功。再按照例子用setwd()去设置即可。
(3)函数setwd()是设置临时的工作路径(也就是说一旦你关闭了Rstudio后,工作目录又变回原来的工作目录)。
---------------------
作者:语希丫头
来源:CSDN
原文:https://blog.csdn.net/mona_sunshine/article/details/53042413
版权声明:本文为博主原创文章,转载请附上博文链接!
getGEO(GEO = NULL, filename = NULL, destdir = tempdir(), GSElimits=NULL,
GSEMatrix=TRUE,AnnotGPL=FALSE)
例如:
gds <- getGEO("GDS10") 会返回一个对象,而且下载数据一般会在tmp目录下面,当然如果你需要保存这些文件,你可以自己制定下载目录及文件名。
gse2553 <- getGEO("GSE2553")
GDS2eSet函数可以把上面这个下载函数得到的对象(要确定是GDS而不是GSE)变成表达对象
pData和exprs函数都可以处理上面这个表达对象,从而分别得到样品描述矩阵和样品表达量矩阵
综合一起就是
g4100 <- GDS2eSet(getGEO("GDS4100"))
g4102 <- GDS2eSet(getGEO("GDS4102"))
e4102<-exprs(g4102)
e4100<-exprs(g4100)
这样的代码,这个e4100和e4102就都是一个数值矩阵啦,可以进行下游分析,但是如果是下载的GSM数据
就用下面这个代码,GSE26253_series_matrix.txt是通过GSEMatrix=TRUE这个参数特意下载到你的目录的
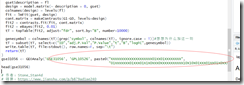 直接在下面加上这个语句,看出现什么结果。上面的funtion是一个函数,这个是调用那个函数的,在这中间会下载那个数据,最后得出return(tT)
直接在下面加上这个语句,看出现什么结果。上面的funtion是一个函数,这个是调用那个函数的,在这中间会下载那个数据,最后得出return(tT)
https://www.jianshu.com/p/b879ad1ae240
我估计会下载很长时间,我看这两个数据包都很大'GSE31056', 'GPL10526'
这里好像是一个在线GEO分析的工具
http://www.360doc.com/content/16/1030/23/19913717_602657026.shtml
转我的博客
???
GSE26253
Platforms (1) GPL8432 Illumina HumanRef-8 WG-DASL v3.0
R语言提供了批量处理函数,可以循环遍历某个集合内的所有或部分元素,以简化操作。
这些函数底层是通过C来实现的,所以效率也比手工遍历来的高效。
批量处理函数有很重要的apply族函数:lapply sapply apply tapply mapply。apply族函数是高效能计算的运算向量化(Vectorization)实现方法之一,比起传统的for,while常常能获得更好的性能。
apply : 用于遍历数组中的行或列,并且使用指定函数来对其元素进行处理。
lapply : 遍历列表向量内的每个元素,并且使用指定函数来对其元素进行处理。返回列表向量。
sapply : 与lapply基本相同,只是对返回结果进行了简化,返回的是普通的向量。
mapply: 支持传入两个以上的列表。
tapply: 接入参数INDEX,对数据分组进行运算,就和SQL中的by group一样。
---------------------
作者:wa2003
来源:CSDN
原文:https://blog.csdn.net/wa2003/article/details/45887055
版权声明:本文为博主原创文章,转载请附上博文链接!
R语言-attach、detach、with
with(onedata.frame,{ name })
R语言reads.table 自动将字符串变成了逻辑值
今天遇到了一个问题,文件中有一列的值为全为F, 用read.table 读取的时候,自动将F 变成了false
对于这样的转换,可以通过 colClass 参数控制
colClass 参数指定每一列的类型,numeric, integer, character, logical 等等,只需要将全是F的那一列指定为 character 就可以了
read.table( "file.txt" , sep = " ", header=FALSE, stringsAsFactors=FALSE, colClasses = c("character"))
其实在R里面还有一个内置的函数 .libPaths , 可以直接查看所有的路径
用法示例:
.libPaths() [1] "/usr/lib64/R/library"