使用humann2:
===================参考晨宇大大的学习文档==========================
HUMAnN2是描述微生物代谢通路的。使用宏基因组或宏转录组的序列信息。
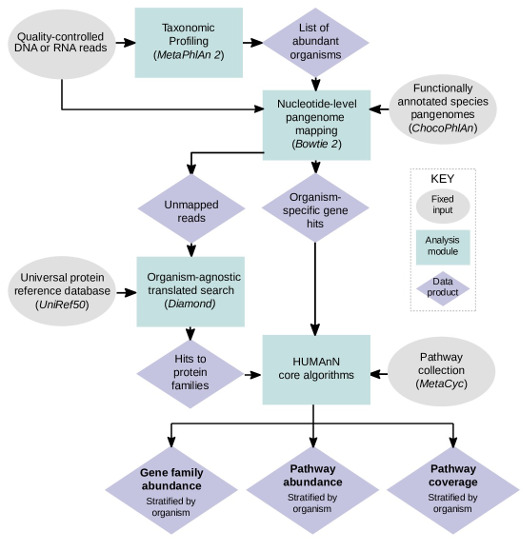
http://huttenhower.sph.harvard.edu/humann2
安装的顺序是:Bowtie2 ===> Metaphlan2 ===> HUMAnN2
(个人建议安装anaconda,里面集成了多个python包,安装其他软件的时候就不用担心缺少什么numpy、scipy这些额外的python包了。)
安装没什么可讲的,下载安装包(源码),解压,设置环境变量就可以了。要注意的是HUMAnN2依赖于两个数据库(ChocoPhlAn和UniRef),他的安装包里面只有这两个数据库的DEMO版本。所以我们首先要下载完整的版本。使用迅雷,简单粗暴(微笑脸)
ChocoPhlAn:http://huttenhower.sph.harvard.edu/humann2_data/chocophlan/full_chocophlan_plus_viral.v0.1.1.tar.gz
配置:
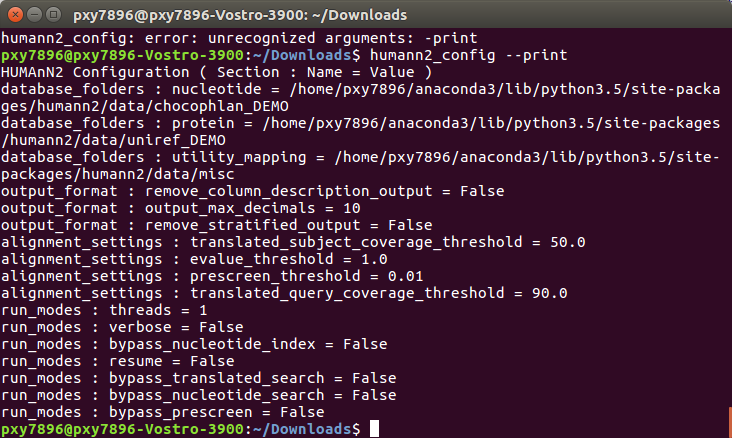
我用迅雷下载的文件有问题,解压之后是空的==所以使用命令下载:
humann2_databases --download chocophlan full /home/pxy7896/Downloads/humann2/humann2/data/chocophlan_FULL4
该命令会自动配置数据库的路径,所以放着就可以了。
IDs="G45084 G45072 G45071 G45109 G45125 G45124 G45049 G45054 G45121 G45099"
for s in ${IDs}
do
humann2 --input ${s}_pe_1.fastq.gz --output /home/pxy7896/Desktop/20161205/result2
humann2 --input ${s}_pe_2.fastq.gz --output /home/pxy7896/Desktop/20161205/result2
done
运行了一晚,产生了很多很多文件。。。