class sklearn.linear_model.LogisticRegression (penalty=’l2’, dual=False, tol=0.0001, C=1.0,fifit_intercept=True, intercept_scaling=1, class_weight=None, random_state=None, solver=’warn’, max_iter=100,multi_class=’warn’, verbose=0, warm_start=False, n_jobs=None)
1 二元逻辑回归的损失函数
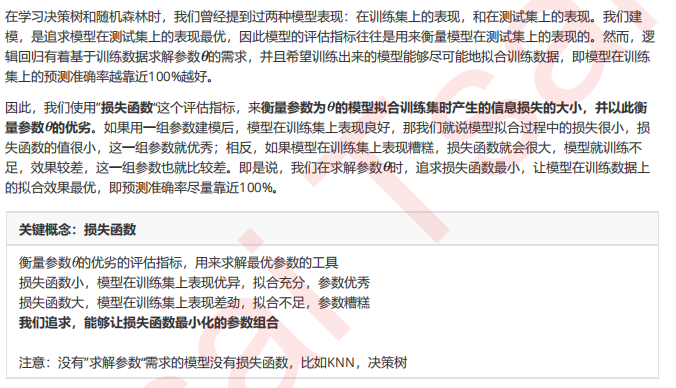
1.1 损失函数的概念与解惑

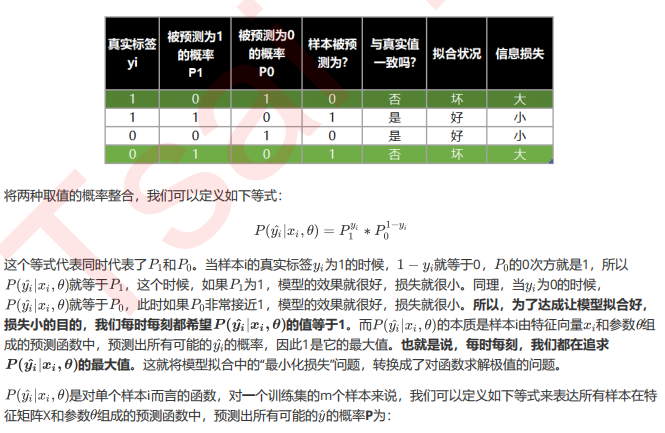
1.2【选学】二元逻辑回归损失函数的数学解释,公式推导与解惑



2 重要参数penalty & C
2.1 正则化



from sklearn.linear_model import LogisticRegression as LR from sklearn.datasets import load_breast_cancer import numpy as np import matplotlib.pyplot as plt from sklearn.model_selection import train_test_split from sklearn.metrics import accuracy_score data = load_breast_cancer() X = data.data y = data.target data.data.shape lrl1 = LR(penalty="l1",solver="liblinear",C=0.5,max_iter=1000) lrl2 = LR(penalty="l2",solver="liblinear",C=0.5,max_iter=1000) #逻辑回归的重要属性coef_,查看每个特征所对应的参数 lrl1 = lrl1.fit(X,y) lrl1.coef_ (lrl1.coef_ != 0).sum(axis=1) lrl2 = lrl2.fit(X,y) lrl2.coef_
可以看见,当我们选择L1正则化的时候,许多特征的参数都被设置为了0,这些特征在真正建模的时候,就不会出现在我们的模型当中了,而L2正则化则是对所有的特征都给出了参数。
究竟哪个正则化的效果更好呢?还是都差不多?
l1 = [] l2 = [] l1test = [] l2test = [] Xtrain, Xtest, Ytrain, Ytest = train_test_split(X,y,test_size=0.3,random_state=420) for i in np.linspace(0.05,1,19): lrl1 = LR(penalty="l1",solver="liblinear",C=i,max_iter=1000) lrl2 = LR(penalty="l2",solver="liblinear",C=i,max_iter=1000) lrl1 = lrl1.fit(Xtrain,Ytrain) l1.append(accuracy_score(lrl1.predict(Xtrain),Ytrain)) l1test.append(accuracy_score(lrl1.predict(Xtest),Ytest)) lrl2 = lrl2.fit(Xtrain,Ytrain) l2.append(accuracy_score(lrl2.predict(Xtrain),Ytrain)) l2test.append(accuracy_score(lrl2.predict(Xtest),Ytest)) graph = [l1,l2,l1test,l2test] color = ["green","black","lightgreen","gray"] label = ["L1","L2","L1test","L2test"] plt.figure(figsize=(6,6)) for i in range(len(graph)): plt.plot(np.linspace(0.05,1,19),graph[i],color[i],label=label[i]) plt.legend(loc=4) #图例的位置在哪里?4表示,右下角 plt.show()
可见,至少在我们的乳腺癌数据集下,两种正则化的结果区别不大。但随着C的逐渐变大,正则化的强度越来越小,模型在训练集和测试集上的表现都呈上升趋势,直到C=0.8左右,训练集上的表现依然在走高,但模型在未知数据集上的表现开始下跌,这时候就是出现了过拟合。我们可以认为,C设定为0.8会比较好。在实际使用时,基本就默认使用l2正则化,如果感觉到模型的效果不好,那就换L1试试看。
2.2 逻辑回归中的特征工程


高效的嵌入法embedded

from sklearn.linear_model import LogisticRegression as LR from sklearn.datasets import load_breast_cancer import numpy as np import matplotlib.pyplot as plt from sklearn.model_selection import cross_val_score from sklearn.feature_selection import SelectFromModel data = load_breast_cancer() data.data.shape LR_ = LR(solver="liblinear",C=0.9,random_state=420) cross_val_score(LR_,data.data,data.target,cv=10).mean() X_embedded = SelectFromModel(LR_,norm_order=1).fit_transform(data.data,data.target) X_embedded.shape cross_val_score(LR_,X_embedded,data.target,cv=10).mean()

fullx = [] fsx = [] threshold = np.linspace(0,abs((LR_.fit(data.data,data.target).coef_)).max(),20) k=0 for i in threshold: X_embedded = SelectFromModel(LR_,threshold=i).fit_transform(data.data,data.target) fullx.append(cross_val_score(LR_,data.data,data.target,cv=5).mean()) fsx.append(cross_val_score(LR_,X_embedded,data.target,cv=5).mean()) print((threshold[k],X_embedded.shape[1])) k+=1 plt.figure(figsize=(20,5)) plt.plot(threshold,fullx,label="full") plt.plot(threshold,fsx,label="feature selection") plt.xticks(threshold) plt.legend() plt.show()
然而,这种方法其实是比较无效的,大家可以用学习曲线来跑一跑:当threshold越来越大,被删除的特征越来越多,模型的效果也越来越差,模型效果最好的情况下需要保证有17个以上的特征。实际上我画了细化的学习曲线,如果要保证模型的效果比降维前更好,我们需要保留25个特征,这对于现实情况来说,是一种无效的降维:需要30个指标来判断病情,和需要25个指标来判断病情,对医生来说区别不大。
2)第二种调整方法,是调逻辑回归的类LR_,通过画C的学习曲线来实现:
fullx = [] fsx = [] C=np.arange(0.01,10.01,0.5) for i in C: LR_ = LR(solver="liblinear",C=i,random_state=420) fullx.append(cross_val_score(LR_,data.data,data.target,cv=10).mean()) X_embedded = SelectFromModel(LR_,norm_order=1).fit_transform(data.data,data.target) fsx.append(cross_val_score(LR_,X_embedded,data.target,cv=10).mean()) print(max(fsx),C[fsx.index(max(fsx))]) plt.figure(figsize=(20,5)) plt.plot(C,fullx,label="full") plt.plot(C,fsx,label="feature selection") plt.xticks(C) plt.legend() plt.show()
继续细化学习曲线:
fullx = [] fsx = [] C=np.arange(6.05,7.05,0.005) for i in C: LR_ = LR(solver="liblinear",C=i,random_state=420) fullx.append(cross_val_score(LR_,data.data,data.target,cv=10).mean()) X_embedded = SelectFromModel(LR_,norm_order=1).fit_transform(data.data,data.target) fsx.append(cross_val_score(LR_,X_embedded,data.target,cv=10).mean()) print(max(fsx),C[fsx.index(max(fsx))]) plt.figure(figsize=(20,5)) plt.plot(C,fullx,label="full") plt.plot(C,fsx,label="feature selection") plt.xticks(C) plt.legend() plt.show() #验证模型效果:降维之前 LR_ = LR(solver="liblinear",C=6.069999999999999,random_state=420) cross_val_score(LR_,data.data,data.target,cv=10).mean() #验证模型效果:降维之后 LR_ = LR(solver="liblinear",C=6.069999999999999,random_state=420) X_embedded = SelectFromModel(LR_,norm_order=1).fit_transform(data.data,data.target) cross_val_score(LR_,X_embedded,data.target,cv=10).mean() X_embedded.shape
这样我们就实现了在特征选择的前提下,保持模型拟合的高效,现在,如果有一位医生可以来为我们指点迷津,看看剩下的这些特征中,有哪些是对针对病情来说特别重要的,也许我们还可以继续降维。当然,除了嵌入法,系数累加法或者包装法也是可以使用的。
比较麻烦的系数累加法
系数累加法的原理非常简单。在PCA中,我们通过绘制累积可解释方差贡献率曲线来选择超参数,在逻辑回归中我们可以使用系数coef_来这样做,并且我们选择特征个数的逻辑也是类似的:找出曲线由锐利变平滑的转折点,转折点之前被累加的特征都是我们需要的,转折点之后的我们都不需要。不过这种方法相对比较麻烦,因为我们要先对特征系数进行从大到小的排序,还要确保我们知道排序后的每个系数对应的原始特征的位置,才能够正确找出那些重要的特征。如果要使用这样的方法,不如直接使用嵌入法来得方便。
简单快速的包装法
相对的,包装法可以直接设定我们需要的特征个数,逻辑回归在现实中运用时,可能会有”需要5~8个变量”这种需求,包装法此时就非常方便了。不过逻辑回归的包装法的使用和其他算法一样,并不具有特别之处,因此在这里就不在赘述,具体大家可以参考03期:数据预处理和特征工程中的代码。