在以前,我讲过如何利用3ds MAX中的阵列进行DNA双螺旋结构的创建,但是有与MAXScript也能够实现在3DS MAX界面中的大部分功能,而且得到的结构更加的精确,所以这一片博客中我主要讲述,如何利用MAXScript进行DNA双螺旋结构的创建。
首先按照阵列中的做法,我先查找如何利用Listener进行MAXScript的代码的编写,发现在3DS MAX中轴的操作以及阵列的操作不能够被准确的记录在宏记录器中,所以我就利用数学上的计算实现了DNA双螺旋结构的创建,发现其实阵列说白了就是利用MAXScript中的循环来减少用户在3DS MAX 中的复杂而重复的操作,从而来方便用户在3DS MAX界面中进行一些模型的构建与编辑,那我们学习与了解了3DS MAX中的内置语言MAXScript之后我们就能够利用MAXScript中已有的函数和循环,利用数学计算推导出阵列的位置即可构建出DNA 双螺旋结构。此外利用MAXScript代码进行DNA双螺旋结构的构建有很多的好处与优势,你能够根据自己的需要设立自己DNA分子的小球的颜色等等来更好地体现出你所要构建的模型的功能与内容。
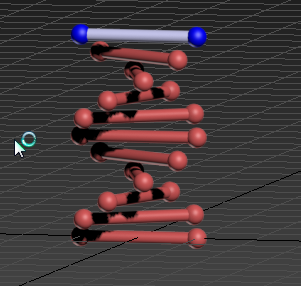
下面就是利用MAXScript代码来实现DNA双螺旋结构的构建的具体代码:
sph01 = Sphere radius:1.7 wireColor:blue pos:[-10,0,0] sph02 = Sphere radius:1.7 wireColor:blue pos:[10,0,0] cydr01 = Cylinder radius:1.0 height:20 pos:[-10,0,0] wireColor:(color 154 154 229) rotate cydr01 (eulerangles 0 90 0) for i = 1 to 10 do( copy sph01 copy sph02 copy cydr01 sph01.pos = [-10*cos(36*i),-10*sin(36*i),3.4*i] sph02.pos = [10*cos(36*i),10*sin(36*i),3.4*i] cydr01.pos = [-10*cos(36*i),-10*sin(36*i),3.4*i] j = 36 rotate cydr01 (eulerangles 0 0 j) )
运行代码后的结果图如下: