原文链接:http://tecdat.cn/?p=8287
介绍
缺失值被认为是预测建模的首要障碍。因此,掌握克服这些问题的方法很重要。
估算缺失值的方法的选择在很大程度上影响了模型的预测能力。在大多数统计分析方法中,按列表删除是用于估算缺失值的默认方法。但是,它不那么好,因为它会导致信息丢失。
您是否知道R具有用于遗漏价值估算的可靠软件包?
在本文中,我列出了5个R语言方法。
链式方程进行的多元插补
通过链式方程进行的多元插补是R用户常用的。与单个插补(例如均值)相比,创建多个插补可解决缺失值的不确定性。
MICE假定丢失数据是随机(MAR)丢失,这意味着,一个值丢失概率上观测值仅取决于并且可以使用它们来预测。通过为每个变量指定插补模型,可以按变量插补数据。
例如:假设我们有X1,X2….Xk变量。如果X1缺少值,那么它将在其他变量X2到Xk上回归。然后,将X1中的缺失值替换为获得的预测值。同样,如果X2缺少值,则X1,X3至Xk变量将在预测模型中用作自变量。稍后,缺失值将被替换为预测值。
默认情况下,线性回归用于预测连续缺失值。Logistic回归用于分类缺失值。一旦完成此循环,就会生成多个数据集。这些数据集仅在估算的缺失值上有所不同。通常,将这些数据集分别构建模型并组合其结果被认为是一个好习惯。
确切地说,此软件包使用的方法是:
- PMM(预测均值匹配)–用于数字变量
- logreg(逻辑回归)–对于二进制变量(具有2个级别)
- polyreg(贝叶斯多元回归)–用于因子变量(> = 2级)
- 比例赔率模型(有序,> = 2个级别)
现在让我们实际了解它。
我删除了分类变量。让我们在这里关注连续值。要处理分类变量,只需对级别进行编码并按照以下步骤进行即可。
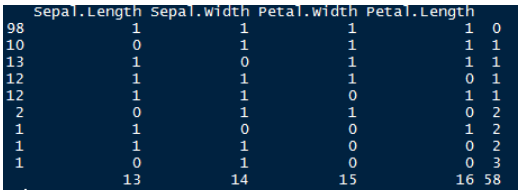
md.pattern()的功能,它返回数据集中每个变量中存在的缺失值的表格形式。
让我们了解一下这张表。有98个观测值,没有缺失值。Sepal.Length中有10个观测值缺失的观测值。同样,Sepal.Width等还有13个缺失值。
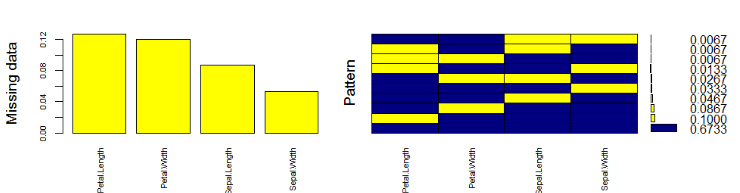
我们还可以创建代表缺失值的视觉效果。
让我们快速了解这一点。数据集中有67%的值,没有缺失值。在Petal.Length中缺少10%的值,在Petal.Width中缺少8%的值,依此类推。您还可以查看直方图,该直方图清楚地描述了变量中缺失值的影响。
现在,让我们估算缺失的值。
这是使用的参数的说明:
- m – 估算数据集
- maxit – 插补缺失值的迭代次数
- method –是指插补中使用的方法。我们使用了预测均值匹配。
由于有5个估算数据集,因此可以使用complete()函数选择任何数据集。
还可以合并来自这些模型的结果,并使用pool()命令获得合并的输出。
请注意,我仅出于演示目的使用了上面的命令。您可以在最后替换变量值并尝试。
多重插补
该程序包还执行多个插补(生成插补数据集)以处理缺失值。多重插补有助于减少偏差并提高效率。它可以通过基于引导程序的EMB算法启用,从而可以更快速,更可靠地插入许多变量,包括横截面,时间序列数据等。此外,还可以使用多核CPU的并行插入功能来启用它。
它做出以下假设:
- 数据集中的所有变量均具有多元正态分布(MVN)。它使用均值和协方差汇总数据。
- 丢失的数据本质上是随机的(随机丢失)
因此,当数据具有多变量正态分布时,此 最有效。如果没有,将进行转换以使数据接近常态。
现在让我们实际了解它。
您唯一需要注意的是对变量进行分类。
要检查数据集中的特定列,请使用以下命令
随机森林
顾名思义,missForest是一个实现随机森林算法。它适用于各种变量类型的非参数插补法。那么,什么是非参数方法?
非参数方法不会有关于函数形式明确的假设˚F 。取而代之的是,它尝试估计f,使其可以与数据点尽可能接近,而似乎并不切实际。
它是如何工作的 ?简而言之,它为每个变量建立一个随机森林模型。然后,它使用模型在观测值的帮助下预测变量中的缺失值。
它产生OOB(袋外)估算误差估计。而且,它对插补过程提供了高水平的控制。它有选择分别返回OOB(每个变量),而不是聚集在整个数据矩阵。这有助于更仔细地为每个变量如何准确的模型估算值。
NRMSE是归一化的均方误差。它用于表示从估算连续值得出的误差。PFC(错误分类的比例)用于表示从估算类别值得出的错误。
这表明类别变量的误差为6%,连续变量的误差为15%。这可以通过调整mtry和ntree参数的值来改善 。mtry是指在每个分割中随机采样的变量数。ntree是指在森林中生长的树木数量。
非参数回归方法
对多个插补中的每个插补使用不同的引导程序重采样。然后,将 加性模型(非参数回归方法)拟合到从原始数据中进行替换得到的样本上,并使用非缺失值(独立变量)预测缺失值(充当独立变量)。
然后,它使用预测均值匹配(默认)来插补缺失值。预测均值匹配非常适合连续和分类(二进制和多级),而无需计算残差和最大似然拟合。
、
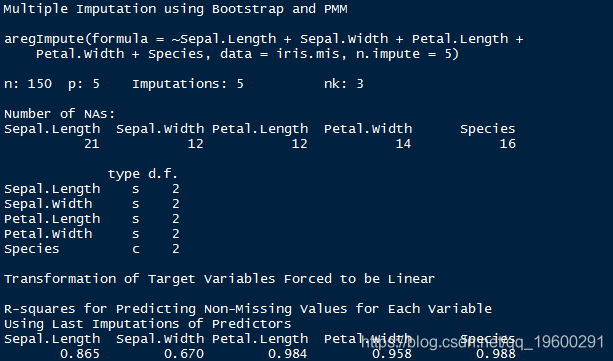
argImpute()自动识别变量类型并对其进行相应处理。
> impute_arg
输出显示R²值作为预测的缺失值。该值越高,预测的值越好。您还可以使用以下命令检查估算值
带有诊断的多重插补
带有诊断的多重插补 提供了一些用于处理缺失值的功能。 它也构建了多个插补模型来近似缺失值。并且,使用预测均值匹配方法。
虽然,我已经在上面解释了预测均值匹配(pmm) :对于变量中缺失值的每个观察值,我们都会从可用值中找到最接近的观察值该变量的预测均值。然后将来自“匹配”的观察值用作推定值。
- 它可以对插补模型进行图形诊断,并可以实现插补过程的收敛。
- 它使用贝叶斯版本的回归模型来处理分离问题。
- 插补模型规范类似于R中的回归输出
- 它会自动检测数据中的不规则性,例如变量之间的高共线性。
- 而且,它在归算过程中增加了噪声,以解决加性约束的问题。
如图所示,它使用汇总统计信息来定义估算值。
尾注
在本文中,我说明使用5个方法进行缺失值估算。这种方法可以帮助您在建立预测模型时获得更高的准确性。