









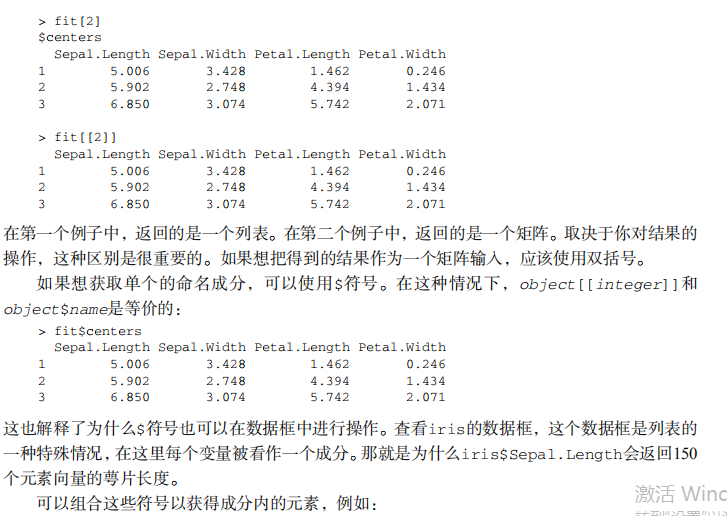





运行的条件是一元逻辑向量(TRUE或FALSE)并且不能有缺失(NA)。else部分是可选的。如果 13 仅有一个语句,花括号也是可以省略的。 下面的代码片段是一个例子: if(interactive()){ 14 plot(x, y) } else { png("myplot.png") plot(x, y) dev.off() 15 } 如果代码交互运行,interactive()函数返回TRUE,同时输出一个曲线图。否则,曲线图被存 在磁盘里。你可以使用第21章中的if()函数。 16 3. ifelse() ifelse()是函数if()的量化版本。矢量化允许一个函数来处理没有明确循环的对象。 ifelse()的格式是: 17 ifelse(test, yes, no) 其中test是已强制为逻辑模式的对象,yes返回test元素为真时的值,no返回test元素为假时 的值。 18 比如你有一个p值向量,是从包含六个统计检验的统计分析中提取出来的,并且你想要标记 p<0.05水平下的显著性检验。可以使用下面的代码: > pvalues <- c(.0867, .0018, .0054, .1572, .0183, .5386) 19 > results <- ifelse(pvalues <.05, "Significant", "Not Significant") > results [1] "Not Significant" "Significant" "Significant" [4] "Not Significant" "Significant" "Not Significant" 20 ifelse()函数通过pvalues向量循环并返回一个包括"Significant"或"Not Significant" 的字符串。返回的结果依赖于pvalues返回的值是否大于0.05。 同样的结果可以使用显式循环完成: 21 pvalues <- c(.0867, .0018, .0054, .1572, .0183, .5386) results <- vector(mode="character", length=length(pvalues)) for(i in 1:length(pvalues)){ 22 if (pvalues[i] < .05) results[i] <- "Significant" else results[i] <- "Not Significant" } 可以看出,向量化的版本更快且更有效。 23 有一些其他的控制结构,包括while()、repeat()和switch(),但是这里介绍的是最常用 的。有了数据结构和控制结构,我们就可以讨论创建函数了。
20.1.3 创建函数 在R中处处是函数。算数运算符+、-、/和*实际上也是函数。例如,2 + 2等价于 "+"(2, 2)。 本节将主要描述函数语法。语句环境将在20-2节描述。 1. 函数语法 函数的语法格式是: functionname <- function(parameters){ statements return(value) } 如果函数中有多个参数,那么参数之间用逗号隔开。 参数可以通过关键字和/或位置来传递。另外,参数可以有默认值。请看下面的函数: f <- function(x, y, z=1){ result <- x + (2*y) + (3*z) return(result) } > f(2,3,4) [1] 20 > f(2,3) [1] 11 > f(x=2, y=3) [1] 11 > f(z=4, y=2, 3) [1] 19 在第一个例子中,参数是通过位置(x=2,y=3,z=4)传递的。在第二个例子中,参数也是通过 位置传递的,并且z默认为1。在第三个例子中,参数是通过关键字传递的,z也默认为1。在最后 一个例子中,y和z是通过关键字传递的,并且x被假定为未明确指定的(这里x=3)第一个参数。 参数是可选的,但即使没有值被传递也必须使用圆括号。return()函数返回函数产生的对 象。它也是可选的;如果缺失,函数中最后一条语句的结果也会被返回。 你可以使用args()函数来观测参数的名字和默认值: > args(f) function (x, y, z = 0) NULL args()被设计用于交互式观测。如果你需要以编程方式获取参数名称和默认值,可以使用 formals()函数。它返回含有必要信息的列表。 参数是按值传递的,而不是按地址传递。请看下面这个函数语句: result <- lm(height ~ weight, data=women) women数据集不是直接得到的。需要形成一个副本然后传递给函数。如果women数据集很大的话, 内存(RAM)可能被迅速用完。这可能成为处理大数据问题时的难题能需要使用特殊的技术(见






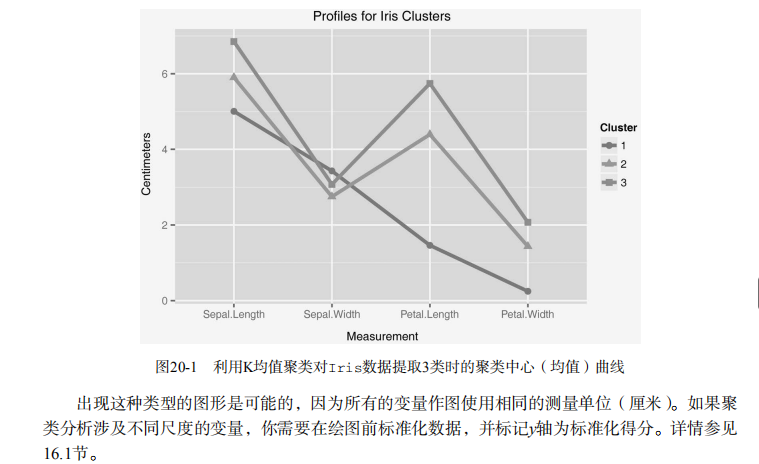
#--------------------------------------------------------------------# # R in Action (2nd ed): Chapter 20 # # Advanced R programming # # requires packages ggplot2, reshape2, foreach, doParallel # # install.packages(c("ggplot2", "reshap2e", "foreach", "doParallel"))# #--------------------------------------------------------------------# # Atomic vectors passed <- c(TRUE, TRUE, FALSE, TRUE) ages <- c(15, 18, 25, 14, 19) cmplxNums <- c(1+2i, 0+1i, 39+3i, 12+2i) names <- c("Bob", "Ted", "Carol", "Alice") # Matrices x <- c(1,2,3,4,5,6,7,8) class(x) print(x) attr(x, "dim") <- c(2,4) print(x) class(x) attributes(x) attr(x, "dimnames") <- list(c("A1", "A2"), c("B1", "B2", "B3", "B4")) print(x) attr(x, "dim") <- NULL class(x) print(x) # Generic vectors (lists) head(iris) unclass(iris) attributes(iris) set.seed(1234) fit <- kmeans(iris[1:4], 3) names(fit) unclass(fit) sapply(fit, class) # Indexing atomic vectors x <- c(20, 30, 40) x[3] x[c(2,3)] x <- c(A=20, B=30, C=40) x[c(2,3)] x[c("B", "C")] # Indexing lists fit[c(2,7)] fit[2] fit[[2]] fit$centers fit[[2]][1,] fit$centers$Petal.Width # should give an error # Listing 20.1 - Plotting the centroides from a k-mean cluster analysis fit <- kmeans(iris[1:4], 3) means <- fit$centers library(reshape2) dfm <- melt(means) names(dfm) <- c("Cluster", "Measurement", "Centimeters") dfm$Cluster <- factor(dfm$Cluster) head(dfm) library(ggplot2) ggplot(data=dfm, aes(x=Measurement, y=Centimeters, group=Cluster)) + geom_point(size=3, aes(shape=Cluster, color=Cluster)) + geom_line(size=1, aes(color=Cluster)) + ggtitle("Profiles for Iris Clusters") # for loops for(i in 1:5) print(1:i) for(i in 5:1)print(1:i) # ifelse pvalues <- c(.0867, .0018, .0054, .1572, .0183, .5386) results <- ifelse(pvalues <.05, "Significant", "Not Significant") results pvalues <- c(.0867, .0018, .0054, .1572, .0183, .5386) results <- vector(mode="character", length=length(pvalues)) for(i in 1:length(pvalues)){ if (pvalues[i] < .05) results[i] <- "Significant" else results[i] <- "Not Significant" } results # Creating functions f <- function(x, y, z=1){ result <- x + (2*y) + (3*z) return(result) } f(2,3,4) f(2,3) f(x=2, y=3) f(z=4, y=2, 3) args(f) # object scope x <- 2 y <- 3 z <- 4 f <- function(w){ z <- 2 x <- w*y*z return(x) } f(x) x y z # Working with environments x <- 5 myenv <- new.env() assign("x", "Homer", env=myenv) ls() ls(myenv) x get("x", env=myenv) myenv <- new.env() myenv$x <- "Homer" myenv$x parent.env(myenv) # function closures trim <- function(p){ trimit <- function(x){ n <- length(x) lo <- floor(n*p) + 1 hi <- n + 1 - lo x <- sort.int(x, partial = unique(c(lo, hi)))[lo:hi] } trimit } x <- 1:10 trim10pct <- trim(.1) y <- trim10pct(x) y trim20pct <- trim(.2) y <- trim20pct(x) y ls(environment(trim10pct)) get("p", env=environment(trim10pct)) makeFunction <- function(k){ f <- function(x){ print(x + k) } } g <- makeFunction(10) g (4) k <- 2 g (5) ls(environment(g)) environment(g)$k # Generic functions summary(women) fit <- lm(weight ~ height, data=women) summary(fit) class(women) class(fit) methods(summary) # Listing 20.2 - An example of a generic function mymethod <- function(x, ...) UseMethod("mymethod") mymethod.a <- function(x) print("Using A") mymethod.b <- function(x) print("Using B") mymethod.default <- function(x) print("Using Default") x <- 1:5 y <- 6:10 z <- 10:15 class(x) <- "a" class(y) <- "b" mymethod(x) mymethod(y) mymethod(z) class(z) <- c("a", "b") mymethod(z) class(z) <- c("c", "a", "b") mymethod(z) # Vectorization and efficient code set.seed(1234) mymatrix <- matrix(rnorm(10000000), ncol=10) accum <- function(x){ sums <- numeric(ncol(x)) for (i in 1:ncol(x)){ for(j in 1:nrow(x)){ sums[i] <- sums[i] + x[j,i] } } } system.time(accum(mymatrix)) # using loops system.time(colSums(mymatrix)) # using vectorization # Correctly size objects set.seed(1234) k <- 100000 x <- rnorm(k) y <- 0 system.time(for (i in 1:length(x)) y[i] <- x[i]^2) y <- numeric(k) system.time(for (i in 1:k) y[i] <- x[i]^2) y <- numeric(k) system.time(y <- x^2) # Listing 20.3 - Parallelization with foreach and doParallel library(foreach) library(doParallel) registerDoParallel(cores=4) eig <- function(n, p){ x <- matrix(rnorm(100000), ncol=100) r <- cor(x) eigen(r)$values } n <- 1000000 p <- 100 k <- 500 system.time( x <- foreach(i=1:k, .combine=rbind) %do% eig(n, p) ) system.time( x <- foreach(i=1:k, .combine=rbind) %dopar% eig(n, p) ) # Finding common errors mtcars$Transmission <- factor(mtcars$a, levels=c(1,2), labels=c("Automatic", "Manual")) aov(mpg ~ Transmission, data=mtcars) # generates error head(mtcars[c("mpg", "Transmission")]) table(mtcars$Transmission) # here is the source of the error # Listing 20.4 - A sample debugging session args(mad) debug(mad) mad(1:10) # enters debugging mode # Q to quit - see text undebug(mad) # Listing 20.5 - Sample debugging session with recover() f <- function(x, y){ z <- x + y g(z) } g <- function(x){ z <- round(x) h(z) } h <- function(x){ set.seed(1234) z <- rnorm(x) print(z) } options(error=recover) f(2,3) f(2, -3) # enters debugging mode at this point