python机器学习-乳腺癌细胞挖掘(博主亲自录制视频)https://study.163.com/course/introduction.htm?courseId=1005269003&utm_campaign=commission&utm_source=cp-400000000398149&utm_medium=share
Python自然语言处理(三) -- 利用NLTK进行聚类
这篇文章介绍如何利用NLTK进行聚类,和上两篇文章Python自然语言处理(一)、Python自然语言处理(二)不同,聚类不能算作自然语言处理的内容,但可以很容易应用到NLP中,因此将其划分到自然语言处理下。
-------------------进入正题--------------------
nltk内部封装了常用的聚类方法:Kmeans(K均值)、混合高斯聚类以及GAAC(组平均层次聚类)
使用这些聚类方法之前,需要保证已安装pynum,因为他们聚类传入的数据要求是pynum.array类型。
pynum的安装教程网上一大堆,这里不进行介绍。
一、数据准备
下面所有的聚类数据都是用这组:
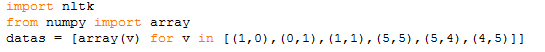
注意:datas的元素是array类型
二、Kmeans聚类
用到的函数:
nltk.cluster.kmeans.KMeansClusterer(num_means, distance )#返回Kmeans聚类器的对象,num_means:目标类别数,distance:自定义距离函数

例子中,我们的目标类别数目为2, 距离函数为欧式距离
km.cluster(datas) #对数据集datas进行聚类
km.classify(data)#返回data被分到的类别
三、GAAC聚类
nltk.cluster.gaac.GAAClusterer(num_clusters , normalise )#num_clusters:目标类别数, normalise:是否归一化
这里需要说明一下,GAAC的距离使用的是点积的结果,并不是余弦相似度,如果normalise设置为True,将相似度进行归一化,此时的距离为余弦相似度
GAAC无法自定义相似度

例子中,我们设置目标类别为3,距离需要归一化,也即相似度为余弦相似度。
上述结果可以看出,所分的三个类为{(1,0)}, {(0,1)}, {[ 1, 1.] [ 5, 5.] [ 5 , 4.] [ 4 , 5.]}
结果和Kmeans非常不同,这是因为使用的相似度为余弦相似度
四、混合高斯聚类
用到的函数:
nltk.cluster.em.EMClusterer(initial_means)#initial_means:the means of the gaussian cluster centers


箭头表示聚类的结果,其余数据为中间数据。