python机器学习-乳腺癌细胞挖掘(博主亲自录制视频)
https://study.163.com/course/introduction.htm?courseId=1005269003&utm_campaign=commission&utm_source=cp-400000000398149&utm_medium=share
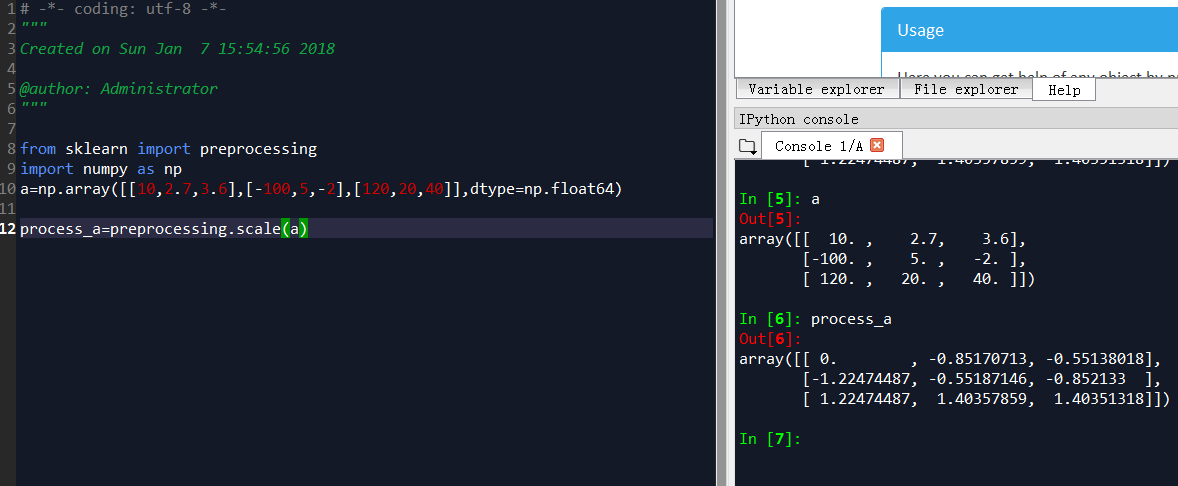
from sklearn import preprocessing import numpy as np a=np.array([[10,2.7,3.6],[-100,5,-2],[120,20,40]],dtype=np.float64) process_a=preprocessing.scale(a)
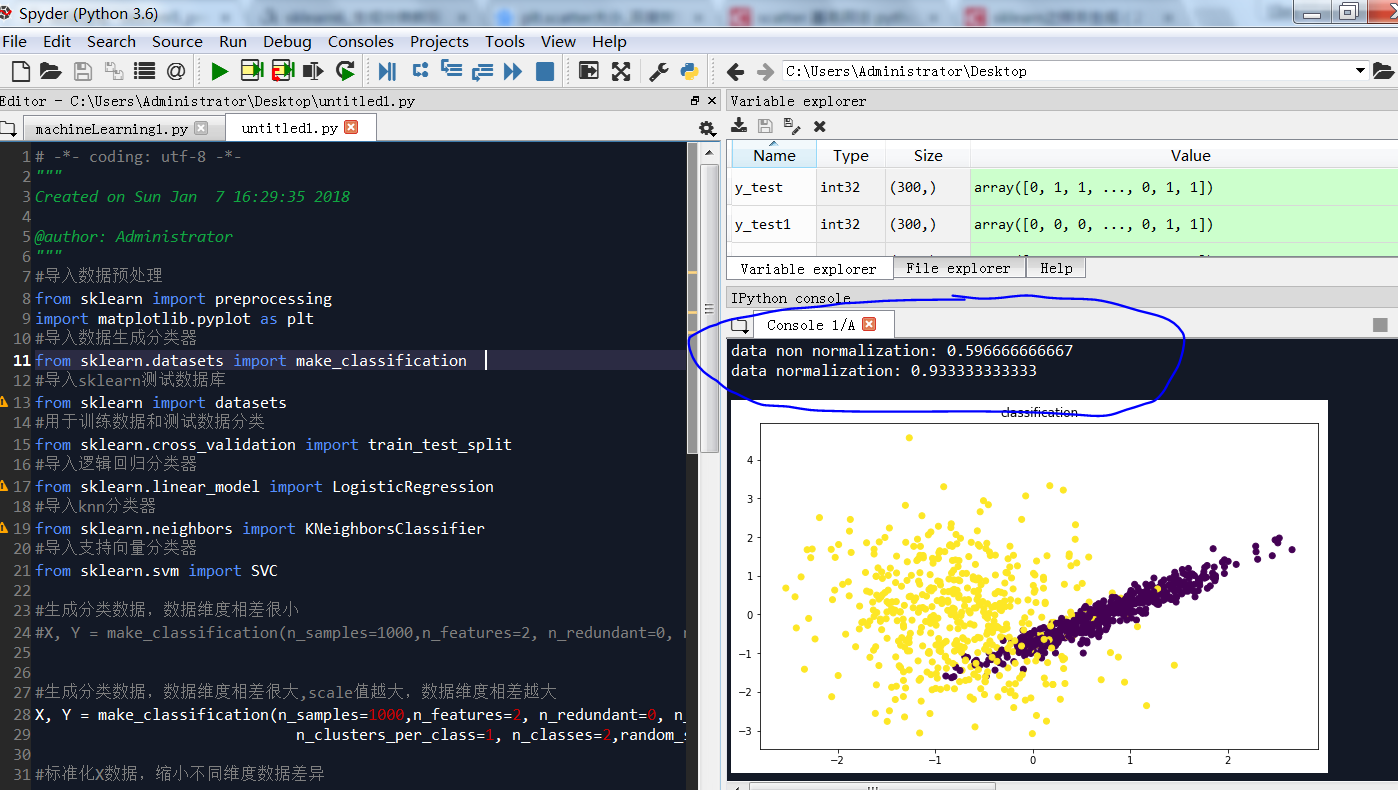
比较数据标准化前后准确性差别
# -*- coding: utf-8 -*-
"""
Created on Sun Jan 7 16:29:35 2018
@author: Administrator
"""
#导入数据预处理
from sklearn import preprocessing
import matplotlib.pyplot as plt
#导入数据生成分类器
from sklearn.datasets import make_classification
#导入sklearn测试数据库
from sklearn import datasets
#用于训练数据和测试数据分类
from sklearn.cross_validation import train_test_split
#导入逻辑回归分类器
from sklearn.linear_model import LogisticRegression
#导入knn分类器
from sklearn.neighbors import KNeighborsClassifier
#导入支持向量分类器
from sklearn.svm import SVC
#生成分类数据,数据维度相差很小
#X, Y = make_classification(n_samples=1000,n_features=2, n_redundant=0, n_informative=2,n_clusters_per_class=1, n_classes=2)
#生成分类数据,数据维度相差很大,scale值越大,数据维度相差越大
X, Y = make_classification(n_samples=1000,n_features=2, n_redundant=0, n_informative=2,
n_clusters_per_class=1, n_classes=2,random_state=22,scale=100)
#标准化X数据,缩小不同维度数据差异
X1=preprocessing.scale(X)
#数据未标准化:分成训练数据和测试数据
X_train, X_test, y_train, y_test = train_test_split(
X, Y, test_size=0.3)
#X数据标准化后:分成训练数据和测试数据
X_train1, X_test1, y_train1, y_test1 = train_test_split(
X1, Y, test_size=0.3)
#画布的大小为长20cm高20cm
plt.figure(figsize=(10,6))
#标题,fontsize为标题字体大小
plt.title("classification", fontsize='large')
#绘制点,X1[:, 0]为点的x列表值, X1[:, 1]为点的y列表值, c=Y1表示颜色,c为color缩写
plt.scatter(X1[:, 0], X1[:, 1], marker='o', c=Y)
#创建两个支持向量模型
model=SVC()
model_normal=SVC()
#未标准化的训练模型
model.fit(X_train,y_train)
#标准化的训练模型
model_normal.fit(X_train1,y_train1)
#测试未标准化得分
print("data non normalization:",model.score(X_test,y_test))
#测试标准化得分
print("data normalization:",model_normal.score(X_test1,y_test1))
预处理数
1. 标准化:去均值,方差规模化
Standardization标准化:将特征数据的分布调整成标准正太分布,也叫高斯分布,也就是使得数据的均值维0,方差为1.
标准化的原因在于如果有些特征的方差过大,则会主导目标函数从而使参数估计器无法正确地去学习其他特征。
标准化的过程为两步:去均值的中心化(均值变为0);方差的规模化(方差变为1)。
在sklearn.preprocessing中提供了一个scale的方法,可以实现以上功能。
from sklearn import preprocessing
import numpy as np
# 创建一组特征数据,每一行表示一个样本,每一列表示一个特征
x = np.array([[1., -1., 2.],
[2., 0., 0.],
[0., 1., -1.]])
# 将每一列特征标准化为标准正太分布,注意,标准化是针对每一列而言的
x_scale = preprocessing.scale(x)
x_scalearray([[ 0. , -1.22474487, 1.33630621],
[ 1.22474487, 0. , -0.26726124],
[-1.22474487, 1.22474487, -1.06904497]])
# 可以查看标准化后的数据的均值与方差,已经变成0,1了
x_scale.mean(axis=0)array([ 0., 0., 0.])
# axis=1表示对每一行去做这个操作,axis=0表示对每一列做相同的这个操作x_scale.mean(axis=1)array([ 0.03718711, 0.31916121, -0.35634832])# 同理,看一下标准差
x_scale.std(axis=0)array([ 1., 1., 1.])preprocessing这个模块还提供了一个实用类StandarScaler,它可以在训练数据集上做了标准转换操作之后,把相同的转换应用到测试训练集中。
这是相当好的一个功能。可以对训练数据,测试数据应用相同的转换,以后有新的数据进来也可以直接调用,不用再重新把数据放在一起再计算一次了。
# 调用fit方法,根据已有的训练数据创建一个标准化的转换器
scaler = preprocessing.StandardScaler().fit(x)
scalerStandardScaler(copy=True, with_mean=True, with_std=True)# 使用上面这个转换器去转换训练数据x,调用transform方法
scaler.transform(x)array([[ 0. , -1.22474487, 1.33630621],
[ 1.22474487, 0. , -0.26726124],
[-1.22474487, 1.22474487, -1.06904497]])# 好了,比如现在又来了一组新的样本,也想得到相同的转换
new_x = [[-1., 1., 0.]]
scaler.transform(new_x)array([[-2.44948974, 1.22474487, -0.26726124]])
恩,完美。
另外,StandardScaler()中可以传入两个参数:with_mean,with_std.这两个都是布尔型的参数,默认情况下都是true,但也可以自定义成false.即不要均值中心化或者不要方差规模化为1.
1.1 规模化特征到一定的范围内
也就是使得特征的分布是在一个给定最小值和最大值的范围内的。一般情况下是在[0,1]之间,或者是特征中绝对值最大的那个数为1,其他数以此维标准分布在[[-1,1]之间
以上两者分别可以通过MinMaxScaler 或者 MaxAbsScaler方法来实现。
之所以需要将特征规模化到一定的[0,1]范围内,是为了对付那些标准差相当小的特征并且保留下稀疏数据中的0值。
MinMaxScaler
在MinMaxScaler中是给定了一个明确的最大值与最小值。它的计算公式如下:
X_std = (X - X.min(axis=0)) / (X.max(axis=0) - X.min(axis=0))
X_scaled = X_std / (max - min) + min
以下这个例子是将数据规与[0,1]之间,每个特征中的最小值变成了0,最大值变成了1,请看:
min_max_scaler = preprocessing.MinMaxScaler()
x_minmax = min_max_scaler.fit_transform(x)
x_minmaxarray([[ 0.5 , 0. , 1. ],
[ 1. , 0.5 , 0.33333333],
[ 0. , 1. , 0. ]])同样的,如果有新的测试数据进来,也想做同样的转换咋办呢?请看:
x_test = np.array([[-3., -1., 4.]])
x_test_minmax = min_max_scaler.transform(x_test)
x_test_minmaxarray([[-1.5 , 0. , 1.66666667]])MaxAbsScaler
原理与上面的很像,只是数据会被规模化到[-1,1]之间。也就是特征中,所有数据都会除以最大值。这个方法对那些已经中心化均值维0或者稀疏的数据有意义。
来个小例子感受一下:
max_abs_scaler = preprocessing.MaxAbsScaler()
x_train_maxsbs = max_abs_scaler.fit_transform(x)
x_train_maxsbsarray([[ 0.5, -1. , 1. ],
[ 1. , 0. , 0. ],
[ 0. , 1. , -0.5]])
# 同理,也可以对新的数据集进行同样的转换
x_test = np.array([[-3., -1., 4.]])
x_test_maxabs = max_abs_scaler.transform(x_test)
x_test_maxabsarray([[-1.5, -1. , 2. ]])
1.2 规模化稀疏数据
如果对稀疏数据进行去均值的中心化就会破坏稀疏的数据结构。虽然如此,我们也可以找到方法去对稀疏的输入数据进行转换,特别是那些特征之间的数据规模不一样的数据。
MaxAbsScaler 和 maxabs_scale这两个方法是专门为稀疏数据的规模化所设计的。
1.3 规模化有异常值的数据
如果你的数据有许多异常值,那么使用数据的均值与方差去做标准化就不行了。
在这里,你可以使用robust_scale 和 RobustScaler这两个方法。它会根据中位数或者四分位数去中心化数据。
2 正则化Normalization
正则化是将样本在向量空间模型上的一个转换,经常被使用在分类与聚类中。
函数normalize 提供了一个快速有简单的方式在一个单向量上来实现这正则化的功能。正则化有l1,l2等,这些都可以用上:
x_normalized = preprocessing.normalize(x, norm='l2')
print x
print x_normalized[[ 1. -1. 2.]
[ 2. 0. 0.]
[ 0. 1. -1.]]
[[ 0.40824829 -0.40824829 0.81649658]
[ 1. 0. 0. ]
[ 0. 0.70710678 -0.70710678]]
preprocessing这个模块还提供了一个实用类Normalizer,实用transform方法同样也可以对新的数据进行同样的转换
# 根据训练数据创建一个正则器
normalizer = preprocessing.Normalizer().fit(x)
normalizerNormalizer(copy=True, norm='l2')# 对训练数据进行正则
normalizer.transform(x)array([[ 0.40824829, -0.40824829, 0.81649658],
[ 1. , 0. , 0. ],
[ 0. , 0.70710678, -0.70710678]])
# 对新的测试数据进行正则
normalizer.transform([[-1., 1., 0.]])array([[-0.70710678, 0.70710678, 0. ]])
normalize和Normalizer都既可以用在密集数组也可以用在稀疏矩阵(scipy.sparse)中
对于稀疏的输入数据,它会被转变成维亚索的稀疏行表征(具体请见scipy.sparse.csr_matrix)
3 二值化–特征的二值化
特征的二值化是指将数值型的特征数据转换成布尔类型的值。可以使用实用类Binarizer。
from sklearn import preprocessing
import numpy as np
# 创建一组特征数据,每一行表示一个样本,每一列表示一个特征
x = np.array([[1., -1., 2.],
[2., 0., 0.],
[0., 1., -1.]])
binarizer = preprocessing.Binarizer().fit(x)
binarizer.transform(x)array([[ 1., 0., 1.],
[ 1., 0., 0.],
[ 0., 1., 0.]])
默认是根据0来二值化,大于0的都标记为1,小于等于0的都标记为0。
当然也可以自己设置这个阀值,只需传出参数threshold即可。
binarizer = preprocessing.Binarizer(threshold=1.5)
binarizer.transform(x)array([[ 0., 0., 1.],
[ 1., 0., 0.],
[ 0., 0., 0.]])
binarize and Binarizer都可以用在密集向量和稀疏矩阵上。
4 为类别特征编码
我们知道特征可能是连续型的也可能是类别型的变量,比如说:
[“male”, “female”], [“from Europe”, “from US”, “from Asia”], [“uses Firefox”, “uses Chrome”, “uses Safari”, “uses Internet Explorer”].
这些类别特征无法直接进入模型,它们需要被转换成整数来表征,比如:
[“male”, “from US”, “uses Internet Explorer”] could be expressed as [0, 1, 3] while [“female”, “from Asia”, “uses Chrome”] would be [1, 2, 1].
然而上面这种表征的方式仍然不能直接为scikit-learn的模型所用,因为模型会把它们当成序列型的连续变量。
要想使得类别型的变量能最终被模型直接使用,可以使用one-of-k编码或者one-hot编码。这些都可以通过OneHotEncoder实现,它可以将有n种值的一个特征变成n个二元的特征。
enc = preprocessing.OneHotEncoder()
enc.fit([[0, 0, 3], [1, 1, 0], [0, 2, 1], [1, 0, 2]])
enc.transform([[0,1,3]]).toarray()- 1
- 2
- 3
array([[ 1., 0., 0., 1., 0., 0., 0., 0., 1.]])
- 1
- 2
特征1中有(0,1)两个值,特征2中有(0,1,2)3个值,特征3中有(0,1,2,3)4个值,所以编码之后总共有9个二元特征。
但是呢,也会存在这样的情况,某些特征中可能对一些值有缺失,比如明明有男女两个性别,样本数据中都是男性,这样就会默认被判别为我只有一类值。这个时候我们可以向OneHotEncoder传如参数n_values,用来指明每个特征中的值的总个数。如下:
enc = preprocessing.OneHotEncoder(n_values=[2,3,4])
enc.fit([[1, 2, 3], [0, 2, 0]])
enc.transform([[1,0,0]]).toarray()- 1
- 2
- 3
array([[ 0., 1., 1., 0., 0., 1., 0., 0., 0.]])
- 1
- 2
5 弥补缺失数据
在scikit-learn的模型中都是假设输入的数据是数值型的,并且都是有意义的,如果有缺失数据是通过NAN,或者空值表示的话,就无法识别与计算了。
要弥补缺失值,可以使用均值,中位数,众数等等。Imputer这个类可以实现。请看:
import numpy as np
from sklearn.preprocessing import Imputer
imp = Imputer(missing_values='NaN', strategy='mean', axis=0)
imp.fit([[1, 2], [np.nan, 3], [7, 6]])
x = [[np.nan, 2], [6, np.nan], [7, 6]]
imp.transform(x)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
array([[ 4. , 2. ],
[ 6. , 3.66666667],
[ 7. , 6. ]])
- 1
- 2
- 3
- 4
Imputer类同样也可以支持稀疏矩阵,以下例子将0作为了缺失值,为其补上均值
import scipy.sparse as sp
# 创建一个稀疏矩阵
x = sp.csc_matrix([[1, 2], [0, 3], [7, 6]])
imp = Imputer(missing_values=0, strategy='mean', verbose=0)
imp.fit(x)
x_test = sp.csc_matrix([[0, 2], [6, 0], [7, 6]])
imp.transform(x_test)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
array([[ 4. , 2. ],
[ 6. , 3.66666667],
[ 7. , 6. ]])
- 1
- 2
- 3
- 4
6 创建多项式特征
有的时候线性的特征并不能做出美的模型,于是我们会去尝试非线性。非线性是建立在将特征进行多项式地展开上的。
比如将两个特征 (X_1, X_2),它的平方展开式便转换成5个特征(1, X_1, X_2, X_1^2, X_1X_2, X_2^2). 代码案例如下:
import numpy as np
from sklearn.preprocessing import PolynomialFeatures
# 自建一组3*2的样本
x = np.arange(6).reshape(3, 2)
# 创建2次方的多项式
poly = PolynomialFeatures(2)
poly.fit_transform(x)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
array([[ 1., 0., 1., 0., 0., 1.],
[ 1., 2., 3., 4., 6., 9.],
[ 1., 4., 5., 16., 20., 25.]])
- 1
- 2
- 3
- 4
看,变成了3*6的特征矩阵,里面有5个特征,加上第一列的是Bias.
也可以自定义选择只要保留特征相乘的项。
即将 (X_1, X_2, X_3) 转换成 (1, X_1, X_2, X_3, X_1X_2, X_1X_3, X_2X_3, X_1X_2X_3).
x = np.arange(9).reshape(3, 3)
poly = PolynomialFeatures(degree=3, interaction_only=True)
poly.fit_transform(x)- 1
- 2
- 3
- 4
array([[ 1., 0., 1., 2., 0., 0., 2., 0.],
[ 1., 3., 4., 5., 12., 15., 20., 60.],
[ 1., 6., 7., 8., 42., 48., 56., 336.]])
- 1
- 2
- 3
- 4
7 自定义特征的转换函数
通俗的讲,就是把原始的特征放进一个函数中做转换,这个函数出来的值作为新的特征。
比如说将特征数据做log转换,做倒数转换等等。
FunctionTransformer 可以实现这个功能
import numpy as np
from sklearn.preprocessing import FunctionTransformer
transformer = FunctionTransformer(np.log1p)
x = np.array([[0, 1], [2, 3]])
transformer.transform(x)- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
array([[ 0. , 0.69314718],
[ 1.09861229, 1.38629436]])
- 1
- 2
- 3
将上面讲到的7个预处理的方法综合起来。
当我们拿到一批原始的数据
- 首先要明确有多少特征,哪些是连续的,哪些是类别的。
- 检查有没有缺失值,对确实的特征选择恰当方式进行弥补,使数据完整。
- 对连续的数值型特征进行标准化,使得均值为0,方差为1。
- 对类别型的特征进行one-hot编码。
- 将需要转换成类别型数据的连续型数据进行二值化。
- 为防止过拟合或者其他原因,选择是否要将数据进行正则化。
- 在对数据进行初探之后发现效果不佳,可以尝试使用多项式方法,寻找非线性的关系。
- 根据实际问题分析是否需要对特征进行相应的函数转换。
恩,准备好美美的数据将为我们寻找美美的模型如虎添翼。
官方文档链接http://scikit-learn.org/stable/modules/preprocessing.html#preprocessing
python风控建模实战lendingClub(博主录制,catboost,lightgbm建模,2K超清分辨率)
https://study.163.com/course/courseMain.htm?courseId=1005988013&share=2&shareId=400000000398149
微信扫二维码,免费学习更多python资源