转载 https://blog.csdn.net/dark_scope/article/details/53229427#
维度打击,机器学习中的降维算法:ISOMAP & MDS
降维是机器学习中很有意思的一部分,很多时候它是无监督的,能够更好地刻画数据,对模型效果提升也有帮助,同时在数据可视化中也有着举足轻重的作用。
一说到降维,大家第一反应总是PCA,基本上每一本讲机器学习的书都会提到PCA,而除此之外其实还有很多很有意思的降维算法,其中就包括isomap,以及isomap中用到的MDS。
ISOMAP是‘流形学习’中的一个经典算法,流形学习贡献了很多降维算法,其中一些与很多机器学习算法也有结合,但上学的时候还看了蛮多的机器学习的书,从来没听说过流形学习的概念,还是在最新的周志华版的《机器学习》里才看到,很有意思,记录分享一下。
流形学习
流形学习应该算是个大课题了,它的基本思想就是在高维空间中发现低维结构。比如这个图: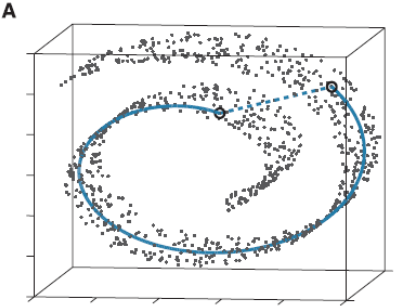
这些点都处于一个三维空间里,但我们人一看就知道它像一块卷起来的布,图中圈出来的两个点更合理的距离是A中蓝色实线标注的距离,而不是两个点之间的欧式距离(A中蓝色虚线)。
此时如果你要用PCA降维的话,它根本无法发现这样卷曲的结构(因为PCA是典型的线性降维,而图示的结构显然是非线性的),最后的降维结果就会一团乱麻,没法很好的反映点之间的关系。而流形学习在这样的场景就会有很好的效果。
我对流形学习本身也不太熟悉,还是直接说算法吧。
ISOMAP
在降维算法中,一种方式是提供点的坐标进行降维,如PCA;另一种方式是提供点之间的距离矩阵,ISOMAP中用到的MDS(Multidimensional Scaling)就是这样。
在计算距离的时候,最简单的方式自然是计算坐标之间的欧氏距离,但ISOMAP对此进行了改进,就像上面图示一样:
1.通过kNN(k-Nearest Neighbor)找到点的k个最近邻,将它们连接起来构造一张图。
2.通过计算同中各点之间的最短路径,作为点之间的距离dijdij放入距离矩阵DD
3.将DD传给经典的MDS算法,得到降维后的结果。
ISOMAP本身的核心就在构造点之间的距离,初看时不由得为其拍案叫绝,类似的思想在很多降维算法中都能看到,比如能将超高维数据进行降维可视化的t-SNE。
ISOMAP效果,可以看到选取的最短路径比较好地还原了期望的蓝色实线,用这个数据进行降维会使流形得以保持: 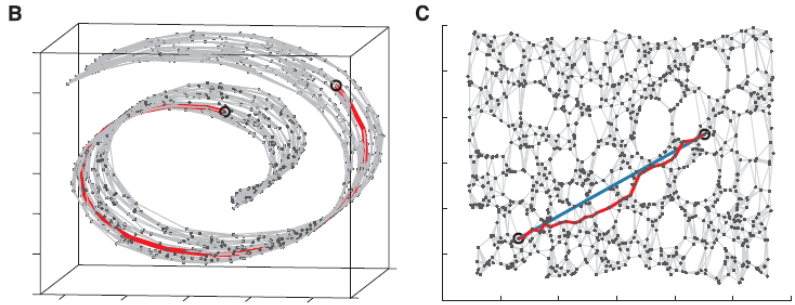
ISOMAP算法步骤可谓清晰明了,所以本文主要着重讲它中间用到的MDS算法,也是很有意思的。
经典MDS(Multidimensional Scaling)
如上文所述,MDS接收的输入是一个距离矩阵DD,我们把一些点画在坐标系里: 
如果只告诉一个人这些点之间的距离(假设是欧氏距离),他会丢失那些信息呢?
a.我们对点做平移,点之间的距离是不变的。
b.我们对点做旋转、翻转,点之间的距离是不变的。
所以想要从DD还原到原始数据XX是不可能的,因为只给了距离信息之后本身就丢掉了很多东西,不过不必担心,即使这样我们也可以对数据进行降维。
我们不妨假设:XX是一个n×qn×q的矩阵,n为样本数,q是原始的维度
计算一个很重要的矩阵BB:
可以看到我们通过MM对XX做正交变换并不会影响BB的值,而正交变换刚好就是对数据做旋转、翻转操作的。
所以如果我们想通过BB反算出XX,肯定是没法得到真正的XX,而是它的任意一种正交变换后的结果。
B中每个元素的值为:
计算距离矩阵DD,其中每个元素值为:
ag{dij_square}label{dij_square}
这时候我们有的只有DD,如果能通过DD计算出BB,再由BB计算出XX,不就达到效果了吗。
所以思路是:从D->B->X
此时我们要对X加一些限制,前面说过我们平移所有点是不会对距离矩阵造成影响的,所以我们就把数据的中心点平移到原点,对X做如下限制(去中心化):
所以有
类似的
可以看到即BB的任意行(row)之和以及任意列(column)之和都为0了。
设T为BB的trace,则有:
得到B:根据公式 (???)(???)我们有:
而(根据前面算∑ni=1d2ij∑i=1ndij2,∑nj=1d2ij∑j=1ndij2和∑ni=1∑nj=1d2ij∑i=1n∑j=1ndij2的公式可以得到)
所以
可以看到d2i⋅di⋅2 是D2D2行均值;d2⋅jd⋅j2是列均值;d2⋅⋅d⋅⋅2 是矩阵的均值。
这样我们就可以通过矩阵DD得到矩阵BB了
因为B是对称的矩阵,所以可以通过特征分解得到:
在最开始我们其实做了一个假设,即DD是由一个n×qn×q的数据XX生成的,如果事实是这样的,DD会是一个对称实矩阵,此时得到的BB刚好会有qq个非0的特征值,也就是说BB的秩等于qq,如果我们想还原XX,就选择前qq个特征值和特征向量;如果想要达到降维的目的,就选择制定的pp个(p<qp<q)。
此时我们选择前pp个特征值和特征向量,(这一步和PCA里面很类似):
所以有(ΛΛ是特征值组成的对角矩阵):
因此
如果选择p=qp=q的话,此时得到的X∗X∗就是原数据去中心化并做了某种正交变换后的值了。
MDS的例子
举个例子:拿美国一些大城市之间的距离作为矩阵传进去,简单写一写代码:
import numpy as np
import matplotlib.pyplot as plt
def mds(D,q):
D = np.asarray(D)
DSquare = D**2
totalMean = np.mean(DSquare)
columnMean = np.mean(DSquare, axis = 0)
rowMean = np.mean(DSquare, axis = 1)
B = np.zeros(DSquare.shape)
for i in range(B.shape[0]):
for j in range(B.shape[1]):
B[i][j] = -0.5*(DSquare[i][j] - rowMean[i] - columnMean[j]+totalMean)
eigVal,eigVec = np.linalg.eig(B)
X = np.dot(eigVec[:,:q],np.sqrt(np.diag(eigVal[:q])))
return X
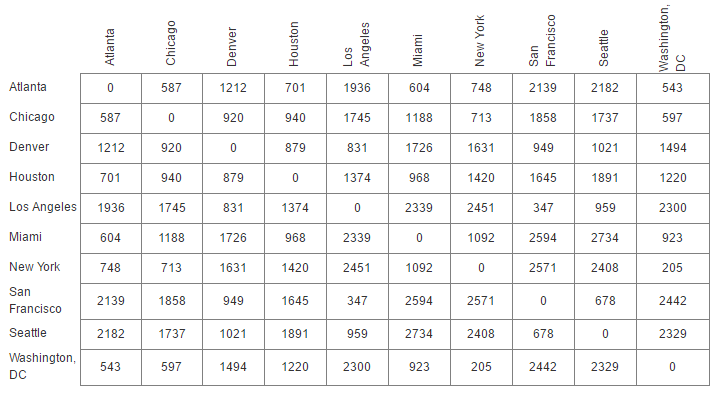
D = [[0,587,1212,701,1936,604,748,2139,2182,543],
[587,0,920,940,1745,1188,713,1858,1737,597],
[1212,920,0,879,831,1726,1631,949,1021,1494],
[701,940,879,0,1374,968,1420,1645,1891,1220],
[1936,1745,831,1374,0,2339,2451,347,959,2300],
[604,1188,1726,968,2339,0,1092,2594,2734,923],
[748,713,1631,1420,2451,1092,0,2571,2408,205],
[2139,1858,949,1645,347,2594,2571,0,678,2442],
[2182,1737,1021,1891,959,2734,2408,678,0,2329],
[543,597,1494,1220,2300,923,205,2442,2329,0]]
label = ['Atlanta','Chicago','Denver','Houston','Los Angeles','Miami','New York','San Francisco','Seattle','Washington, DC']
X = mds(D,2)
plt.plot(X[:,0],X[:,1],'o')
for i in range(X.shape[0]):
plt.text(X[i,0]+25,X[i,1]-15,label[i])
plt.show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
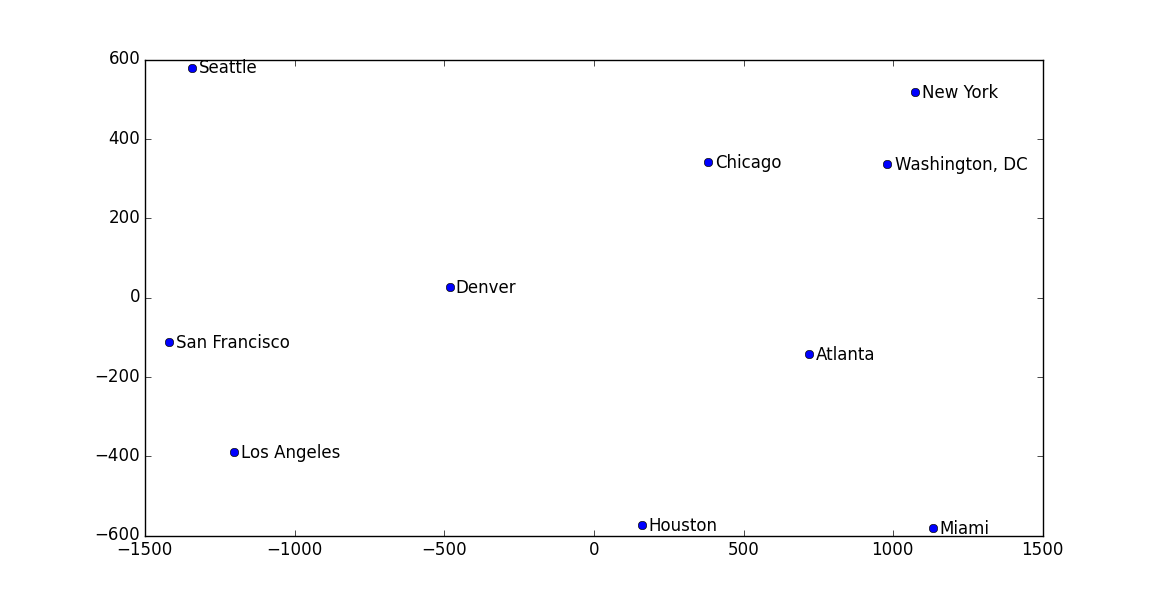
最后画出来的图中,各个城市的位置和真实世界中的相对位置都差不多:
注意,这个例子中其实也有‘流形’在里面,因为我们的地球其实是一个三维,而城市间距离刻画的是在球面上的距离,所以最后如果你去看求出来的特征值,并不像前面说的那样只有q个非0的值。
reference
- 一个nthu的课程,除了pdf还有视频,本文绝大多数关于MDS的内容都是从这里整理的:http://101.96.10.65/www.stat.nthu.edu.tw/~swcheng/Teaching/stat5191/lecture/06_MDS.pdf
- 一个MDS的例子,用于数据可视化,例子的数据来源于这里。http://www.benfrederickson.com/multidimensional-scaling/
- 周志华《机器学习》