密度聚类
密度聚类方法的指导思想是,只要一个区域中的点的密度大于某个阈值,就把它加到与之相近的聚类中去。这类算法优点在于可发现任意形状的聚类,且对噪声数据不敏感。但计算密度单元的计算复杂度大,需要建立空间索引来降低计算量。
这个方法的指导思想就是,只要一个区域中的点的密度大过某个阈值,就把它加到与之相近的聚类中去。
一.DBSCAN算法:
它将簇定义为a密度相连的点的最大集合,所有的点被分为核心点,(密度)可达点及局外点.
核心点:
如果一个点 p 在距离 ε 范围内有至少 minPts 个点(包括自己),则这个点被称为核心点,那些 ε 范围内的则被称为由 p 直接可达的,根据此定义,没有任何点是由非核心点直接可达的。
(密度)可达点:
如果存在一条道路 p1, ..., pn ,有 p1 = p和pn = q, 且每个 pi+1 都是由 pi 直接可达的(道路上除了 q 以外所有点都一定是核心点),则称 q 是由 p 可达的。
局外点:
所有不由任何点可达的点都被称为局外点。
如果 p 是核心点,则它与所有由它可达的点(包括核心点和非核心点)形成一个聚类,每个聚类拥有最少一个核心点,非核心点也可以是聚类的一部分,但它是在聚类的“边缘”位置,因为它不能达至更多的点。

在上图中,minPts = 4,点 A 和其他红色点是核心点,因为它们的 ε-邻域(图中红色圆圈)里包含最少 4 个点(包括自己),由于它们之间相互相可达,它们形成了一个聚类。点 B 和点 C 不是核心点,但它们可由 A 经其他核心点可达,所以也属于同一个聚类。点 N 是局外点,它既不是核心点,又不由其他点可达。
算法:
需要两个参数:ε (eps) 和形成高密度区域所需要的最少点数 (minPts)
它由一个任意未被访问的点开始,然后探索这个点的 ε-邻域,如果 ε-邻域里有足够的点,则建立一个新的聚类,否则这个点被标签为杂音。注意这个点之后可能被发现在其它点的 ε-邻域里,而该 ε-邻域可能有足够的点,届时这个点会被加入该聚类中。
伪代码:
DBSCAN(D, eps, MinPts) {
C = 0
for each point P in dataset D {
if P is visited
continue next point
mark P as visited
NeighborPts = regionQuery(P, eps)
if sizeof(NeighborPts) < MinPts
mark P as NOISE
else {
C = next cluster
expandCluster(P, NeighborPts, C, eps, MinPts)
}
}
}
expandCluster(P, NeighborPts, C, eps, MinPts) {
add P to cluster C
for each point P' in NeighborPts {
if P' is not visited {
mark P' as visited
NeighborPts' = regionQuery(P', eps)
if sizeof(NeighborPts') >= MinPts
NeighborPts = NeighborPts joined with NeighborPts'
}
if P' is not yet member of any cluster
add P' to cluster C
}
}
regionQuery(P, eps)
return all points within P's eps-neighborhood (including P)

sklearn中调用dbscan:
# -*- coding: utf-8 -*-
import numpy as np
from sklearn.cluster import DBSCAN
from sklearn import metrics
from sklearn.datasets.samples_generator import make_blobs
from sklearn.preprocessing import StandardScaler
##############################################################################
# Generate sample data
centers = [[1, 1], [-1, -1], [1, -1]]
X, labels_true = make_blobs(n_samples=750, centers=centers, cluster_std=0.4,
random_state=0)
X = StandardScaler().fit_transform(X)
##############################################################################
# Compute DBSCAN
db = DBSCAN(eps=0.3, min_samples=10).fit(X)
core_samples_mask = np.zeros_like(db.labels_, dtype=bool)
core_samples_mask[db.core_sample_indices_] = True
labels = db.labels_
# Number of clusters in labels, ignoring noise if present.
n_clusters_ = len(set(labels)) - (1 if -1 in labels else 0)
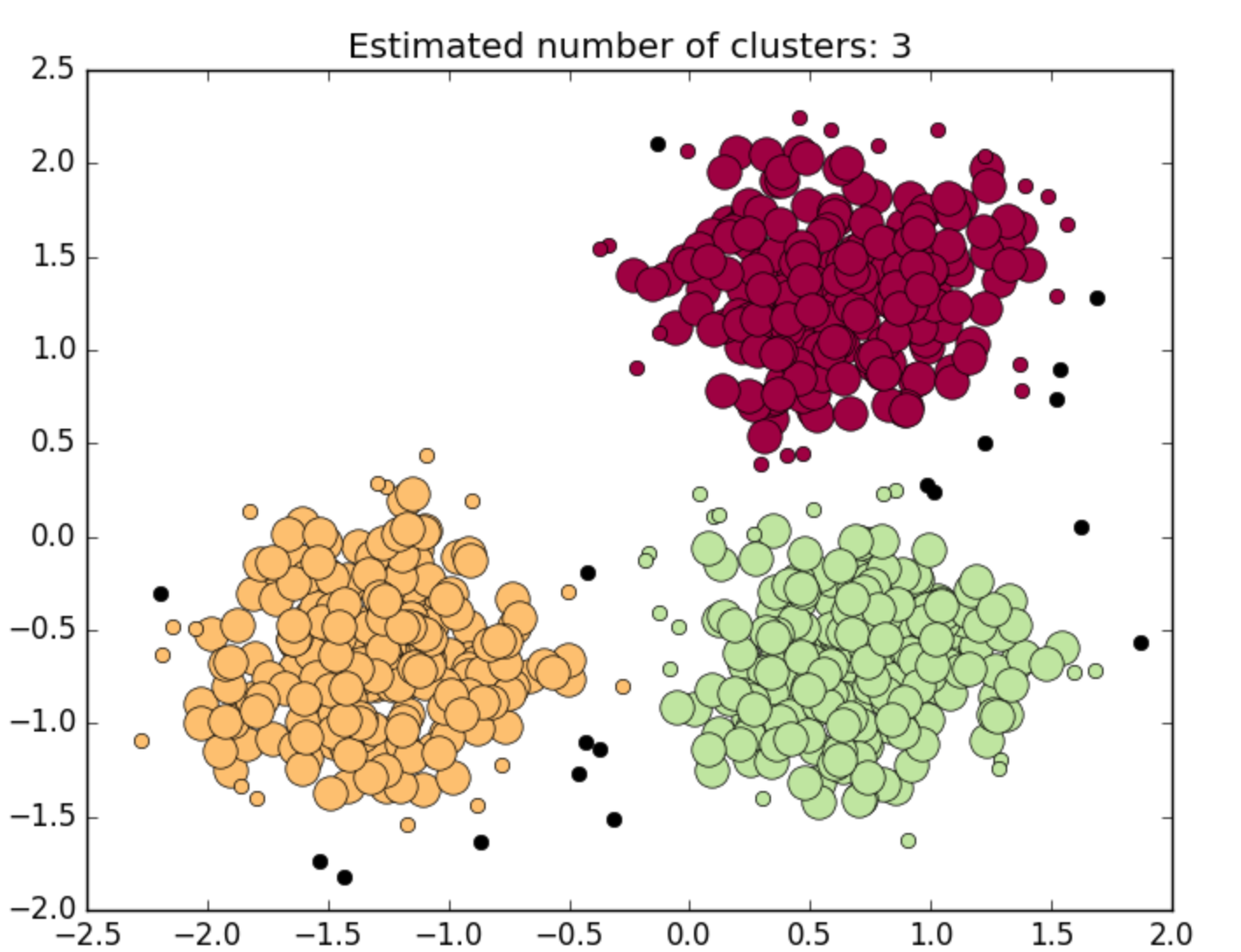
print('Estimated number of clusters: %d' % n_clusters_)
print("Homogeneity: %0.3f" % metrics.homogeneity_score(labels_true, labels))
print("Completeness: %0.3f" % metrics.completeness_score(labels_true, labels))
print("V-measure: %0.3f" % metrics.v_measure_score(labels_true, labels))
print("Adjusted Rand Index: %0.3f"
% metrics.adjusted_rand_score(labels_true, labels))
print("Adjusted Mutual Information: %0.3f"
% metrics.adjusted_mutual_info_score(labels_true, labels))
print("Silhouette Coefficient: %0.3f"
% metrics.silhouette_score(X, labels))
##############################################################################
# Plot result
import matplotlib.pyplot as plt
# Black removed and is used for noise instead.
unique_labels = set(labels)
colors = plt.cm.Spectral(np.linspace(0, 1, len(unique_labels)))
for k, col in zip(unique_labels, colors):
if k == -1:
# Black used for noise.
col = 'k'
class_member_mask = (labels == k)
xy = X[class_member_mask & core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=col,
markeredgecolor='k', markersize=14)
xy = X[class_member_mask & ~core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=col,
markeredgecolor='k', markersize=6)
plt.title('Estimated number of clusters: %d' % n_clusters_)
plt.show()
二.密度最大值算法:
密度最大值聚类是一种简洁优美的聚类算法, 可以识别各种形状的类簇, 并且参数很容易确定。
该聚类算法可以得到非球形的聚类结果,可以很好地描述数据分布,同时在算法复杂度上也比一般的K-means算法的复杂度低。
同时此算法的只考虑点与点之间的距离,因此不需要将点映射到一个向量空间中。
1.局部密度ρi:
![]()
2.高局部密度点距离δi:

在密度高于对象i的所有对象中,到对象i最近的距离,即高局部密度点距离
3.簇中心:
那些有着比较大的局部密度ρi和很大的高密距离δi的点被认为是簇的中心
4.异常点
高密距离δi较大但局部密度ρi较小的点
聚类过程:

左图是所有点在二维空间的分布, 右图是以ρ为横坐标, 以δ为纵坐标绘制的决策图。可以看到,1和10两个点的ρi和δi都比较大,作为簇的中心点。26、27、28三个点的δi也比较大,但是ρi较小,所以是异常点。