对医学3D图像中的病灶区域进行Detection时,需要提供Bounding Box标签。3D Slicer(4.10.2)中的Segment Editor与Legacy/Editor都可以对三维体数据中的ROI进行矩形标签框注。Segment Editor较为方便,虽然这个模块本身是用于像素级Segmentation的标注,但是也可以用来标注ROI的矩形框。本文介绍如何使用3D Slicer的Segment Editor模块对DICOM格式的CT数据进行矩形框标注;标注过程中,每个流程都可能会出现问题,建议先随意地标记几张slice,然后导出保存。当整个流程没有出现问题时,再开始正式地标记。
一、安装软件
现在最新的3D slicer是4.10.2版本的,官网可以免费下载。https://download.slicer.org/,下载完成后点击安装包傻瓜式安装 (安装路径不能有中文)。
另外,如果电脑用户名user是中文的话,有可能会出现问题,例如最后导出的文件夹为空,解决方法:在3D slicer的导航栏里的Edit – Application settings里,修改Module – Temporary directory为全英文路径: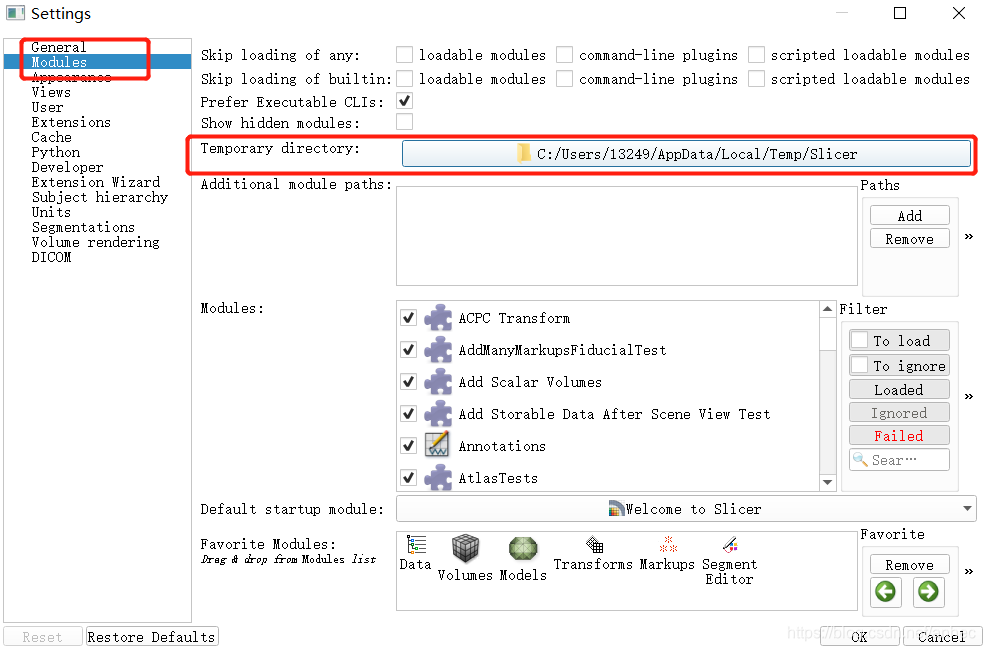
二、读取数据
打开3D slicer后,首先导入3D数据(DICOM格式)。
-
将包含.dcm文件序列的文件夹直接拖入3D Slicer,导入文件路径不能有中文,选择reader:
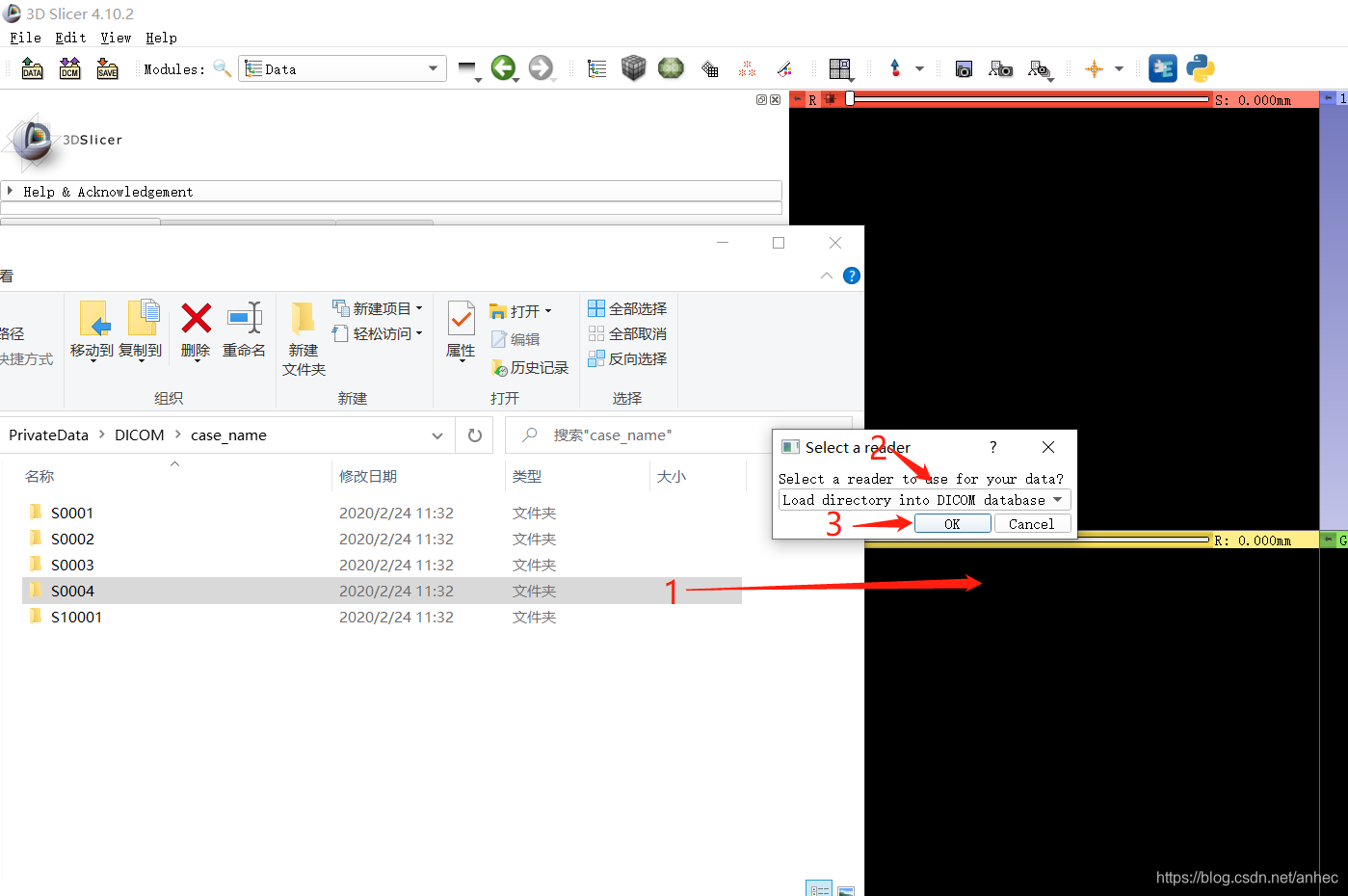
注意:一个病人有多个扫描的CT,选择薄层的,就是上图的S0004文件夹。
-
导入完成后,点击弹窗的OK:
然后会弹出DICOM Brower:
如果没有自动弹出上图的DICOM Brower的话,点击3D slicer左上角的DCM按钮:
-
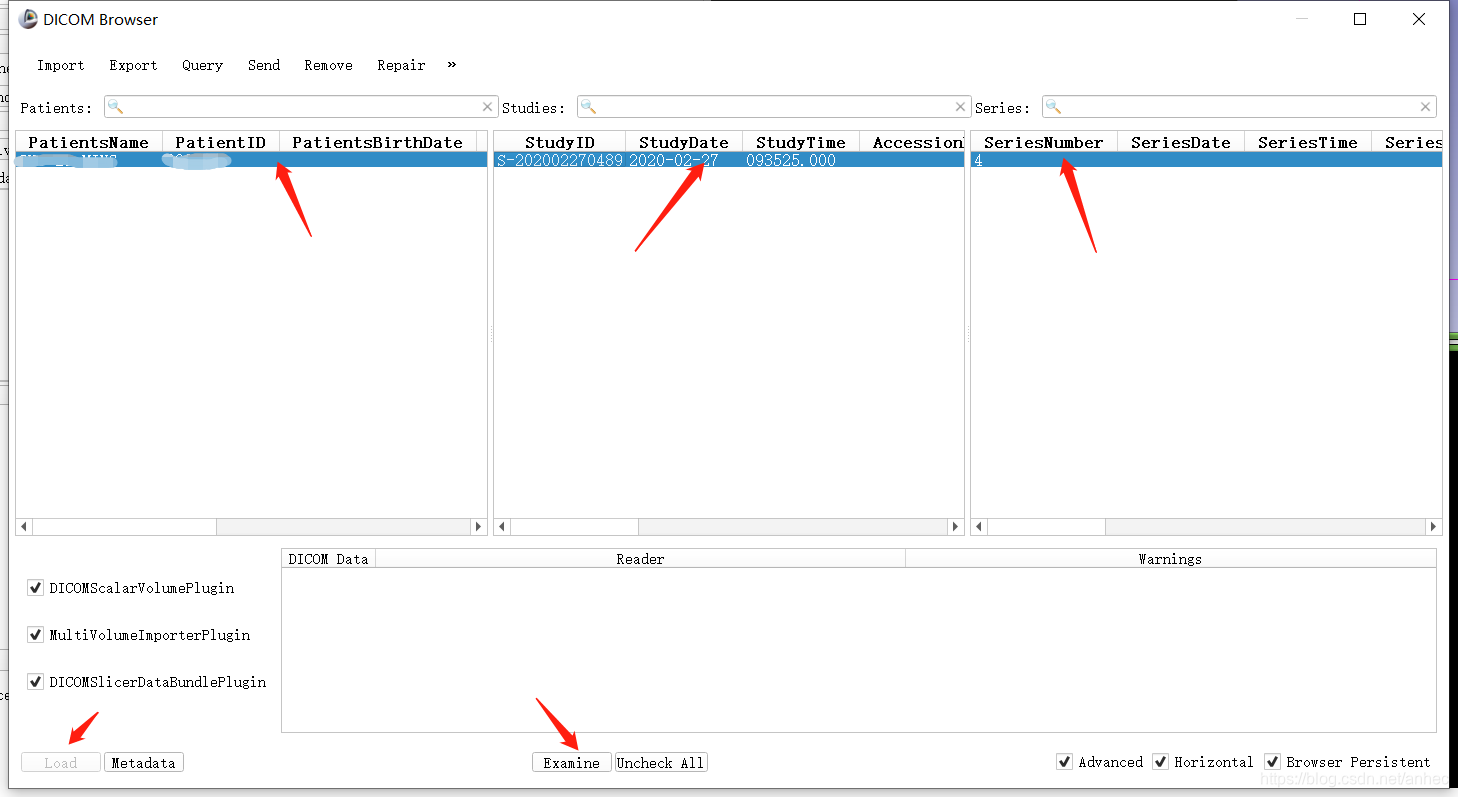
在DICOM Brower中,先选中本次要打标注的case,选择的时候,左中右三列都要选择到。然后点击Examine,再点击load(有些3D slicer没有Examine按钮,这可能是因为安装的路径有问题,退出重新安装到全英文路径下):
-
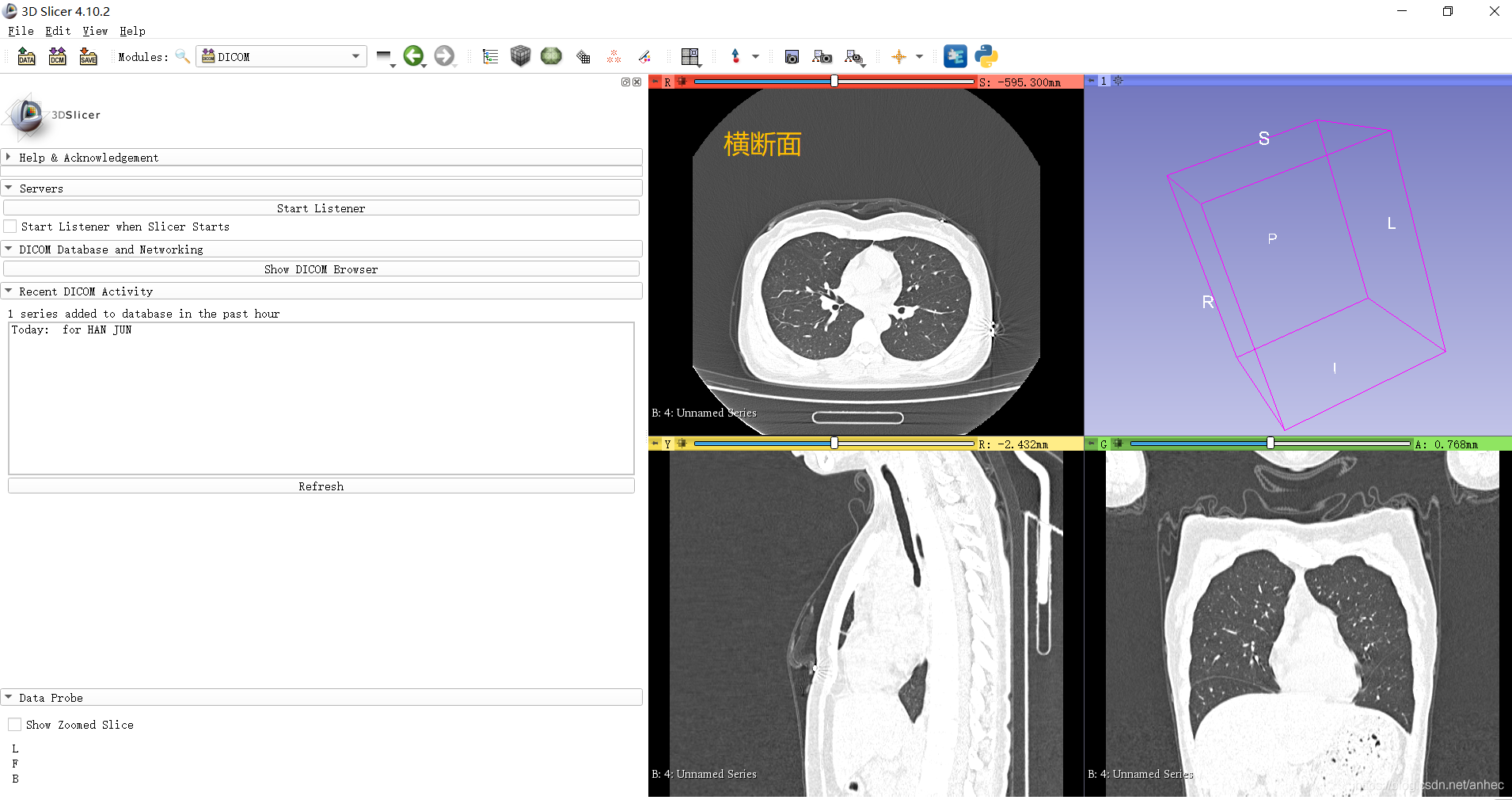
点击load之后,CT的三个维度的2D切片会出现在3D slicer中,退出DICOM Brower。3D slicer的左上视图为CT的横断面,我们在横断面中进行标注:
一些基本的交互操作:https://blog.csdn.net/Huadong_eddy/article/details/80392595 (一定要看)
三、标注工具
-
切换3D slicer的功能模块至Segment Editor:
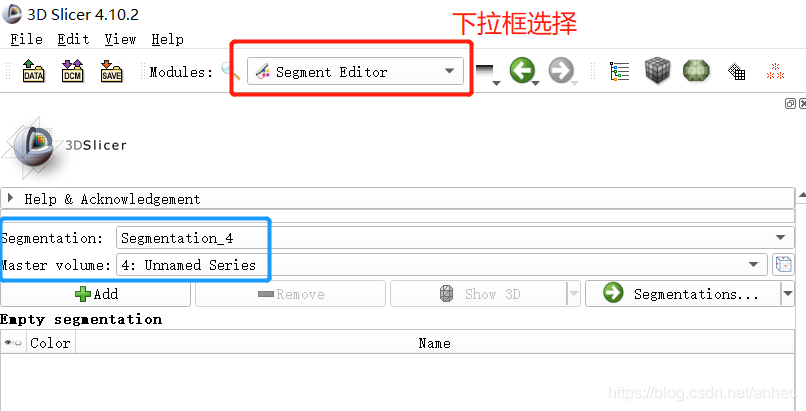
上图蓝色框内,Segmentation表示本次label volume的名字,Master volume表示右边正在显示的Volume的名字,按默认的就行。 -
点击Add,新建一个类别的标签:
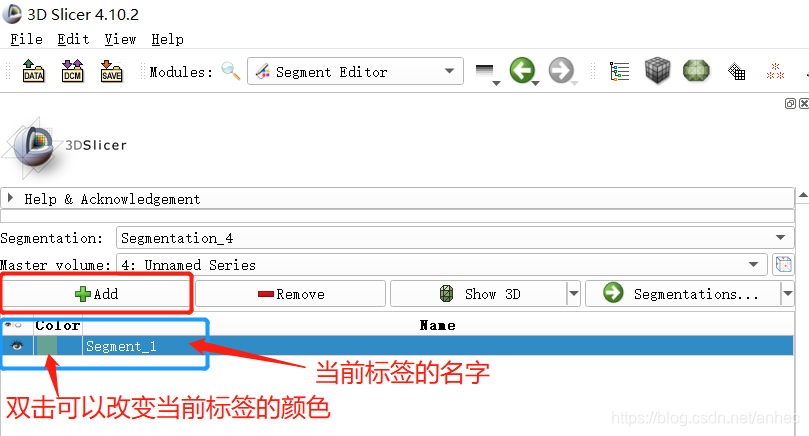
一个Case只需要一个Segment就行,即只需要点击一次Add。
-
下方的effects内,显示了很多种标注的工具,我们需要用到的有:
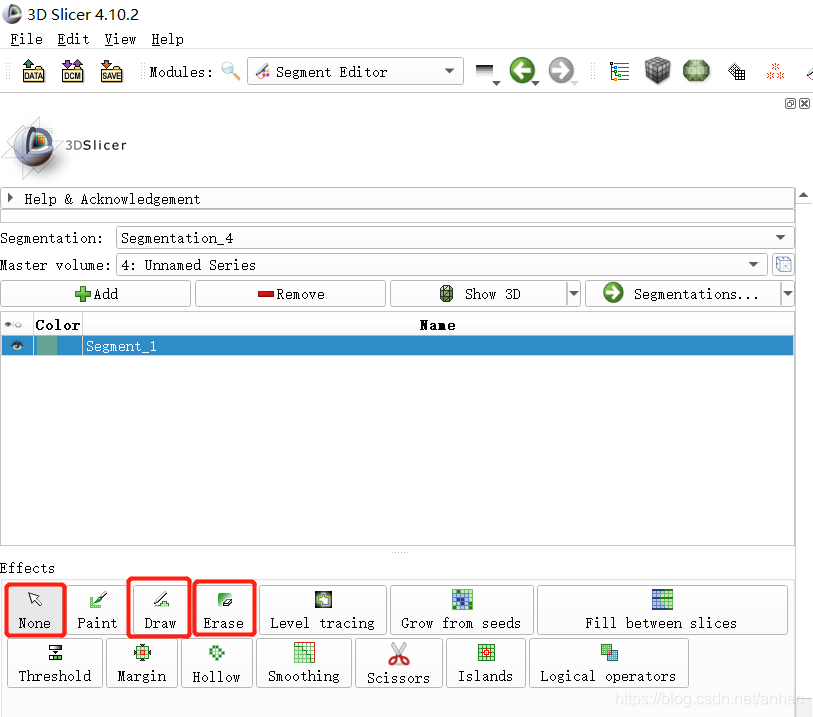
初始是在None的状态下;Draw为画笔工具;Erase为橡皮擦工具。 -
点击Draw画笔,可以看到该工具的操作方式:
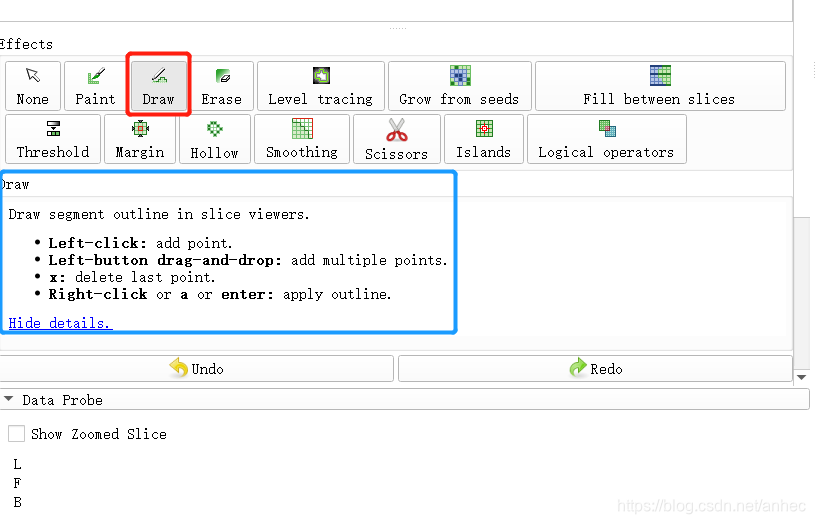
在横断面视图内,点击左键新加点,按住左键拖拽鼠标可以画线(加多个点),点击右键可以将所有加的点闭合起来;按x键删除上一个点;按ctrl+z可以撤销上一组操作。
四、示例标注
-
在横断面内,滚动鼠标浏览slice,找到有病灶的slice,可以通过前文链接里的那些交互操作方式改变窗宽窗位,或者放缩移动图像,来更加有效地查找病灶(注意有部分交互操作方式只有在None状态下才可以进行),例如下图的右下方有个病灶:
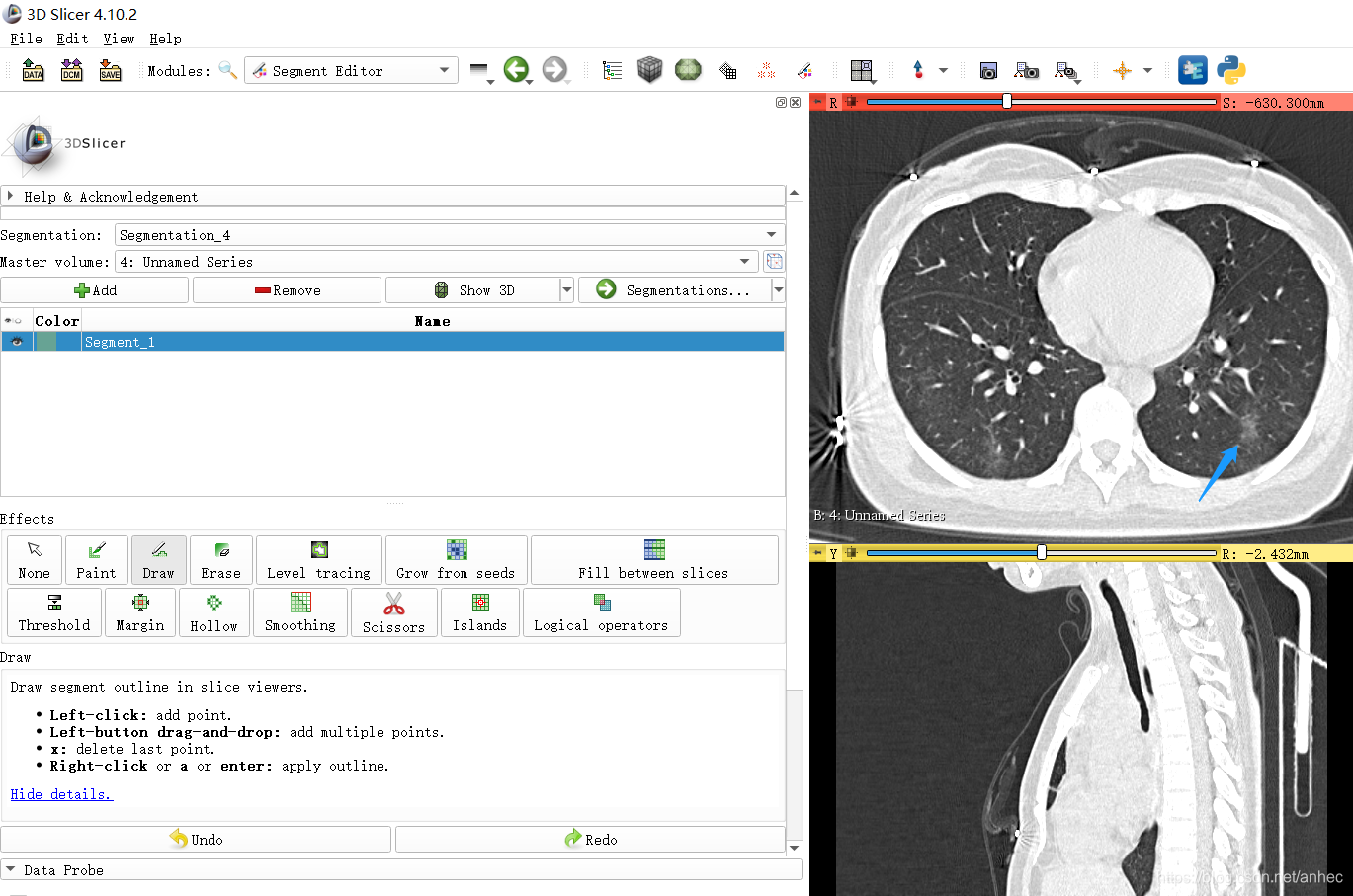
-
使用Draw工具,围绕着病灶左键点3-4个点,然后点击右键,生成一个比较扭曲的矩形包围住这个病灶就行了。
左键点了四个点: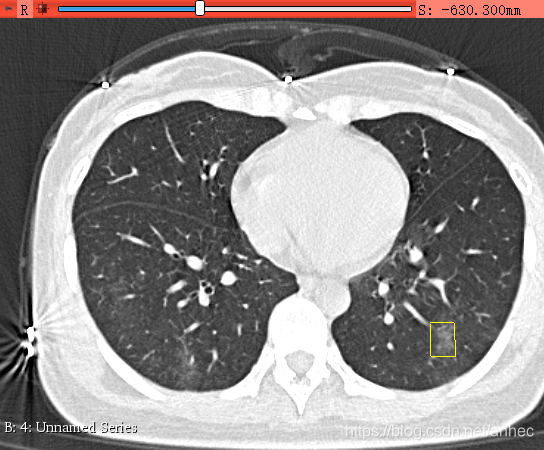
右键生成闭合多边形(伪矩阵):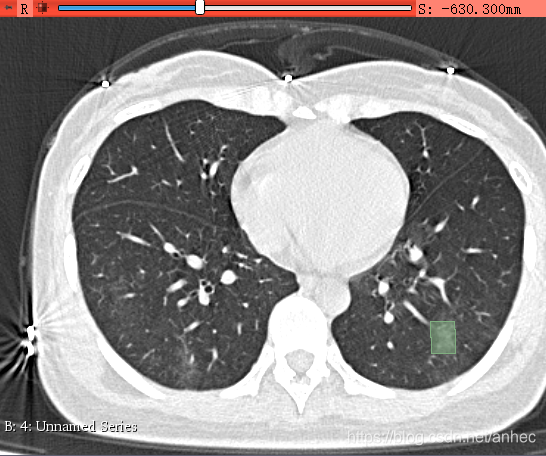
一个病灶区域就标好了。注意:虽然说在Detection时需要的是矩形框,但是只要所生成的多边形包围着病灶就OK了,后期把多边形转为矩形就行了,即包围多边形的最小矩阵。
标注的要求:病灶区域的多边形,不是矩阵也行,也不需要严格地沿着病灶区域的边界画,把病灶区域给包围住就行,但是尽量不要把非病灶区域给包了。
-
标完这个slice的病灶之后,滚动滑轮到下一张slice继续接着标就行,不需要在左边栏再次点击Add添加Segment。把这个Case的所有横断面slice都找一遍,然后把每张slice中的所有病灶区域用伪矩形框给标注出来就行了。
注意:一个Case中,很多slice可能没有病灶,没有病灶的slice就不需要标了;有些slice会有多个病灶,就需要用多个伪矩形把每个病灶都标出来;大部分病灶会跨越多张slice。
-
标注时,点击当前标签名字前面的小眼睛,可以选择显示/不显示标签;
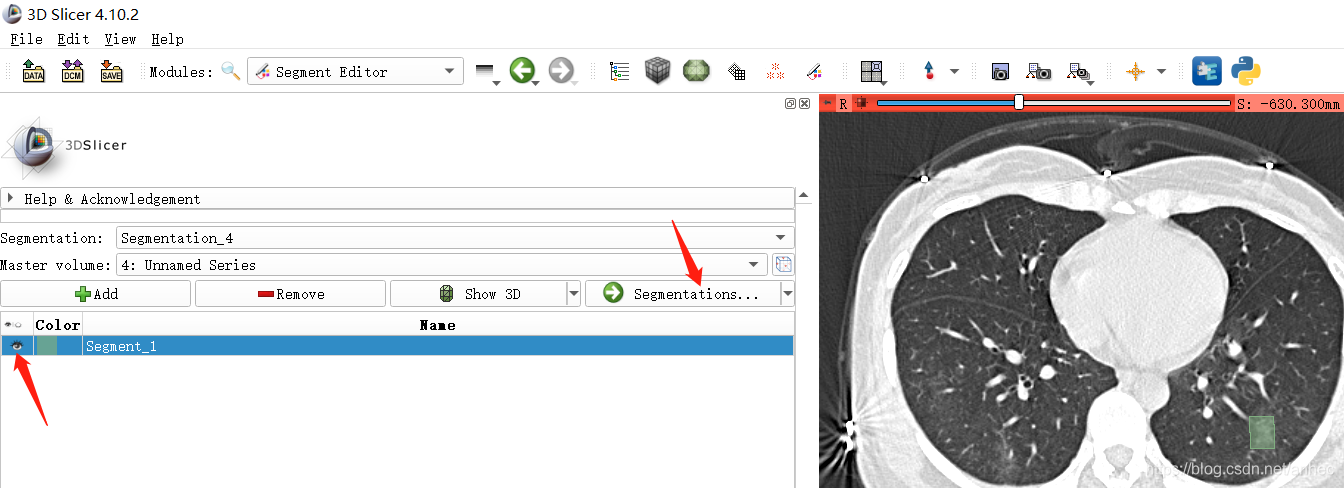
点击Segmentations可以调节标签的透明度: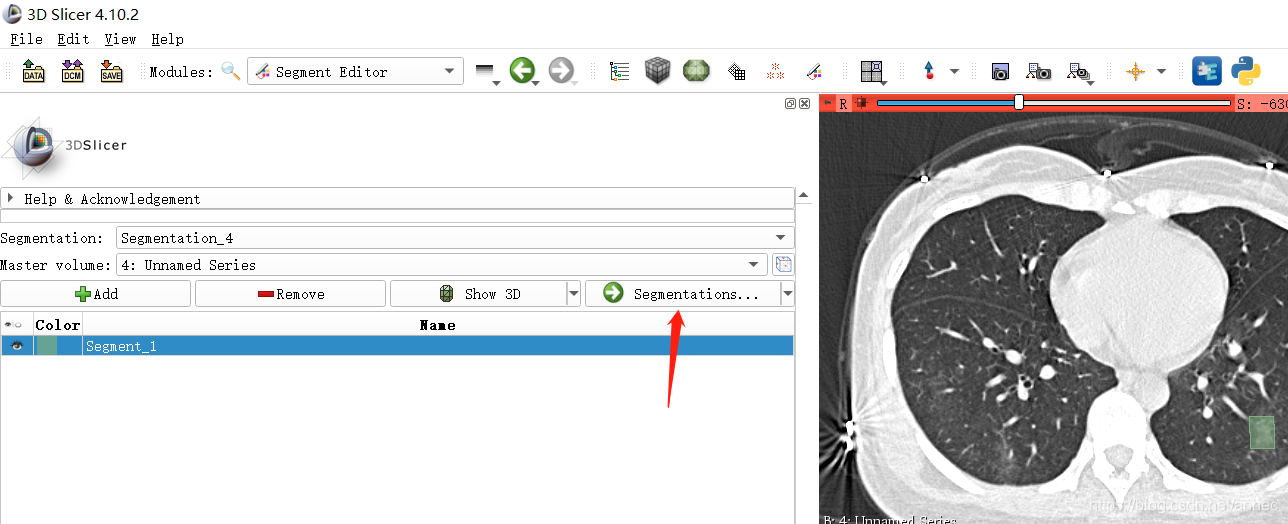
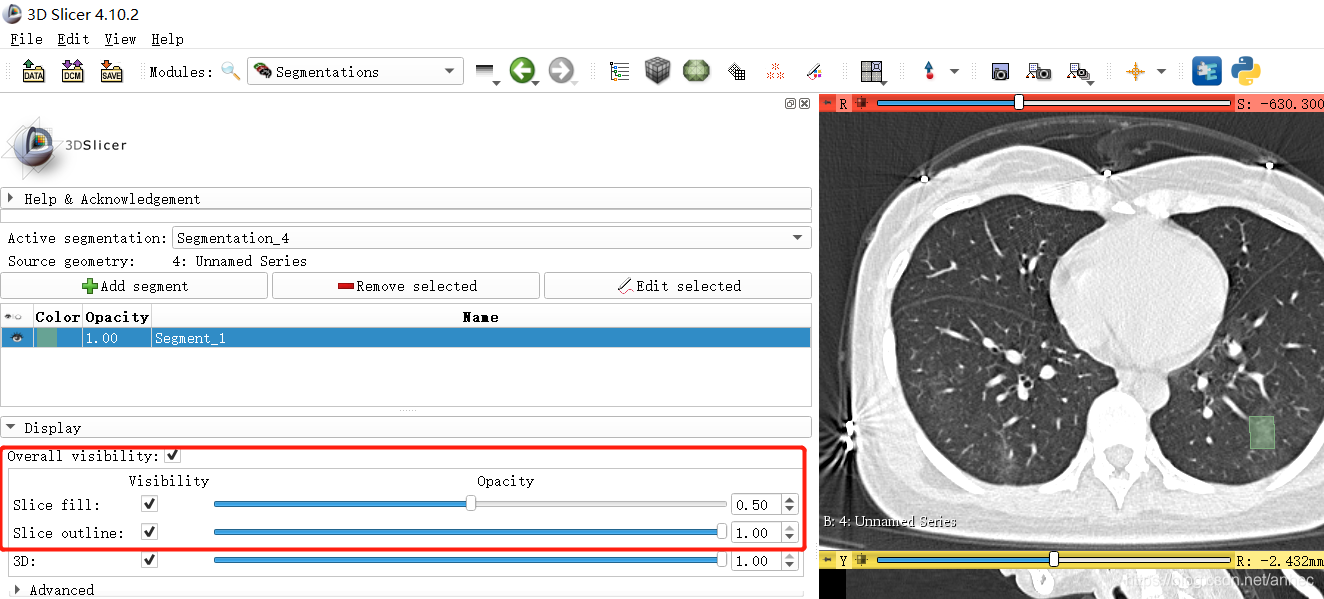
调完之后按Edit selected返回继续标注。
五、保存标注
把Case里所有横断面slice的所有病灶都标记完成之后,需要保存标签。
-
切换至3D slicer的Data模块:
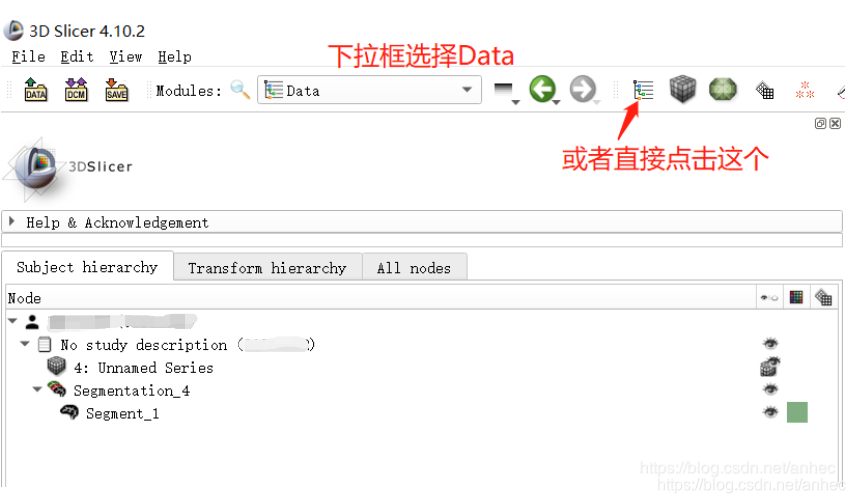
-
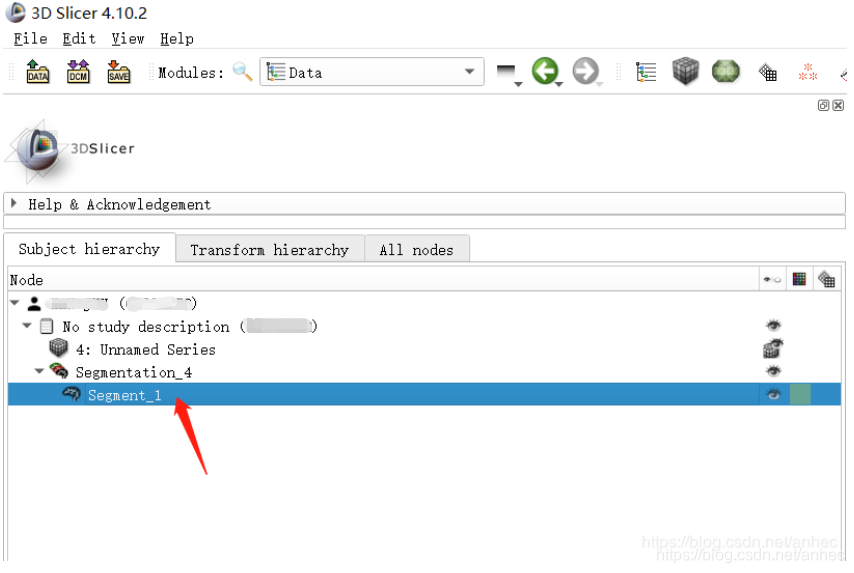
选中刚刚的标签的名字,右键,选择Export visible segments to binary labelmap
-
生成一个LabelMapVolume:
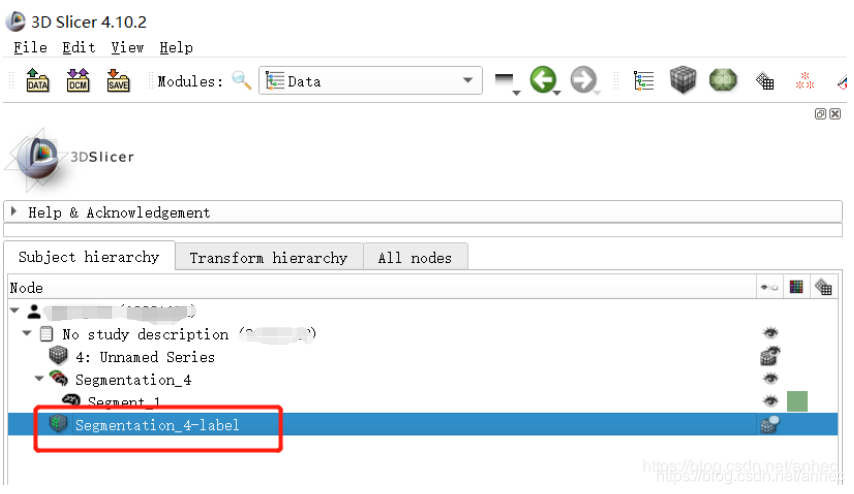
-
选中并右键这个LabelMapVolume,选择Export to Dicom,即选择将标签导出为DICOM格式,会弹出Dicom Export弹窗:
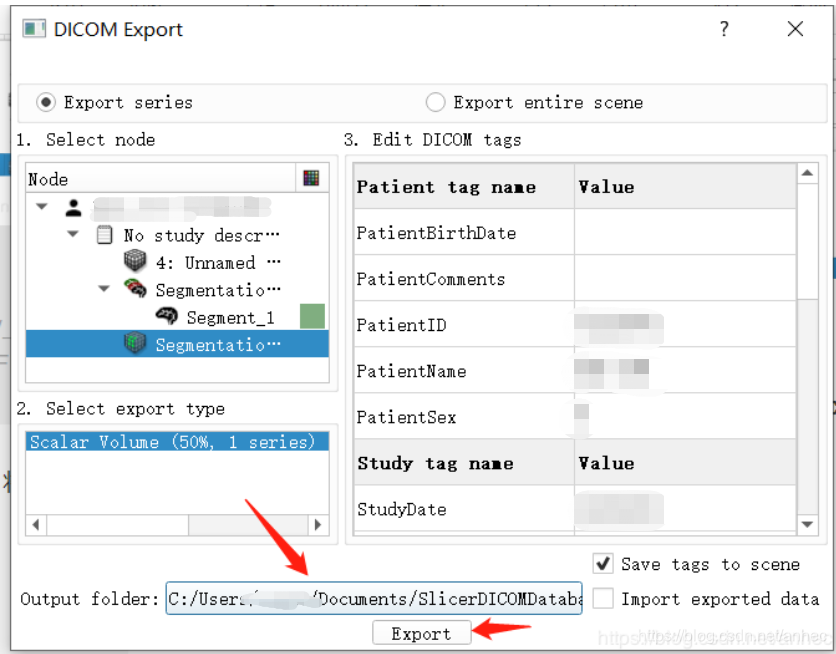
修改导出的保存路径后(使用全英文路径;不要用默认的路径,不然默认路径下只会生成.sql文件) ,点击Export,显示Export successful就成功了:
检查一下导出的DICOM序列的文件夹,建议手动更改一下文件夹名字,改成病例的名字,以防多个病例之间弄混。
六、删除数据
当标记完一个case,并将标签成功导出之后,建议删除3D Slicer中的数据,以防标记下一个case时内存溢出。
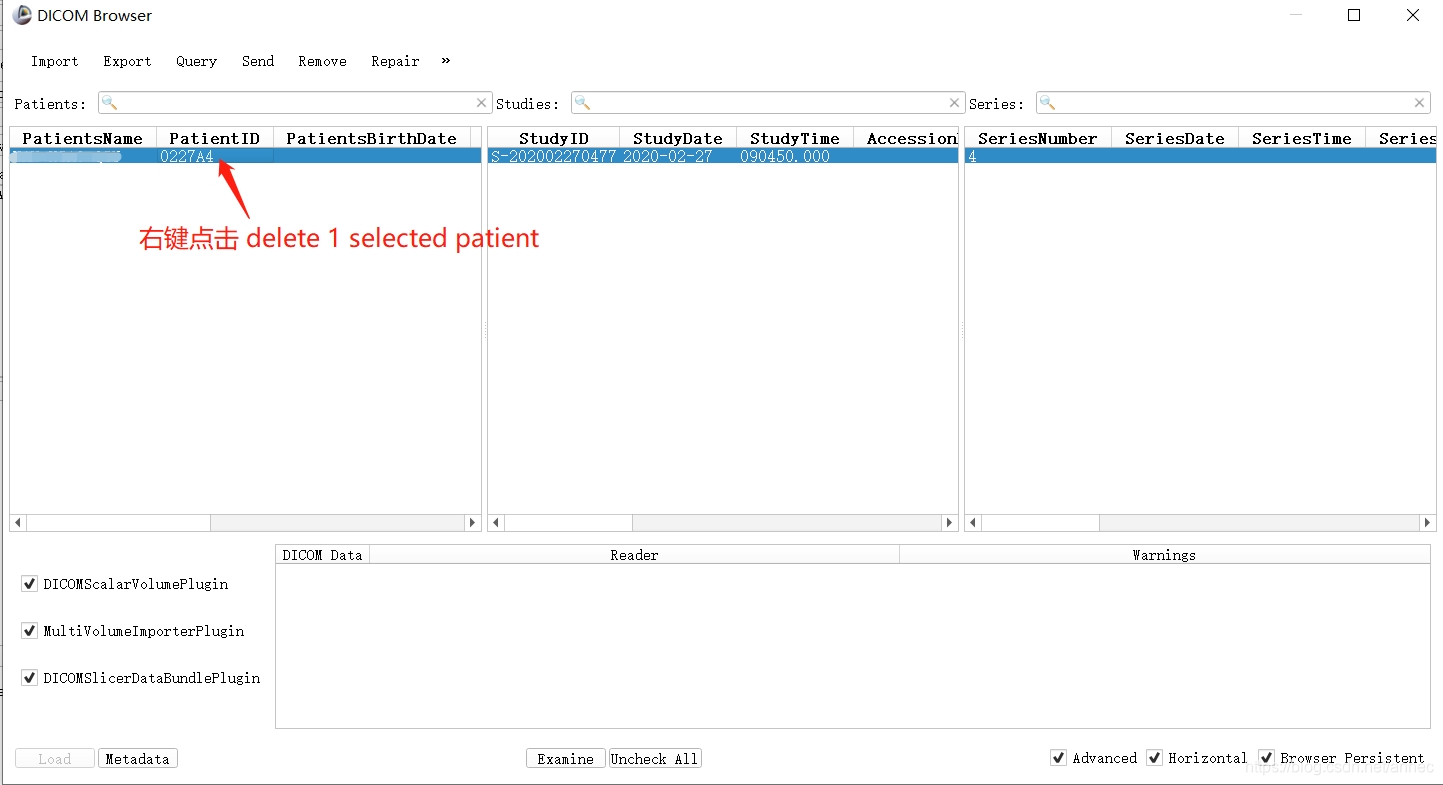
- 删除导入历史:在DICOM Brower里面,选中病例,右键,点击delete 1 selected patient:
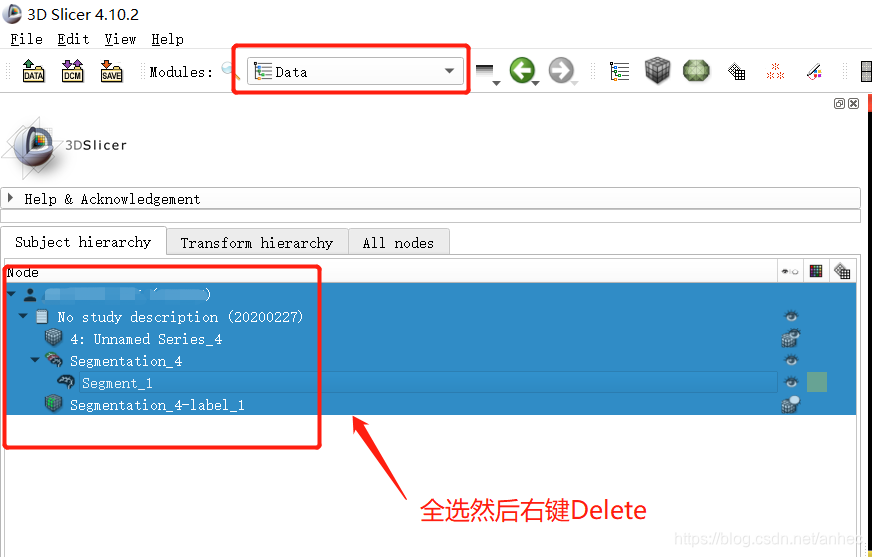
- 删除3D slicer中的数据:在Data模块里,将数据全部选中,然后右键Delete:
删除3D Slicer中的数据并不会删除原始的在文件夹里的CT和已经导出的label。