随着人类基因组计划(Human Genome Project)即全部核苷酸测序的即将完成,人类基因组研究的重心逐渐进入后基因组时代(Postgenome Era),向基因的功能及基因的多样性倾斜。通过对个体在不同生长发育阶段或不同生理状态下大量基因表达的平行分析,研究相应基因在生物体内的功能,阐明不同层次多基因协同作用的机理,进而在人类重大疾病如癌症、心血管疾病的发病机理、诊断治疗、药物开发等方面的研究发挥巨大的作用。它将大大推动人类结构基因组及功能基因组的各项基因组研究计划。生物信息学在基因组学中发挥着重大的作用, 而另一项崭新的技术——基因芯片已经成为大规模探索和提取生物分子信息的强有力手段,将在后基因组研究中发挥突出的作用。基因芯片与生物信息学是相辅相成的,基因芯片技术本身是为了解决如何快速获得庞大遗传信息而发展起来的,可以为生物信息学研究提供必需的数据库,同时基因芯片的数据分析也极大地依赖于生物信息学,因此两者的结合给分子生物学研究提供了一条快捷通道。
本文介绍了几种常用的基因功能分析方法和工具:
一、GO基因本体论分类法
最先出现的芯片数据基因功能分析法是GO分类法。Gene Ontology(GO,即基因本体论)数据库是一个较大的公开的生物分类学网络资源的一部分,它包含38675 个EntrezGene注释基因中的17348个,并把它们的功能分为三类:分子功能,生物学过程和细胞组分。在每一个分类中,都提供一个描述功能信息的分级结构。这样,GO中每一个分类术语都以一种被称为定向非循环图表(DAGs)的结构组织起来。研究者可以通过GO分类号和各种GO数据库相关分析工具将分类与具体 基因联系起来,从而对这个基因的功能进行描述。在芯片的数据分析中,研究者可以找出哪些变化基因属于一个共同的GO功能分支,并用统计学方法检定结果是否 具有统计学意义,从而得出变化基因主要参与了哪些生物功能。
EASE(Expressing Analysis SystematicExplorer)是比较早的用于芯片功能分析的网络平台。由美国国立卫生研究院(NIH)的研究人员开发。研究者可以用多种不同的格式将芯片中得到的基因导入EASE 进行分析,EASE会找出这一系列的基因都存在于哪些GO分类中。其最主要特点是提供了一些统计学选项以判断得到的GO分类是否符合统计学标准。EASE 能进行的统计学检验主要包括Fisher 精确概率检验,或是对Fisher精确概率检验进行了修饰的EASE 得分(EASE score)。
由于进行统计学检验的GO分类的数量很多,所以EASE采取了一系列方法对“多重检验”的结果进行校正。这些方法包括弗朗尼校正法
(Bonferroni),本杰明假阳性率法(Benjamini falsediscovery rate)和靴带法(bootstraping)。同年出现的基于GO分类的芯片基因功能分析平台还有底特律韦恩大学开发的Onto-Express。 2002年,挪威大学和乌普萨拉大学联合推出的Rosetta系统将GO分类与基因表达数据相联系,引入了“最小决定法则”(minimal decision rules)的概念。它的基本思想是在对多张芯片结果进行聚类分析之后,与表达模式不相近的基因相比,相近的基因更有可能参与相同的生物学功能的实现。
GCBI分析平台: 是一个新型的网络分析平台,全称是Gene-Cloud of Biotechnology Information。
GO-Analysis是对基因进行显著性功能(GO)的分析。由于GO的条目中包含功能的层级关系,从而GO中包含基因的数目变化较大,通常在一到几百个基因之间。GCBI的功能分析主要用Fisher精确检验,即利用如下的四格表:
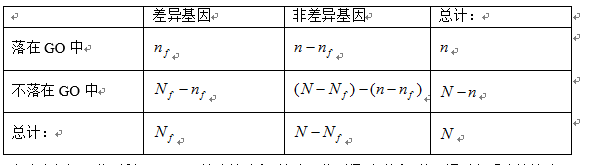
在这个假设下分别利用fisher精确检验和 检验,分别得到 值和 值,通过多重比较检验,确定GO的FDR。最后得出显著性GO,完成GO-Analysis。
ENRICHMENT计算公式为:
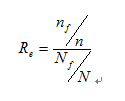
比较著名的基于GO分类法的芯片数据分析网络平台还有七十多个,表1列举了其中的一部分。
|
Name |
Internet Site |
|
GCBI |
https://www.gcbi.com.cn/gclab |
|
Onto-Tools |
http://vortex.cs.wayne.edu/projects.htm |
|
ROSETTA |
http://rosetta.lcb.uu.se/general/ |
|
GOToolBox |
http://burgundy.cmmt.ubc.ca/GOToolBox/ |
|
GOstat |
http://gostat.wehi.edu.au/ |
|
GFINDer |
http://www.medinfopoli.polimi.it/GFINDer/ |
|
FatiGO |
http://www.fatigo.org/ |
|
EASE |
http://david.abcc.ncifcrf.gov/ease/ease.jsp |
表1 用GO 分类法进行芯片功能分析的网络平台
二、Pathway通路分析法
通路分析是现在经常被使用的芯片数据基因功能分析法。与GO分类法(应用单个基因的GO分类信息)不同,通路分析法利用的资源是许 多已经研究清楚的基因之间的相互作用,即生物学通路。研究者可以把表达发生变化的基因列表导入通路分析软件中,进而得到变化的基因都存在于哪些已知通路中,并通过统计学方法计算哪些通路与基因表达的变化最为相关。现在已经有丰富的数据库资源帮助研究人员了解及检索生物学通路,对芯片的结果进行分析。主要的生物学通路数据库有以下两个:
①KEGG 数据库:迄今为止,KEGG数据库(Kyotoencyclopedia of genes and genomes)是向公众开放的最为著名的生物学通路方面的资源网站。在这个网站中,每一种生物学通路都有专门的图示说明。
②BioCarta 数据库:BioCarta 是一家生物技术公司,它在其公共网站上提供了用于绘制生物学通路的模板。研究者可以把符合标准的生物学通路提供给BioCarta数据库。 BioCarta数据库不会检验这些生物学通路的质量,因此其中的资源质量参差不齐,并且有许多相互重复。然而BioCarta数据库数据量巨大,且不同 于KEGG数据库,包含了大量代谢通路之外的生物学通路,所以也得到广泛的应用。
最先出现的通路分析软件之一是GenMAPP(gene microarray pathwayprofiler)。它可以免费使用,其最新版本为Gen-MAPP2。在这个软件中,使用者可以用几种灵活的文件格式输入自己的表达谱数据,GenMAPP的基因数据库包含许多从常用的资源中得到的物种特异性的基因注释和识别符(ID)。这些ID可以将使用者输入的基因与不同的生物学通路 的基因联系起来。这些生物学通路存在于GenMAPP 的MAPP文件中。MAPP文件需要时常下载更新。它包含有许多KEGG生物学通路,一些GenMAPP自己的生物学通路和许多GO分类的MAPP 文件,全部操作简单明了。而且依靠其自带的MAPPBuilder和MAPPFinder 两个软件,使用者可以自己绘制生物学通路和对MAPP 文件进行检索。由于使用者可以自己绘制生物学通路保存为MAPP 格式,这个文件很小易于在网络上传播,所以GenMAPP数据库更有利于研究者之间的及时交流。由于上述特点,GenMAPP数据库及软件仍是现今免费平台里应用比较广泛的。
2004年推出的Pathway Miner也是应用较为广泛的免费通路分析网络平台,由美国亚利桑那大学癌症中心建立维护,其最突出的特点就是信息全面,操作简便。使用者可以在这个网站中获得单个基因的序列、功能注释,以及有关它们编码的蛋白结构功能,组织分布,OMIM等信息。对于通路分析部分,使用者给出基因列表及他们的表达变化值,网站可以根据三大公用的通路数据库:KEGG、GenMAPP 和BioCarta,生成变化基因参与的通路,并用fisher 精确概率检验。PathwayMiner自动把得到的通路分成两大类:代谢通路和细胞调节通路。方便使用者根据不同的研究目的选择需要查看的结果。
在 2006年国内也开发了用于通路分析的网络平台,即KOBAS(KO-Based Annotation System),其基于KEGG数据库建立,由北京大学生命科学院开发和维护。其特点是可直接采用基因或蛋白质的序列录入基因,并对录入的基因列表进行 KO 注释。对于结果的可靠性检验提供了四种统计方法。使用者可以在网站进行注册,网站会为使用者保存输入的数据,方便日后直接调用。最近推出的软件 Eu.Gene 整合了来自KEGG,Gen-MAPP 以及Reactome 的通路数据,并采用fisher 精确概率检验及基因集富集分析(Gene Set Enrichment Analysis, GSEA)来检验结果是否具有统计学意义。
2014年国内的第二款网络分析平台GCBI也上线了,不过GCBI网络分析平台不仅仅是针对通路分析。最后会详细介绍一下。
表2 列举了部分通路分析的网络平台及它们的网址。
|
Name |
Internet Site |
|
GenMAPP |
http://www.genmapp.org/ |
|
PathwayMiner |
http://www.biorag.org/pathway.html |
|
KOBAS |
http://kobas.cbi.pku.edu.cn |
|
GEPAT |
http://gepat.bioapps.biozentrum.uni-wuerzburg.de/GEPAT/index.faces |
|
VitaPad |
http://bioinformatics.med.yale.edu/group |
|
KEGGanim |
http://biit.cs.ut.ee/kegganim/ |
|
WholePathwayScope |
http://www.abcc.ncifcrf.gov/wps/wps_index.php |
|
VisANT 3.0 |
http://visant.bu.edu/ |
|
Eu.Gene |
http://www.ducciocavalieri.org/bio/Eugene.htm |
|
GCBI |
https://www.gcbi.com.cn/gclib/html/dictSearch/VFA1Mw |
表二通路分析网络平台
三、基因调控网络分析
通路分析法是芯片功能分析的有力工具之一,其与GO分类法的主要区别也正是它的弱点。在生物反应的过程中,发生表达变化的基因通常不只局限在一个通路中, 而是存在于由许多调控因子和通路参与的复杂调控网络中。生物调控网络十分复杂,并没有现成的文献和数据库供参考。而且,把芯片中发生表达变化的基因放在生物调控网络的水平来看,它们通常在多个通路中都有分布,而每个通路只包含几个发生表达变化的基因。这就解释了为什么有些通路只有部分基因表达发生变化,而且表达变化的趋势在整个通路水平上不一定是一致的。
进行生物调控网络的研究需要更多的数据库及分析工具的支持。比如需要关于基因组调控序列(启动子和增强子)的信息,现在已经有许多关于转录因子结合位点(transcriptionfactor binding site, TFBS)的数据库可以满足这个要求,如TRANSFAC及JASPAR。而且芯片检测的基因变化应该深入到转录本水平,因为不同的转录本的转录可能是由不同的启动子启动的。外显子连接芯片(exonjunction microarray)将基因组中外显子与外显子之间的连接序列做成36nt 的探针点到芯片上,与样本mRNA 进行杂交后可检测出样本中多外显子基因pre-mRNA 的剪接状况。
转录调节控制基因表达,调控不同组织中的细胞在各种生理条件及外界刺激下的反应。不同于原核细胞,真核细胞的转录调节涉及大量转录因子的相互作用,而且基因组调控序列不只位于启动子,还包括内含子及许多基因下游序列。所以真正了解真核细胞的基因调控网络是一项非常艰巨的工作。用基因调控网络来分析基因芯片数据还需要更多信息及技术的支持。
四、(差异基因+GO&Pathway analysis+网络图)一键式分析
常见的芯片功能分析有差异分析、GO&Pathway analysis、网络分析,一项差异分析得到的差异基因过多,不利于后面的研究,做芯片功能分析的时候一般是选择多项联合分析,有的时候为了研究的需要还会用到多种类型的芯片数据,比如microRNA-gene-lncRNA的网络图分析就用到了两种芯片。
GCBI一键式分析平台:这个平台可以进行多种芯片的功能分析。
目前支持的分析:差异分析、GO analysis、pathway analysis、STC、STC GO、pathway-Network、Signal-Network、gene-Coexpression-Network、lncRNA-Gene-Network、miRNA-Gene-Network、 miRNA-GO-Network等。
差异分析:为了得到可验证性的高质量差异结果,GCBI实验室推荐每个分组(Each Group)的样本数不少于3。而且为了保证后续交叉分析(如miRNA与mRNA联合分析,多次差异结果的交集、并集或扣除等)结果的可靠性,GCBI实验室推荐每种平台类型的芯片总样本数不少于10。
聚类图:下面给出GCBI实验室处理聚类图的一些步骤细节:
(1)首先计算基因与样本间的相关性,即计算基因间的皮尔森Pearson相关系数ρij,以1−ρij作为基因i与j之间的距离按照先基因、后样本的顺序,将基因表达数据进行层次聚类,连接方法为AverageLink。
(2)然后对差异基因表达值做中位数标准化,数学表示为:
gij= gij-median(gi*)
(3)取标准化后数据信号值的10与90分位数对整个差异表达数据做最低值与最高值数值截断,将上述截断值按照颜色层级分为20个渐变状态,可见于聚类图左上角的标示。
(4)最后在聚类图的上方标示了样本的分组信息,即相同分组的样本标示为同一个颜色。
其他分析方法见www.gcbi.com.cn的实验室。
进行芯片功能分析的方法还有很多,最常见的软件就是R语言、perl语言等,还有一些分析平台,如国外的七桥基因、国内的KOBAS、GCBI等,具体的选择还要根据自己的研究来决定。