http://software.broadinstitute.org/gsea/index.jsp
GSEA(Gene Set Enrichment Analysis)是一种生物信息学的计算方法,用于确定是否存在这样一个基因集,能在两个生物学状态中显示出显著的一致性的差异。表达谱数据里的基因数目众多,我们需要对基因进行功能注释,看哪些基因属于同一通路,以及该通路上的上调、下调情况,这就是富集分析了。
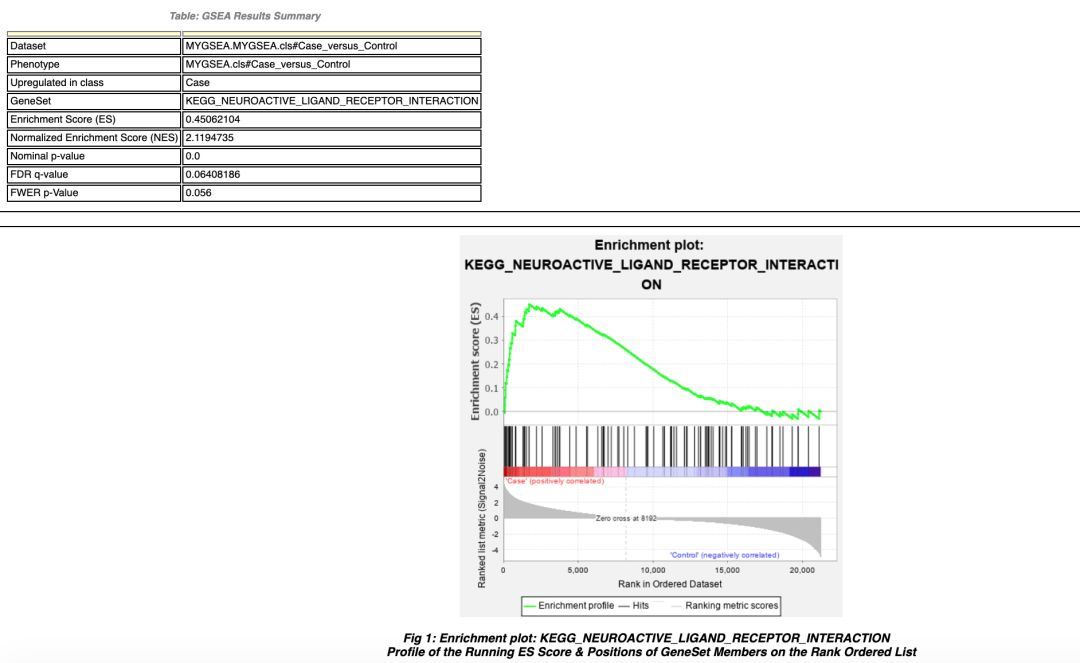
例如2019年4月在Cancer cell(PMID 30991027)上发表的一篇文章中有一张主图,就是通过GSEA分析对RNA-seq的数据进行解读,如下
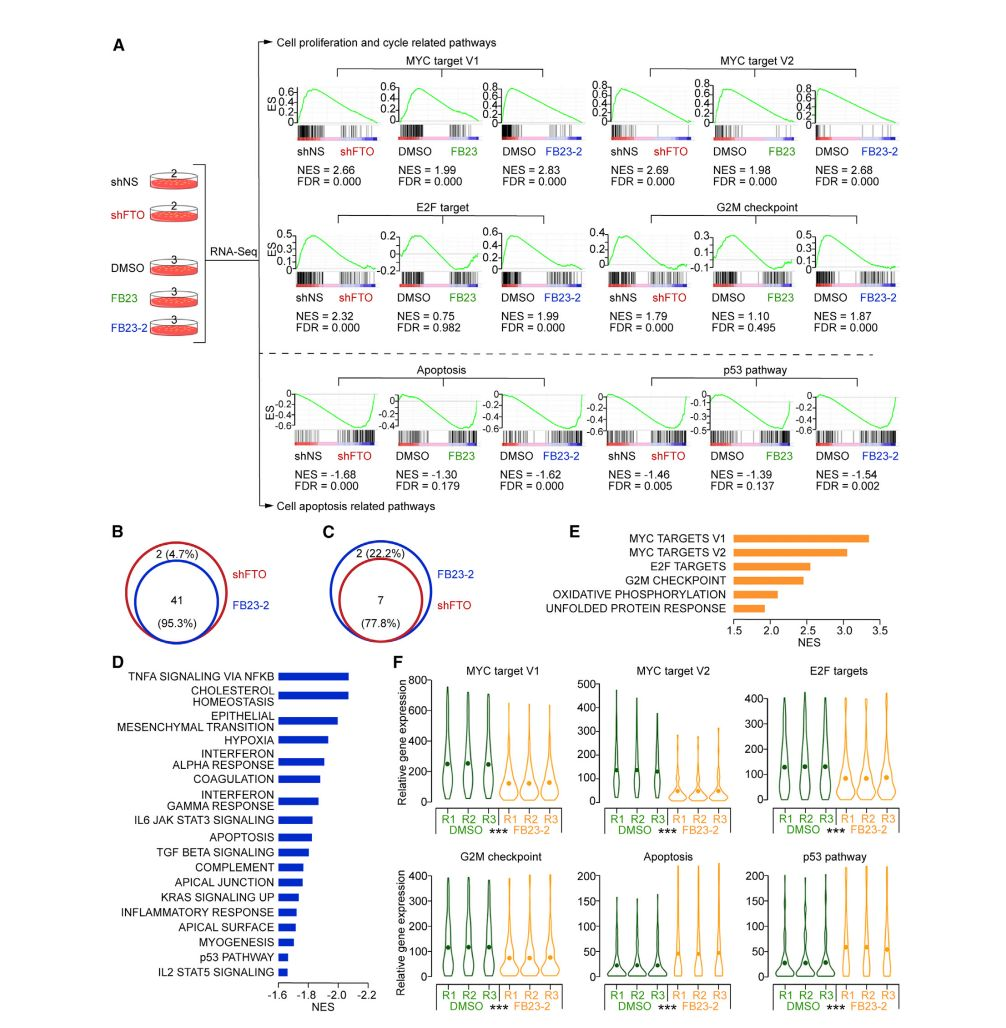
在上图中,图A是经典的GSEA富集图,图B为GSEA得到的通路上调或下调的韦恩图,图C和图D是GSEA分析的NES值,图E是GSEA通路基因表达值。
也就是说,只需要2(实验条件) X 3(生物学重复)个RNA-seq的样本,我们就可以做出上图。
做转录组分析时,大家通常会先筛选差异表达基因,然后再对这些差异表达基因进行功能富集分析。可能不少小伙伴会发现这种情况,就是因为差异基因过少而富集目标/相关的功能/通路,或者差异表达基因虽然很多,但是没有命中到感兴趣的通路或者GO功能。这种先做差异基因筛选的方式,可能由于筛选参数的设置不同,导致一些关键信息的“漏网”。
这种情况下,就可以试试GSEA分析。GSEA无需先做差异分析,会保留更多更多更全面的关键信息。可以帮助我们找到那些差异不是很明显但基因差异趋势很一致的功能基因集。
当然这两种思路没有说哪个更好,实际应用中能解决问题即可。
GSEA的分析步骤
1. GSEA软件的下载
直接通过GSEA官网进行下载
http://software.broadinstitute.org/gsea/index.jsp
2. 准备GSEA的输入文件
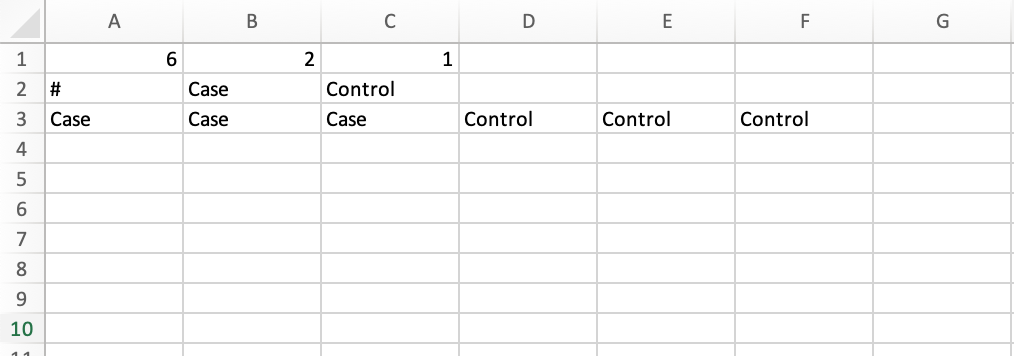
GSEA的输入文件有两个,分别是gct文件和cls文件。
表达谱数据文件格式如下
数据共7列,第一列为基因名,第二至七列为样本表达,分别是三个Case和三个Control

gct文件中包含表达谱数据

cls文件中包含数据比对条件
3. 运行GSEA

最终生成的结果,就是我们Case和Control两组样本相比,差异通路的结果,比如所有通路上调或者下调的情况。

或者单独通路的经典GSEA富集图