DP好题
题目描述
两个基因的相似度的计算方法如下:
对于两个已知基因,例如AGTGATGAGTGATG和GTTAGGTTAG,将它们的碱基互相对应。当然,中间可以加入一些空碱基-,例如:
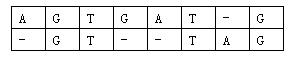
这样,两个基因之间的相似度就可以用碱基之间相似度的总和来描述,碱基之间的相似度如下表所示:
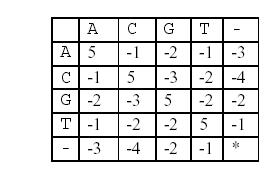
那么相似度就是:(-3)+5+5+(-2)+(-3)+5+(-3)+5=9(−3)+5+5+(−2)+(−3)+5+(−3)+5=9。因为两个基因的对应方法不唯一,例如又有:
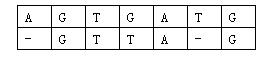
相似度为:(-3)+5+5+(-2)+5+(-1)+5=14(−3)+5+5+(−2)+5+(−1)+5=14。规定两个基因的相似度为所有对应方法中,相似度最大的那个。
输入输出格式
输入格式:
共两行。每行首先是一个整数,表示基因的长度;隔一个空格后是一个基因序列,序列中只含A,C,G,TA,C,G,T四个字母。1 le1≤序列的长度le 100≤100。
输出格式:
仅一行,即输入基因的相似度。
输入输出样例
输入样例#1: 复制
7 AGTGATG 5 GTTAG
输出样例#1: 复制
 View Code
View Code
14
想不出来如何设置状态 果然还是太垃圾
参考大神的做法:
dp[i][j] 代表的是,第一个碱基序列的第i位对应到第二个碱基的第j位的最大值是多少
这样 一共有三种状态转移方式
接下来就是转移方程
7 AGTGATG
5 GTTAG当i=2,j=1时 也就是AG对应G 这个状态可以由这三个状态更新
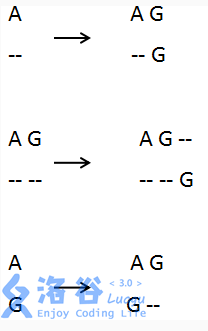
状态更新也就是,直接匹配、第一个碱基序列加一个空格、第二个碱基序列加一个空格,这三个状态。

#include<bits/stdc++.h> using namespace std; //input #define rep(i,x,y) for(int i=(x);i<=(y);++i) #define RI(n) scanf("%d",&(n)) #define RII(n,m) scanf("%d%d",&n,&m); #define RIII(n,m,k) scanf("%d%d%d",&n,&m,&k) #define RS(s) scanf("%s",s) #define LL long long #define REP(i,N) for(int i=0;i<(N);i++) #define CLR(A,v) memset(A,v,sizeof A) ////////////////////////////////// #define N 150 #define inf 0x3f3f3f3f int dp[N][N]; int la,lb,a[N],b[N]; int v[6][6]={ {0,0,0,0,0,0}, {0,5,-1,-2,-1,-3}, {0,-1,5,-3,-2,-4}, {0,-2,-3,5,-2,-2}, {0,-1,-2,-2,5,-1}, {0,-3,-4,-2,-1,0} }; int main() { rep(i,1,N)rep(j,1,N)dp[i][j]=-inf; cin>>la; for(int i=1;i<=la;i++) { char x; cin>>x; if(x=='A') a[i]=1; if(x=='C') a[i]=2; if(x=='G') a[i]=3; if(x=='T') a[i]=4; } cin>>lb; for(int i=1;i<=lb;i++) { char x; cin>>x; if(x=='A') b[i]=1; if(x=='C') b[i]=2; if(x=='G') b[i]=3; if(x=='T') b[i]=4; } for(int i=1;i<=la;i++) dp[i][0]=dp[i-1][0]+v[a[i]][5]; for(int i=1;i<=lb;i++) dp[0][i]=dp[0][i-1]+v[5][b[i]]; for(int i=1;i<=la;i++) { for(int j=1;j<=lb;j++) { dp[i][j]=max(dp[i][j],dp[i-1][j]+v[a[i]][5]);//第一个碱基序列加空格 dp[i][j]=max(dp[i][j],dp[i][j-1]+v[5][b[j]]);//第二个碱基序列加空格 dp[i][j]=max(dp[i][j],dp[i-1][j-1]+v[a[i]][b[j]]);//直接匹配 } } cout<<dp[la][lb]; return 0; }
