作业要求:
实现这个功能的软件也很多,还是烦请大家先自己搜索几个教程,入门请统一用htseq-count,对每个样本都会输出一个表达量文件。
需要用脚本合并所有的样本为表达矩阵。参考:生信编程直播第四题:多个同样的行列式文件合并起来
对这个表达矩阵可以自己简单在excel或者R里面摸索,求平均值,方差。
看看一些生物学意义特殊的基因表现如何,比如GAPDH,β-ACTIN等等。
【1】安装计数软件:htseq-count
1 # conda安装 2 $ conda install -c bioconda htseq 3 4 # 测试 5 # 能够在Python中导入HTSeq这个包,说明安装成功 6 $ python 7 >>> import HTSeq 8 >>>
【2】用htseq对排序后的bam文件进行计数
1 # 用htseq对排序后的bam文件进行计数 2 # htseq-count [options] <alignment_file> <gtf_file> 3 for i in `seq 56 58` 4 do 5 htseq-count -s no -r pos -f bam RNA-Seq/aligned/SRR35899${i}_sorted.bam reference/gencode.v26lift37.annotation.sorted.gtf > RNA-Seq/matrix/SRR35899${i}.count 2> RNA-Seq/matrix/SRR35899${i}.log 6 done 7 8 # (实际情况:没能对gtf文件进行排序,所以使用未排序的bam文件与原始gtf文件) 9 for i in `seq 56 58` 10 do 11 htseq-count -s no -r pos -f bam hisat2_result/SRR35899${i}.bam reference/genome/gencode/gencode.v26lift37.annotation.gtf > htseq-count_result/SRR35899${i}.count 2>log_SRR35899${i}_htseq 12 done 13 -f bam/sam: 指定输入文件格式,默认SAM 14 -r name/pos: 你需要利用samtool sort对数据根据read name或者位置进行排序,默认是name 15 -s yes/no/reverse: 数据是否来自于strand-specific assay。DNA是双链的,所以需要判断到底来自于哪条链。如果选择了no, 那么每一条read都会跟正义链和反义链进行比较。默认的yes对于双端测序表示第一个read都在同一个链上,第二个read则在另一条链上。
程序运行结束后得到矩阵文件:XXXXX.count
【3】合并表达矩阵
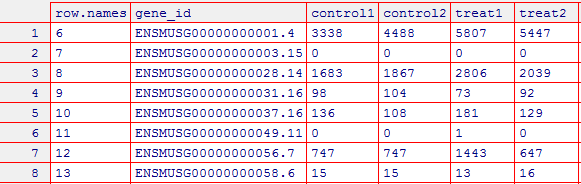
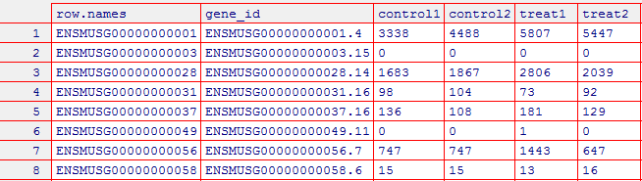
1 # R脚本 2 # 1. 导入数据 3 # 首先将四个文件分别赋值:control1,control2,rep1,rep2 4 >control1 <- read.table("~/disk2/data/rna-seq/matrix/SRR3589959.count", sep=" ", col.names = c("gene_id","control1")) 5 >control2 <- read.table("~/disk2/data/rna-seq/matrix/SRR3589961.count", sep=" ", col.names = c("gene_id","control2")) 6 >rep1 <- read.table("~/disk2/data/rna-seq/matrix/SRR3589960.count", sep=" ", col.names = c("gene_id","treat1")) 7 >rep2 <- read.table("~/disk2/data/rna-seq/matrix/SRR3589962.count", sep=" ",col.names = c("gene_id","treat2")) 8 # 2. 数据整合 9 # 将四个矩阵按照gene_id进行合并,并赋值给raw_count 10 >raw_count <- merge(merge(control1, contol2, by="gene_id"), merge(rep1,rep2, by="gene_id")) 11 # 去掉前五行 12 >raw_count_filter <- raw_count[-1:-5, ] 13 # 因为我们无法在EBI数据库上直接搜索找到ENSMUSG00000024045.5这样的基因,只能是ENSMUSG00000024045的整数,没有小数点,所以需要进一步替换为整数的形式。 14 # 第一步将匹配到的.以及后面的数字连续匹配并替换为空,并赋值给ENSEMBL 15 >ENSEMBL <- gsub("\.\d*", "", raw_count_filter$gene_id) 16 # 将ENSEMBL重新添加到raw_count_filt1矩阵 17 > row.names(raw_count_filter) <- ENSEMBL 18 # 3. 查看数据整体情况 19 > summary(raw_count_filter) 20 # 4. 将raw_count_filter写入csv文件(可用Excel文件) 21 > write.csv(raw_count_filter,"d:/cs_file/r_file/file-lyx0623/H_N_rawcount.csv",row.names=FALSE)

理论知识
关于比对:
】想知道已知基因的表达情况:选择alignment-free工具(例如salmon, sailfish)
】想找noval isoforms:alignment-based工具(如HISAT2, STAR)
】转录本定量:需要考虑更多的因素
1、基因表达定量有3个水平:基因水平、转录本水平、外显子使用水平

基因水平:
Htseq-count等工具作用:根据read和基因位置的overlap,判断该read属于哪个基因
转录本水平:
Cufflinks等工具处理的难题:转录本亚型(isoforms)之间通常是有重叠的,当二代测序读长低于转录本长度时,如何进行区分?这些工具大多采用的都是expectation maximization(EM)。好在我们有三代测序。
外显子使用水平:与基因水平类似。为了更好的计数,需要提供无重叠的外显子区域的gtf文件,用于分析差异外显子使用的DEXSeq提供了一个Python脚本(dexseq_prepare_annotation.py)执行这个任务。
2、reads计数后得到的基因定量结果(count matrix),在进行不同维度的比较时需要进行不同的处理(标准化):
【】比较同一个样本(within-sample)不同基因之间的表达情况---主要考虑转录本长度的影响
(因为转录本越长,那么检测的片段也会更多,直接比较等于让小孩和大人进行赛跑)
【】比较不同样本(across-sample)同一个基因的表达情况---主要考虑测序深度的影响
(测序深度越高,检测到的概率越大)
标准化的算法:RPKM(SE), FPKM(PE),TPM, TMM,RSEM等等。
一般,标准化之后才能进行差异表达分析(某些软件要求原始数据,未经标准化)
htseq参数说明
用法:htseq-count [options] alignment_file gff_file
alignment_file:比对结果文件,可以是sam格式或bam格式,可以通过后面介绍的-f参数指定,默认是sam格式。推荐使用按照name排序后的sam文件,这样的好处是消耗的内存更少,效率更高。关于输入文件排序详见参数-r。
gff_file: 包含单位信息的gff/GTF文件(gff文件格式),大多数情况下就是指注释文件; 由于GTF文件其实就是gff文件格式的变形,在这里同样可以传入GTF格式文件。
-f bam/sam: 指定输入文件格式,默认SAM
-r name/pos: 你需要利用samtool sort对数据根据read name或者位置进行排序,默认是name
-s yes/no/reverse: 数据是否来自于strand-specific assay。DNA是双链的,所以需要判断到底来自于哪条链。如果选择了no, 那么每一条read都会跟正义链和反义链进行比较。默认的yes对于双端测序表示第一个read都在同一个链上,第二个read则在另一条链上。
-a 最低质量, 剔除低于阈值的read
-m 模式 union(默认), intersection-strict and intersection-nonempty。一般而言就用默认的,作者也是这样认为的。
-i id attribute: 在GTF文件的最后一栏里,会有这个基因的多个命名方式(如下), RNA-Seq数据分析常用的是gene_id, 当然你可以写一个脚本替换成其他命名方式。