今天博主博士答辩完毕啦,超开心的,撒花~
虽然很疲惫(昨晚太晚睡了==),但想到今天跟师妹说给她gene-based关联分析的方法。
于是,决定还是整理好了再休息休息。
好,进入主题。
之前的推文我曾经写过使用VEGAS2(Versatile Gene-based Association Study)进行gene based的关联分析研究。
但用过的人就知道,VEGAS2有个很明显的缺点,跑起来很占CPU,因此今天再重新推荐另一个工具GCTA,也是可以计算gene-based关联分析的。
1、下载、安装
首先,下载GCTA:
wget https://cnsgenomics.com/software/gcta/bin/gcta_1.92.4beta2.zip
解压:
unzip gcta_1.92.4beta2.zip
cd gcta_1.92.4beta2
2、准备输入文件
准备summary_statistics的输入文件GWASp,输入文件GWASp包括两列,第一列是SNP的ID,第二列是SNP的P值(这里显示的是P-value)。

准备基因型文件1000G_EUR,基因型文件可以是公共数据库的,比如千人基因组的。值得注意的是,你拿到的数据是什么人群的,基因型文件就选用相应的人群数据,比如你的数据是欧洲祖先,那么基因型的话就选用千人基因组的欧洲祖先人群,而不是所有样本(两千多个)。
基因型文件1000G_EUR为PLINK格式(bed,bim,fam或者map,ped),PLINK格式还不了解?见推文GWAS分析基本流程及分析思路
准备glist-hg19.txt文件,格式如下所示:
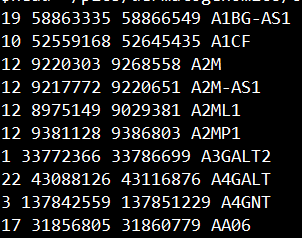
glist-hg19.txt文件出自GCTA网站,各位找不到的话在后台发送gli关键词即可获得该文件。
3、gene-based关联分析
准备好GWASp、1000G_EUR和glist-hg19.txt后,输入如下命令:
gcta64 --bfile 1000G_EUR --maf 0.01 --fastBAT GWASp --fastBAT-gene-list glist-hg19.txt --out GWASp_result --thread-num 10
4、结果解读
完成以上分析后,会得到GWASp_result.gene.fastbat的结果文件,其示例如下所示:
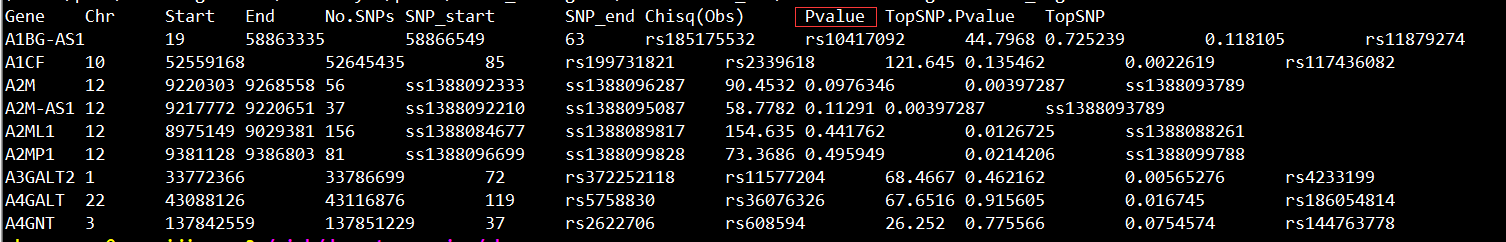
其中,圈出来的红框即为我们感兴趣的基因与表型的相关性P值。显著阈值的设定为:0.05/(GWASp_result.gene.fastbat文件行数-1)
今天的介绍就到这,明天有时间的话再推一篇超实用的工具!
祝各位周一愉快!
