Sun C, Dong Z, Zhao L, et al. The Wheat 660K SNP array demonstrates great potential for marker‐assisted selection in polyploid wheat[J]. Plant Biotechnology Journal, 2020, 18(6): 1354-1360.
作者将小麦660K芯片同35K、55K、90K和820K等芯片进行了比较,从SNP数量、分布、密度、检测到的SNP同基因的关联、杂合性与应用的角度进行了评价。结果表明,660K芯片具有良好的性价比和可靠性,或将是小麦遗传改良中靶向基因分型和标记辅助选择的最佳选择。
1. 标记辅助选择的发展
MAS(marker‐assisted selection)在小麦全基因组水平上加速了小麦的育种过程。其允许早期就对目标性状进行选择。下图展示了基因分型平台及其在标记辅助选择中的应用。
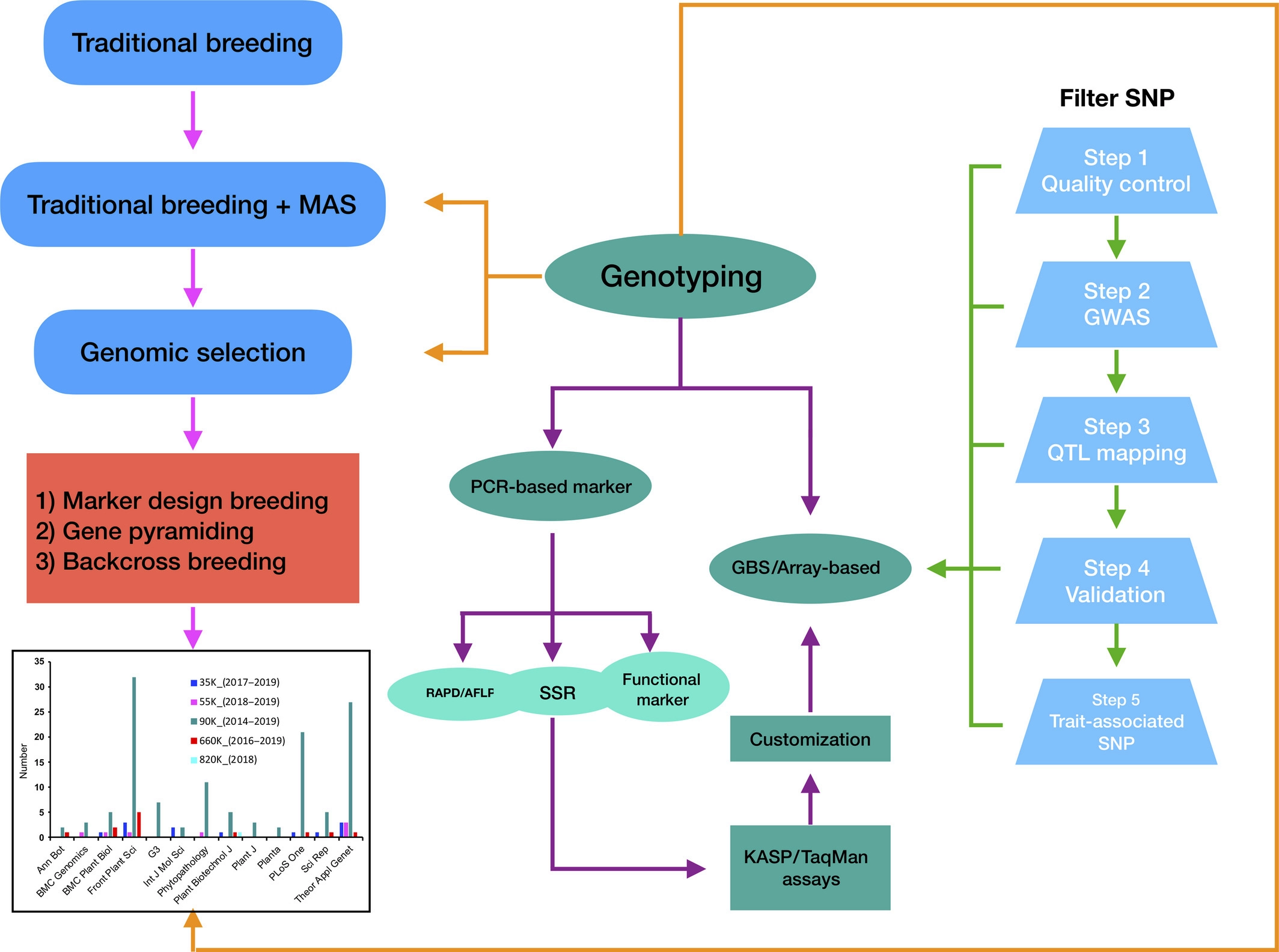
其中,“传统”的、基于PCR的分子标记具有一些缺点,很大程度表现在吞吐量和密度较低。比如,SSR motifs在基因组中是有限的,并且分布不均匀。另外,许多小麦的性状通常受多个基因的影响,导致功能标记对次要基因(minor genes)的检测效果不佳。
NGS提供的测序信息则能够极大提高覆盖范围,降低时间和成本消耗。迄今为止,已有超过15种GBS技术在作物中得到应用。其过程通常包括限制性酶切,衔接子连接,PCR扩增和测序(restriction‐enzyme digestion, adapter ligation, PCR amplification and sequencing)。然而,GBS很容易因低读段覆盖率和检测真正纯合子的能力差(the poor ability to detect true homozygotes)而导致错误,这在异种或早期种群中尤为明显。同时,GBS在建库方面也是一种劳动密集型工作。而且,GBS的数据分析和存储也较为复杂。此外,GBS的性能在很大程度上取决于参考基因组的质量。与现代品种相比,用于建立参考基因组的中国春是具有较大基因组变异的地方品种,导致GBS揭示的标记物覆盖率低,从而限制了GBS在当前小麦品种中的应用。
GBS(Genotyping-by-Sequencing)是一种常见的简化基因组测序技术,其原理是将基因组DNA进行酶切,然后对酶切片段两端序列进行高通量测序。通过调整所需捕获酶切位点的片段数(Tags),可以控制捕获序列的范围。
SNP芯片则在样品与数据点编号的定制上十分灵活。迄今为止,已有多种高密度SNP芯片被设计出来,包括Illumina Wheat 9K iSelect SNP array[1]、Illumina Wheat 90K iSelect SNP genotyping array[2]、Wheat 15K SNP array[3]、Axiom® Wheat 660K SNP array、Wheat 55K SNP array、Axiom® HD Wheat genotyping (820K) array[4]、Wheat Breeders’ 35K Axiom array[5]、Wheat 50K Triticum TraitBreed array[6]等。下表展示了作者整理的一些芯片的信息。

其中,小麦9K和90K阵列中的SNP在检测地方品种、合成小麦和源自二级和三级基因库的种质中的多态性无效。为解决这一问题,Winfield et al. 通过外显子捕获43种小麦和野生种的材料开发出了小麦820K阵列以面向世界上的各种品系。然而,小麦820K SNP芯片上的大多数标记均基于小麦及其亲缘种,从而导致该芯片对于针对六倍体种质特别感兴趣的育种者而言价值有限。35K芯片源自820K芯片,是一种低通量芯片,可提供面向育种者的信息标记。660K芯片是由中国农业科学院设计的,基于六倍体和四倍体小麦、Emmer小麦和Taegchii Aegilops,具有基因组特异性、高密度、经济高效和泛用性的芯片,并且还为D基因组添加了许多标记。660K芯片已被广泛用于鉴定遗传基因,以鉴定普通小麦的品质性状、农艺性状和抗病性。
与基于GBS和PCR的标记相比,基于芯片阵列的基因分型平台已经实现了高度商业化,并且可以灵活地自定义样品和探针位点的数量。由于没有因NGS读段覆盖率低和多倍性而导致的基因分型错误的缺点,基于阵列的平台的SNP检出率很高且稳定,但在农作物野生亲缘种和地方种的SNP检测中也有着明显的偏好(ascertainment bias)。(可以通过对具有广泛遗传背景的样品进行阵列定制来改善此问题)在对大量SNP和样本的基因分型中,基于芯片的平台在每个数据点上性价比明显。但是相较于基于GBS和PCR的标记进行基因分型,芯片法在对单个样本的基因分型的总成本上并无优势。
基于芯片的分型平台,在操作和下游数据分析上更为容易,这对非专业技术人员来说很有吸引力。
2. 七个流行的小麦芯片的比较
作者选取了七个广为使用的小麦芯片进行比较:小麦9K,15K,35K,55K,90K,820K和660K阵列。
The Wheat 9K and 15K SNP arrays, Wheat 35K SNP array and Wheat 55K SNP array are derived from the Wheat 90K SNP array, Wheat 820K SNP array and Wheat 660K SNP array, respectively. 受当时的测序和装配技术的限制,小麦90K SNP基因分型阵列中SNP的物理位置不准确,并且小麦820K和660K SNP阵列中的非特异性探针位点无法相互比较。因此,作者更新了三个高密度SNP阵列Wheat 90K,820K和660K SNP的物理位置。
2.1 SNP数量分布
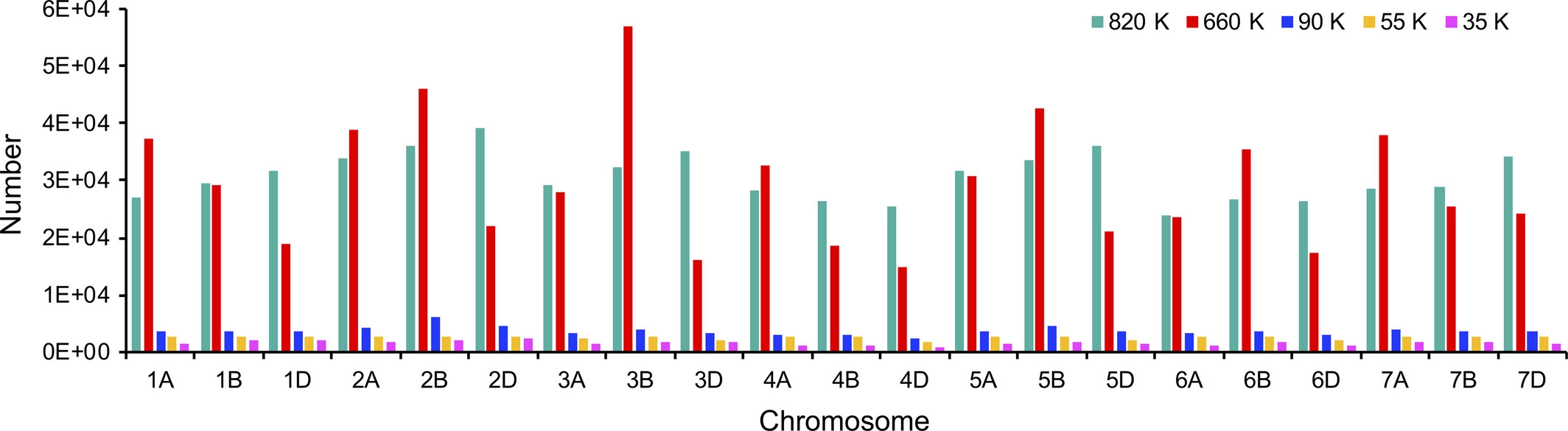
下图展示了5种芯片上具有独特可靠的物理位置的SNP数量在染色体上的分布。在小麦660K SNP阵列中,3B染色体上的SNP数量最多;而在小麦35K,90K和820K SNP阵列中,2D染色体上SNP最多。小麦55K SNP阵列中的SNP几乎均匀地分布在所有染色体上。
2.2 SNP密度分布
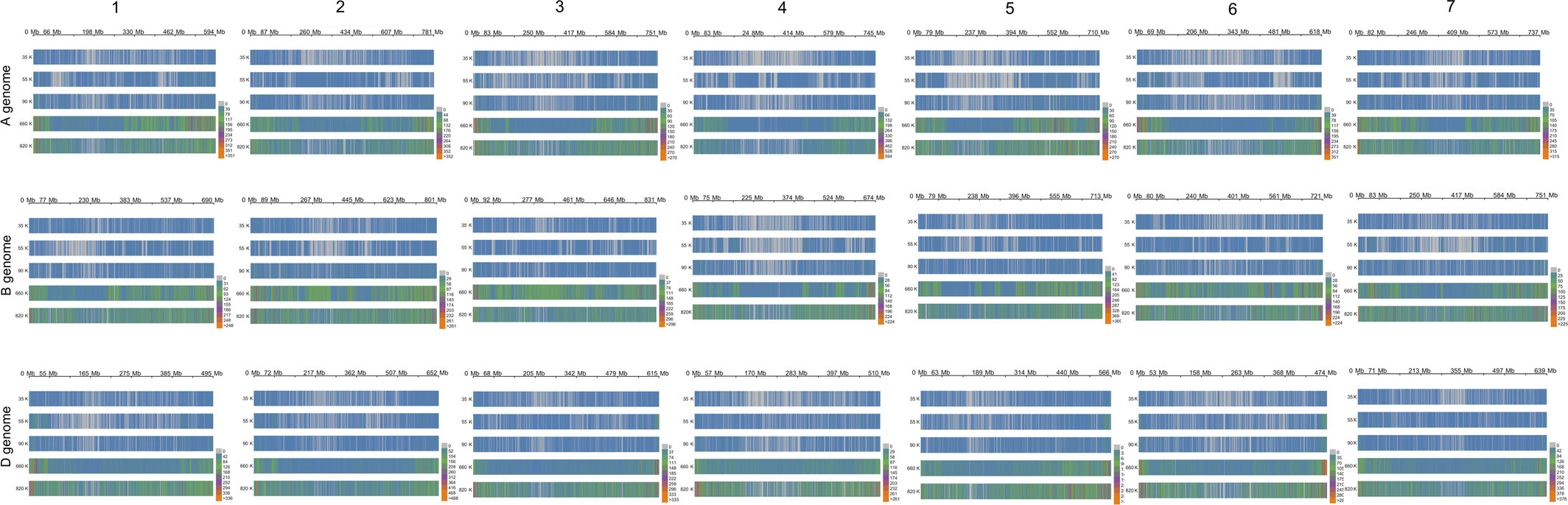
在SNP密度分布的测定中,作者将染色体分为1Mb的窗口进行计算。Wheat 660K SNP阵列中几乎所有bin都包含SNP,而其他四个阵列则检测到许多没有SNP(灰色)的bin(尤其是Wheat 35K,55K和90K SNP阵列)。与Wheat 820K SNP阵列相比,在Wheat 660K SNP阵列的染色体顶部或末端发现了更多的SNP,这是因为在这些区域发生了更多的重组事件,而在着丝粒区域发生的重组较少。Figure S1展示了SNP密度分布。下图展示了SNP数量的热图。
2.3 有注释信息的基因SNP数量对比
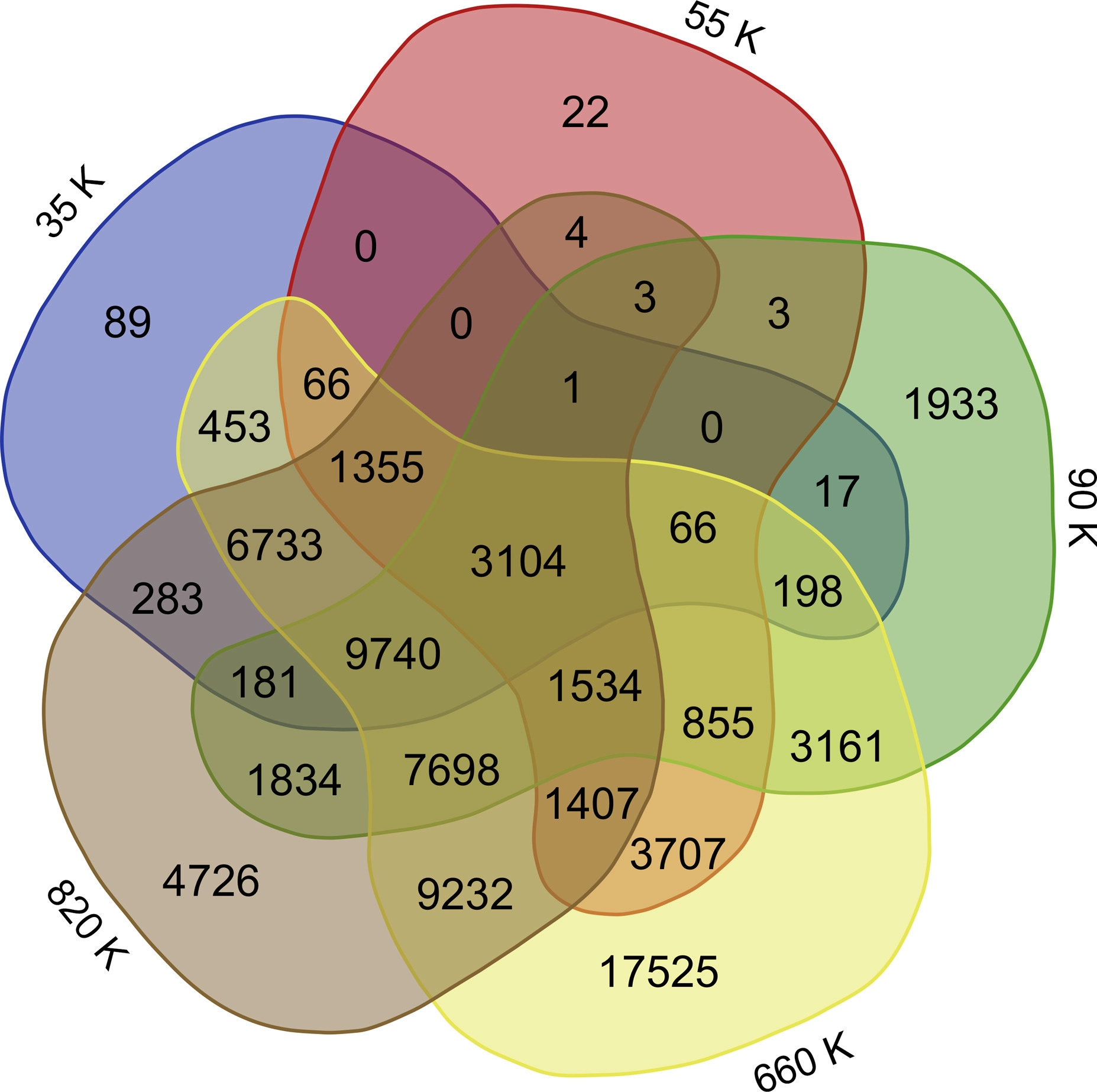
作者接下来统计了检测到的SNP中,位于有注释(IWGSC RefSeq v1.1)的基因或启动子区域的SNP,并分别在小麦35K,55K,90K,660K和820K SNP芯片的注释基因中找到了26236、13191、64643、229266和538079个SNP。小麦820K SNP阵列检测到的基因SNP数量最多(538079),但是这些基因SNP仅位于47835(45.47%)个不同的注释基因(唯一SNP基因)中。660K芯片在66834个(63.52%)不同的注释基因中命中了229266个基因SNP,表明Wheat 660K SNP芯片通过设计数量较少的独特探针位点而比820K芯片整合了更多的注释基因。此外,小麦90K SNP阵列也表现出良好的性能,因为64643个基因SNP与30328个(28.83%)带注释的基因相关。
下图展示了6个芯片之间,测到的注释基因的重叠数量关系。
2.4 测得的品种杂合度
在芯片杂合度的测定上,作者通过不同的小麦SNP芯片对由中国普通小麦组成的三个分组进行了基因分型。Panel_I包含163个中国品种,以小麦90K SNP芯片进行检测。分析表明以小麦90K SNP芯片测定的Panel_I的品种的杂合度为28.67%。Panel_II和Panel_III分别包含243种中国小麦种质和435个高产种系Yanzhan1的基因渗入系,用Wheat 660K SNP阵列进行基因分型。分析表明660K测定的两个分组,平均杂合度为5.5%(2.86% for Panel_II and 8.14% for Panel_III)。有文章[4:1]提到了820K芯片测定的475个小麦品种,杂合率为4.7%。因此,作者认为通过660K芯片和820K芯片测得的小麦品种具有较低的杂合度。
2.5 应用情况
自2014年以来,共检测到171篇相关研究文章,这些文章采用了上述小麦阵列。这些研究主要发表在Theoretical Applied Genetics, PLoS One, Frontiers in Plant Science, Phytopathology, Plant Biotechnology Journal, Scientific Report 和 BMC Plant Biology 上。在与小麦阵列有关的所有论文中(图1),80%使用小麦90K阵列,表明小麦90K阵列在过去五年中最广泛用于鉴定与性状相关的基因位点。在2017年正式使用Wheat 660K阵列后[7],在接下来的两年中发表了12篇使用660K阵列的论文。基于SNP密度,杂合性和染色体上的分布,小麦660K阵列在MAS中具有巨大的应用潜力,可能是在小麦育种计划中鉴定与性状相关的基因座和MAS的最佳选择。
部分其他参考文献
Cavanagh, C.R., Chao, S., Wang, S., Huang, B.E., Stephen, S., Kiani, S., Forrest, K. et al . (2013) Genome‐wide comparative diversity uncovers multiple targets of selection for improvement in hexaploid wheat landraces and cultivars. Proc. Natl Acad. Sci. USA, 110, 8057–8062. ↩︎
Wang, S., Wong, D., Forrest, K., Allen, A., Chao, S., Huang, B.E., Maccaferri, M. et al . (2014) Characterization of polyploid wheat genomic diversity using a high‐density 90000 single nucleotide polymorphism array. Plant Biotechnol. J. 12, 787–796. ↩︎
Boeven, P.H., Longin, C.F.H., Leiser, W.L., Kollers, S., Ebmeyer, E. and Würschum, T. (2016) Genetic architecture of male floral traits required for hybrid wheat breeding. Theor. Appl. Genet. 129, 2343–2357. ↩︎
Winfield, M.O., Allen, A.M., Burridge, A.J., Barker, G.L., Benbow, H.R., Wilkinson, P.A., Coghill, J. et al . (2016) High‐density SNP genotyping array for hexaploid wheat and its secondary and tertiary gene pool. Plant Biotechnol. J. 14, 1195–1206. ↩︎ ↩︎
Allen, A.M., Winfield, M.O., Burridge, A.J., Downie, R.C., Benbow, H.R., Barker, G.L., Wilkinson, P.A. et al . (2017) Characterization of a Wheat Breeders’ Array suitable for high‐throughput SNP genotyping of global accessions of hexaploid bread wheat (Triticum aestivum ). Plant Biotechnol. J. 15, 390–401. ↩︎
Rasheed, A. and Xia, X. (2019) From markers to genome‐based breeding in wheat. Theor. Appl. Genet. 132, 767–784. ↩︎
Cui, F., Zhang, N., Fan, X.L., Zhang, W., Zhao, C.H., Yang, L.J., Pan, R.Q. et al . (2017) Utilization of a Wheat 660K SNP array‐derived high‐density genetic map for high‐resolution mapping of a major QTL for kernel number. Sci. Rep. 7, 3788. ↩︎