准备
相关的库
相关的库包括:
- numpy
- pandas
- sklearn
带入代码如下:
import pandas as pd
import numpy as np
from sklearn.neighbors import KNeighborsClassfier as KNN
数据准备
数据是sklearn的乳腺癌数据。
from skleanr.datasets import load_breast_cancer
data=load_breast_caner()
data主要分为两部分:data和target,把这两部分,设置变量导入DataFrame中可查看基本形状。
X = data.data
y = data.target
sklearn的数据其形式比较固定,data的主要属性有:
data。数据,即变量的值,多行多列target。目标,即因变量的值,一般是一行DESCR。描述,可打印出,描述变量、目标features_names。X的列名target_names。Y的列名filename。数据文件所在位置(一般在libsite-packagessklearndatasetsdata目录下)
分数据集和测试集:
from sklearn.model_selection import train_test_split
Xtrain,Xtest,Ytrain,Ytest=train_test_split(X, y, test_size=0.3)
注意:
- 0.3是指30%数据作为测试。每次运行不同,可通过
random_state控制 - 返回的结果固定,不可错
建立模型
clf = KNN(n_neighbors = 5)
clf=clf.fit(Xtrain,Ytrain)
clf就是训练好的模型,可调用接口查看进行预测和评分。常用是predict、score和kneighbors。三者分别用来预测、评分、求最近邻。
在选择训练集和测试集的时候,可能会存在以下问题。
- 选择测试集和训练集每次都是不同的,因此每次模型的效果都不同。
- 选择测试集和训练集有时会极大影响模型。——特别是当数据是有顺序的时候。
因此需要交叉验证,找到最好的参数,再次训练模型。
K折交叉验证
K折交叉验证的方法:
cvresult=CVS(clf,X,y,cv=5)
CVS的第一个参数是训练过的模型,参数cv是折数。
cvresult.mean() # 取得均值
cvresult.var() #取得方差
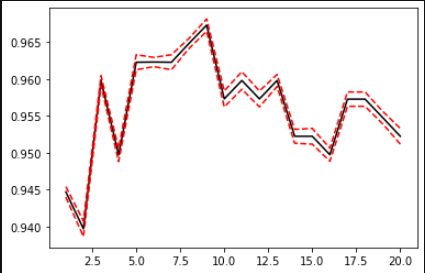
可利用方差,绘制出学习曲线:
score =[]
var_=[]
krange=range(1,21)
for i in krange:
clf=KNN(n_neighbors=i)
cvresult=CVS(clf,X,y,cv=5)
score.append(cvresult.mean())
var_.append(cvresult.var())
plt.plot(krange,score,color='k')
plt.plot(krange,np.array(score)+np.array(var_)*2,c='red',linestyle='--')
plt.plot(krange,np.array(score)-np.array(var_)*2,c='red',linestyle='--')
bestindex=score.index(max(score))
print(bestindex+1)
print(score[bestindex])
常用交叉验证
- K折。特别在回归模型,若数据有顺序,结果会很糟糕
- stratifiedKfold。常用
- shuffleSplit。常用
- GroupKFold。
但是如果把数据分为:训练数据、测试数据。训练数据又分出来一部分验证数据,那么真正用于训练的数据就更小了。
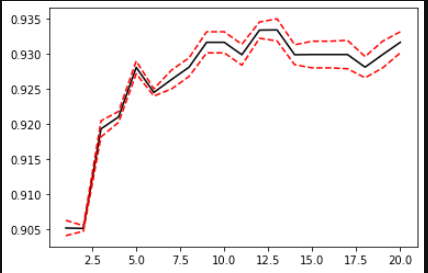
归一化
KNN是距离类的模型,因此需要归一化。也就是把数据减去最差值,处以极差:
[x^*=frac{x-min(x)}{max(x)-min(x)}
]
归一化要分训练集和测试集之后。(因为归一化时候用到的极值,很可能就是测试集的数据,这样事先就把数据透露给模型了)
Xtrain,Xtest,Ytrain,Ytest=train_test_split(X_,y,
test_size=0.3,
random_state=420)
MMS=nms().fit(Xtrain) #MMS中,有Xtrain的min,和极差
Xtest_=MMS.transform(Xtest)
Xtrain_=MMS.transform(Xtrain) #分别对训练集、测试集进行归一化
这样再运行学习曲线的代码,得到的结果就要好一些: