原文链接:http://tecdat.cn/?p=4764
介绍
芯片数据分析流程有些复杂,但使用 R 和 Bioconductor 包进行分析就简单多了。本教程将一步一步的展示如何安装 R 和 Bioconductor,通过 GEO 数据库下载芯片数据, 对数据进行标准化,然后对数据进行质控检查,最后查找差异表达的基因。教程示例安装的各种依赖包和运行命令均是是在 Ubuntu 环境中运行的(版本: Ubuntu 10.04, R 2.121),教程的示例代码和图片在这里。
安装 R 和 Bioconductor 包
打开命令终端,先安装 R 和 Bioconductor 的依赖包,然后安装 R.
之后在 R 环境中安装 Bioconductor 包
> # 下载 Bioconductor 的安装程序> source("http://bioconductor.org/biocLite.R")> # 安装 Bioconductor 的核心包> biocLite()> # 安装 GEO 包> biocLite("GEOquery")
如果你没有管理员权限,你需要将这些包安装到你个人库目录中。安装 Bioconductor 需要一段时间,GEOquery 包也需要安装,GEOquery 是 NCBI 存储标准化的转录组数据的基因表达综合数据库 GEO 的接口程序。
下载芯片数据
本教程中我们使用 Dr Andrew Browning 发表的数据集 GSE20986。HUVECs(人脐静脉内皮细胞)是从人胎儿脐带血中提取出来的,通常用来研究内皮细胞的病理生理机制。这个实验设计中,从捐献者的眼组织中提取的虹膜、视网膜、脉络膜微血管内皮细胞用来和 HUVEC 细胞进行比对,以便考查 HUVEC 细胞能否替代其他细胞作为研究眼科疾病的样本。 GEO 页面同时也包含了关于本实验研究的其他信息。对于每组样本有三次测量,样品分成四组 iris, retina, HUVEC, choroidal 。
实验平台是 GPL570 -- GEO 数据库对人类转录组芯片 Affymetrix Human Genome U133 Plus 2.0 Array 的缩写。通过 GSM 链接 GSM524662,我们可以得到各个芯片的更多实验条件信息。同一个实验设计中的不同芯片的实验条件应该是相同的。不同细胞系的提取条件,细胞生长条件,RNA 提取程序,RNA 处理程序,RNA 与探针杂交条件,用哪种仪器扫描芯片,这些数据都以符合 MIAME 标准的形式存储着。
对于每一个芯片,数据表中存储着探针组和对应探针组标准化之后的基因表达量值。表头中提供了表达量值标准化所用的算法。本教程中使用 GC-RMA 算法进行数据标准化,关于 GC-RMA 算法的详细细节可以参考这篇文献。
首先,我们从 GEO 数据库下载原始数据,导入 GEOquery 包,用它下载原始数据,下载的原始数据约 53MB。
> library(GEOquery)> getGEOSuppFiles("GSE20986")下载完成后,打开文件管理器,在启动 R 程序的文件夹里可以看到当前文件夹下生成了一个 GSE20986 文件夹,可以直接查看文件夹里的内容:
$ ls GSE20986/filelist.txt GSE20986_RAW.tar
GSE20986_RAW.tar 文件是压缩打包的 CEL(Affymetrix array 数据原始格式)文件。先解包数据,再解压数据。这些操作可以直接在 R 命令终端中进行:
> untar("GSE20986/GSE20986_RAW.tar", exdir="data")> cels <- pattern="[gz]"> sapply(paste("data", cels, sep="/"), gunzip)> cels
芯片实验信息整理
在对数据进行分析之前,我们需要先整理好实验设计信息。这其实就是一个文本文件,包含芯片名字、此芯片上杂交的样本名字。为了方便在 R 中 使用 simpleaffy 包读取实验信息文本文件,需要先编辑好格式:
$ ls -1 data/*.CEL > data/phenodata.txt将这个文本文件用编辑器打开,现在其中只有一列 CEL 文件名,最终的实验信息文本需要包含三列数据(用 tab 分隔),分别是 Name, FileName, Target。本教程中 Name 和 FileName 这两栏是相同的,当然 Name 这一栏可以用更加容易理解的名字代替。
Target 这一栏数据是芯片上的样本标签,例如 iris, retina, HUVEC, choroidal,这些标签定义了那些数据是生物学重复以便后面的分析。
这个实验信息文本文件最终格式是这样的:
Name FileName TargetGSM524662.CEL GSM524662.CEL irisGSM524663.CEL GSM524663.CEL retinaGSM524664.CEL GSM524664.CEL retinaGSM524665.CEL GSM524665.CEL irisGSM524666.CEL GSM524666.CEL retinaGSM524667.CEL GSM524667.CEL irisGSM524668.CEL GSM524668.CEL choroidGSM524669.CEL GSM524669.CEL choroidGSM524670.CEL GSM524670.CEL choroidGSM524671.CEL GSM524671.CEL huvecGSM524672.CEL GSM524672.CEL huvecGSM524673.CEL GSM524673.CEL huvec注意:每栏之间是使用 Tab 进行分隔的,而不是空格!
载入数据并对其进行标准化
需要先安装 simpleaffy 包,simpleaffy 包提供了处理 CEL 数据的程序,可以对 CEL 数据进行标准化同时导入实验信息(即前一步中整理好的实验信息文本文件内容),导入数据到 R 变量 celfiles 中:
> biocLite("simpleaffy")> library(simpleaffy)> celfiles <- read.affy(covdesc="phenodata.txt", path="data")
你可以通过输入 ‘celfiles’ 来确定数据导入成功并添加芯片注释(第一次输入 ‘celfiles’ 的时候会进行注释):
> celfilesAffyBatch objectsize of arrays=1164x1164 features (12 kb)cdf=HG-U133_Plus_2 (54675 affyids)number of samples=12number of genes=54675annotation=hgu133plus2notes=
现在我们需要对数据进行标准化,使用 GC-RMA 算法对 GEO 数据库中的数据进行标准化,第一次运行的时候需要下载一些其他的必要文件:
> celfiles.gcrma <- gcrma(celfiles)Adjusting for optical effect............Done.Computing affinitiesLoading required package: AnnotationDbi.Done.Adjusting for non-specific binding............Done.NormalizingCalculating Expression
如果你想看标准化之后的数据,输入 celfiles.gcrma, 你会发现提示已经不是 AffyBatch object 了,而是 ExpressionSet object,是已经标准化了的数据:
> celfiles.gcrmaExpressionSet (storageMode: lockedEnvironment)assayData: 54675 features, 12 sampleselement names: exprsprotocolDatasampleNames: GSM524662.CEL GSM524663.CEL ... GSM524673.CEL (12 total)varLabels: ScanDatevarMetadata: labelDeionphenoDatasampleNames: GSM524662.CEL GSM524663.CEL ... GSM524673.CEL (12 total)varLabels: sample FileName TargetvarMetadata: labelDeionfeatureData: noneexperimentData: use 'experimentData(object)'Annotation: hgu133plus2
数据质量控制
再进行下一步的数据分析之前,我们有必要对数据质量进行检查,确保没有其他的问题。首先,可以通过对标准化之前和之后的数据画箱线图来检查 GC-RMA 标准化的效果:
> # 载入色彩包> library(RColorBrewer)> # 设置调色板> cols <-> # 对标准化之前的探针信号强度做箱线图> boxplot(celfiles, col=cols)> # 对标准化之后的探针信号强度做箱线图,需要先安装 affyPLM 包,以便解析 celfiles.gcrma 数据> biocLite("affyPLM")> library(affyPLM)> boxplot(celfiles.gcrma, col=cols)> # 标准化前后的箱线图会有些变化> # 但是密度曲线图看起来更直观一些> # 对标准化之前的数据做密度曲线图> hist(celfiles, col=cols)> # 对标准化之后的数据做密度曲线图> hist(celfiles.gcrma, col=cols)
数据标准化之前的箱线图
标准化之前的箱线图
数据标准化之后的箱线图
标准化之后的箱线图
数据标准化之前的密度曲线图
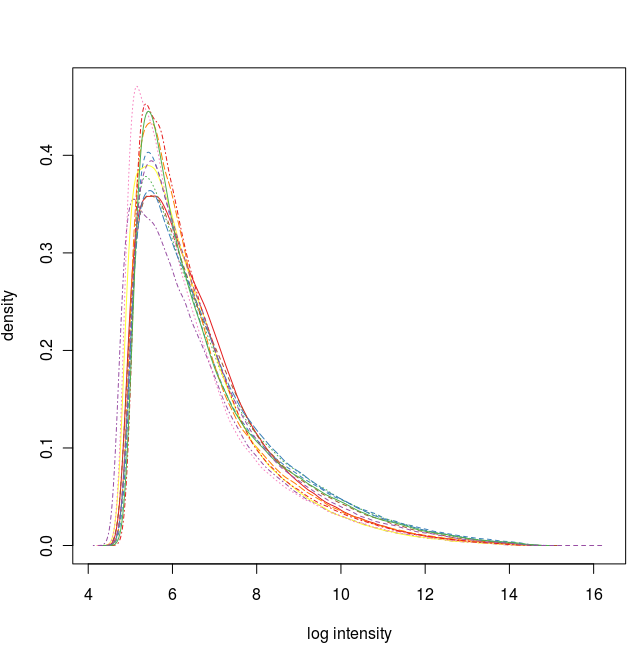
标准化之前的密度曲线图
数据标准化之后的密度曲线图
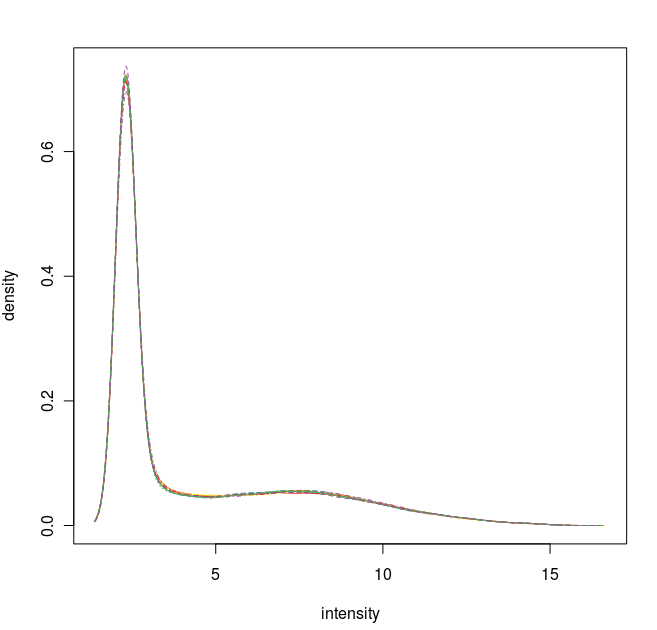
标准化之后的密度曲线图
通过这些图我们可以看出这12张芯片数据之间差异不大,标准化处理将所有芯片信号强度标准化到具有类似分布特征的区间内。为了更详细地了解芯片探针信号强度,我们可以使用 affyPLM 对单个芯片 CEL 数据进行可视化。
> # 从 CEL 文件读取探针信号强度:> celfiles.qc <-> # 对芯片 GSM24662.CEL 信号进行可视化:> image(celfiles.qc, which=1, add.legend=TRUE)> # 对芯片 GSM524665.CEL 进行可视化> # 这张芯片数据有些人为误差> image(celfiles.qc, which=4, add.legend=TRUE)> # affyPLM 包还可以画箱线图> # RLE (Relative Log Expression 相对表达量取对数) 图中> # 所有的值都应该接近于零。 GSM524665.CEL 芯片数据由于有人为误差而例外。> RLE(celfiles.qc, main="RLE")> # 也可以用 NUSE (Normalised Unscaled Standard Errors)作图比较.> # 对于绝大部分基因,标准差的中位数应该是1。> # 芯片 GSM524665.CEL 在这个图中,同样是一个例外> NUSE(celfiles.qc, main="NUSE")
标准的芯片 AffyPLM 信号图

标准的芯片 AffyPLM 信号图
存在人工误差的芯片 AffyPLM 信号图
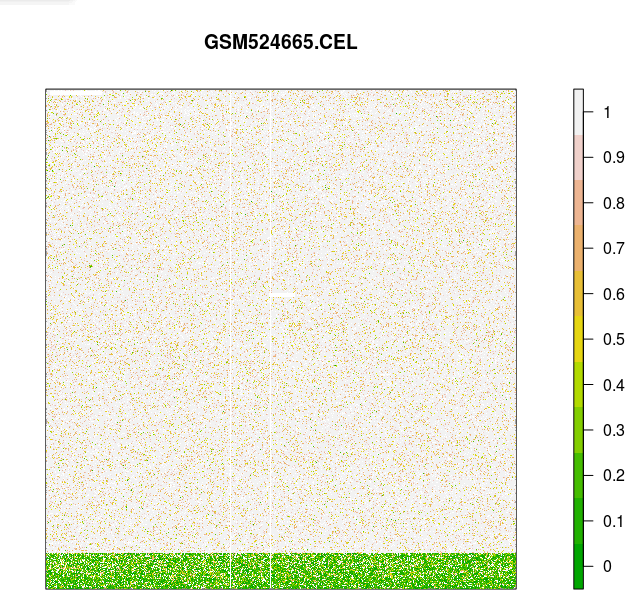
存在人工误差的芯片 AffyPLM 信号图
CEL 数据的 RLE 图

RLE (Relative Log Expression) 图
CEL 数据的 NUSE 图
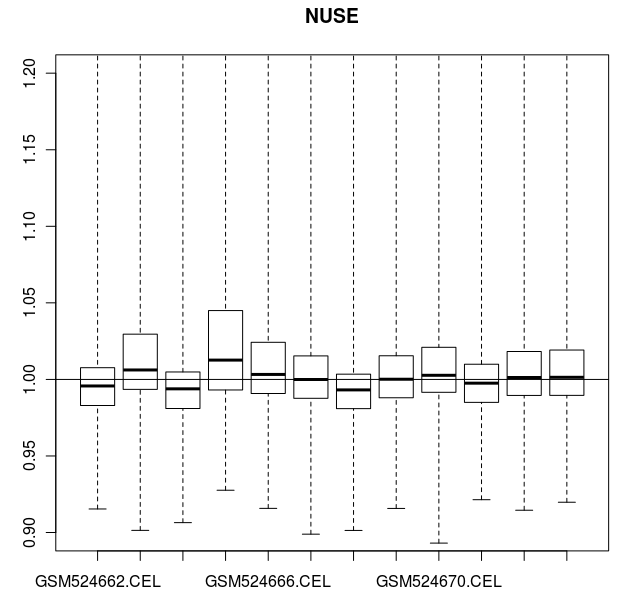
NUSE (Normalised Unscaled Standard Errors) 图
我们还可以通过层次聚类来查看样本之间的关系:
CEL 数据的层次聚类图

层次聚类图
图形显示,与其他眼组织相比 HUVEC 样品是单独的一组,表现出组织类型聚集的一些特征,另外 GSM524665.CEL 数据在此图中并不显示为异常值。
数据过滤
现在我们可以对数据进行分析了,分析的第一步就是要过滤掉数据中的无用数据,例如作为内参的探针数据,基因表达无明显变化的数据(在差异表达统计时也会被过滤掉),信号值与背景信号差不多的探针数据。下面的 nsFilter 参数是为了不删除没有 Entrez Gene ID 的位点,保留有重复 Entrez Gene ID 的位点:
> celfiles.filtered <- require.entrez="FALSE," remove.dupentrez="FALSE)"> # 哪些位点被过滤掉了?为什么?> celfiles.filtered$filter.log$numLowVar[1] 27307$feature.exclude[1] 62
我们可以看出有 27307 个探针位点因为无明显表达差异(LowVar)被过滤掉,有 62 个探针位点因为是内参而被过滤掉。