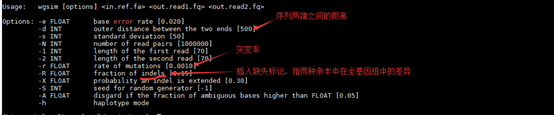
介绍 ============ Wgsim是从参照基因组中模拟序列的小工具。 它能够模拟二倍体基因组与SNP和插入/缺失(INDEL) 多态性,并能够模拟均匀替代测序错误的reads。 它不产生INDEL测序错误,但是这可能是部分地 通过模拟INDEL多态性补偿。 Wgsim输出是模拟多态性,并写入真正的reads坐标 以及在reads名称的多态性和测序错误的数量。 我们可以wgsim_eval.pl自带的包评估映射的准确性或SNP caller。 编译 =========== GCC -g -O2 -Wall -o wgsim wgsim.c -lz -lm 评估 ========== 仿真与评估 ------------------------- 仿真命令行: wgsim -Nxxx -1yyy -d0 -S11 -e0 -rzzz hs37m.fa YYY-zzz.fq的/ dev / null的 其中yyy是read长,zzz为错误率和$ XXX * $ YYY =10000000。 默认情况下,多态性的15%以上是插入缺失和它们的长度是从绘制 几何分布密度0.7 * 0.3 ^ {L-1}。 评估命令行: wgsim_eval.pl独特aln.sam | wgsim_eval.pl alneval -g 20 在'-g'选项可以和映射器来改变。 系统 ------
GCC: 4.1.2 CPU: AMD Opteron 8350 @ 2.0GHz Mem: 128GB
实际操作:
使用过的命令:
../seqtk_concat out_1.fq out_2.fq | head -n 8
../seqtk_concat out_1.fq out_2.fq > ../wgsim.fq #双端序列合并
seqtk_names wgsim.fq | cut -f1,2 -d'_'| cut -f3 -d'|' | perl -ane 's/chrS+/9606/; print' | perl -ne 'print qq{$. $_}' | tabtk_bins - 1 >tabtk_stats
seqtk_names wgsim.fq | cut -f1,2 -d'_'| cut -f3 -d'|' | perl -ane 's/chrS+/9606/; print' | perl -ne 'print qq{$. $_}' | tabtk_bins - 1 >tabtk_stats.tsv
cut -f1,2 tabtk_stats.tsv | tabtk_decorate /biostack/database/taxonomy/node-name.tsv 0 - > tab.xls
/*
解释:
perl -ane 's/chrS+/9606/; print' 将每一行中包含chr并且其后非空格部分(比如chrab和chrbc)替换为9606
perl -ne 'print qq{$. $_}' 打印每一行的行号和内容;
perl -ane 's/chrS+/9606/; print' 将每一行中包含chr并且其后非空格部分(比如chrab和chrbc)替换为9606
perl -ne 'print qq{$. $_}' 打印每一行的行号和内容;
可简化为:
|perl -ane 's/chrS+/9606/; print qq{$. $_}' 或 perl -ane 's/chrS+/9606/; print qq{$. $_}'
*/
cut说明:
cut 不就是『切』吗?没错啦!这个指令可以将一段讯息的某一段给他『切』出来~ 处理的讯息是以
『行』为单位喔!
常用的参数:

