Sansaloni, C., Franco, J., Santos, B. et al. Diversity analysis of 80,000 wheat accessions reveals consequences and opportunities of selection footprints. Nat Commun 11, 4572 (2020). https://doi.org/10.1038/s41467-020-18404-w
本文作者利用DArTseq对56342种驯化六倍体、18946种驯化四倍体,以及3903种野生近缘种进行了测序,鉴定出了三十余万个高质量SNP和SilicoDArT marker。测序结果被比对到IWGSC RefSeq v1.0、durum wheat genome assembly (cv. Svevo)和DArT genetic map。研究中的小麦来自CIMMYT和ICARDA。
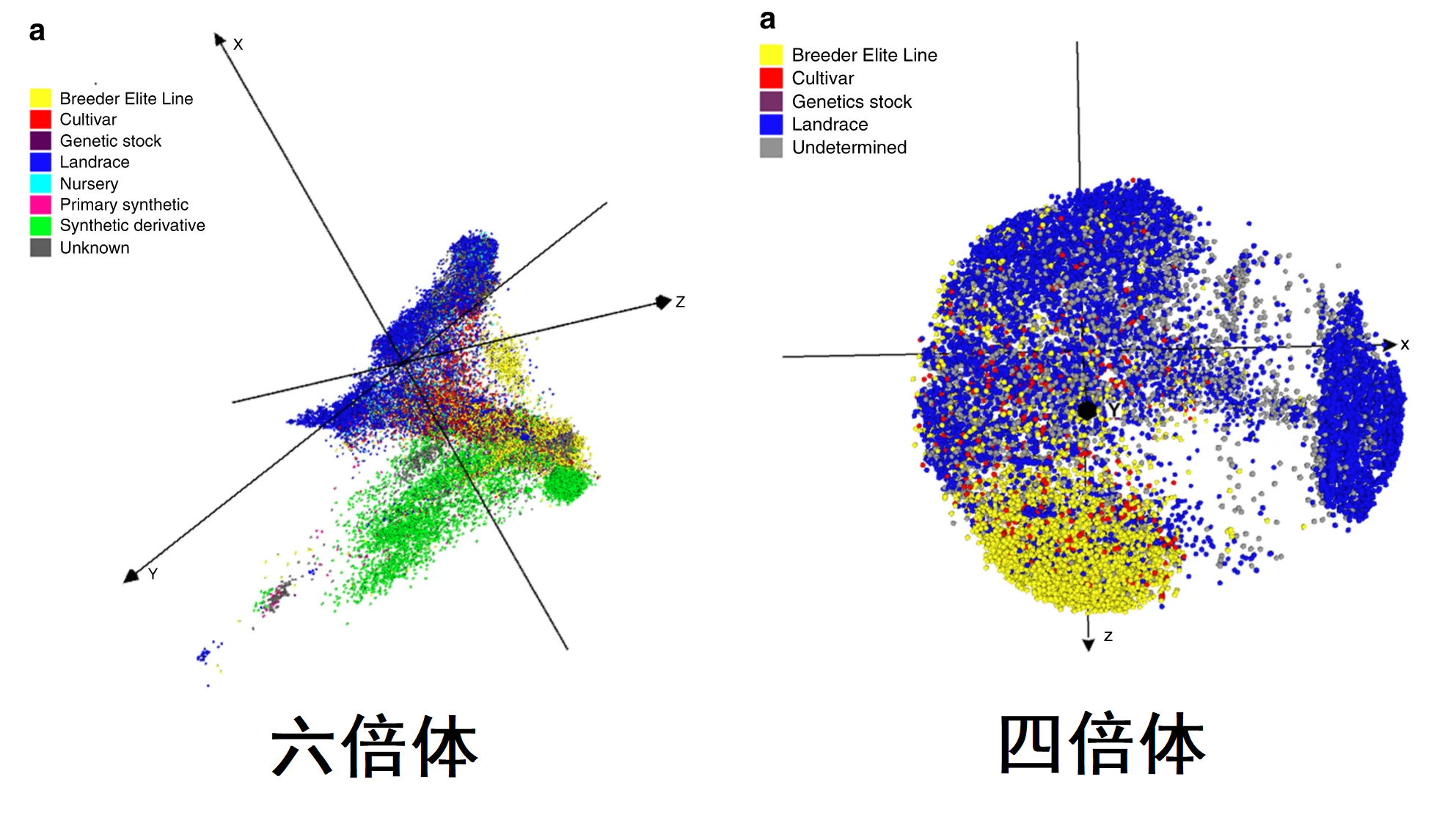
通过测序结果对样本进行分析,作者发现六倍体elite小麦相较于landrace品种,尚有大量变异未被利用;四倍体elite品种则均匀分布在“遗传多样性空间”内,象征着大部分多样性都已被利用(除了一大批来自Ethiopia的品种)。下图是压缩到三个特征维度的MDS图。作者首先采用“modified Roger's distance matrix”得出了样本间的距离,随后采用CurlyWhirly进行了可视化作图。由于采用了三个维度,作者在支持文件中提供了视频来从多个角度观察MDS图形。作者还对近缘种做了此分析。
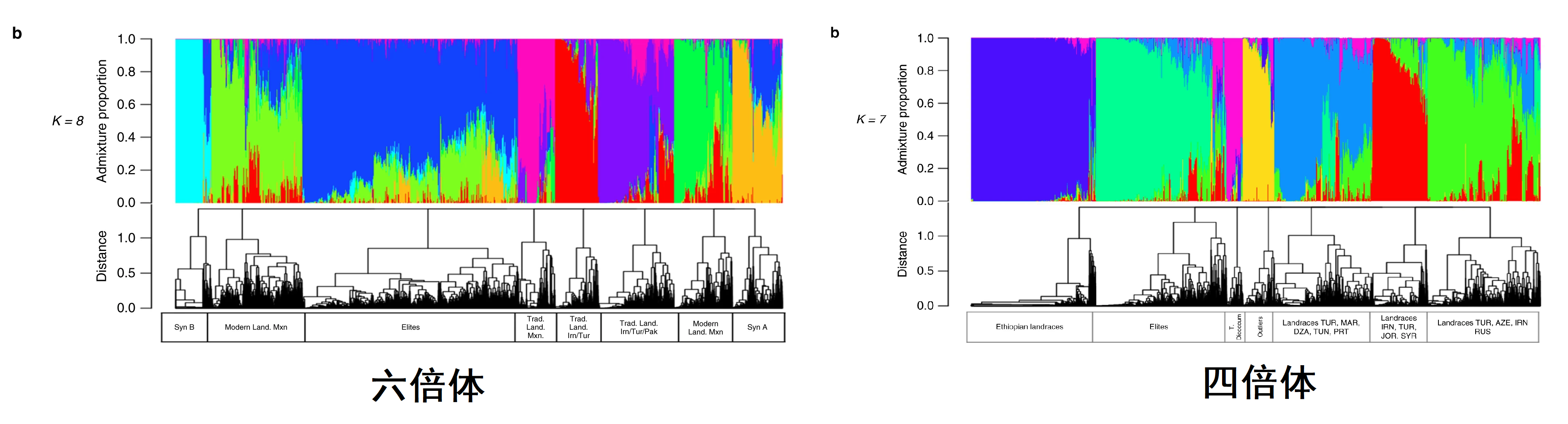
同时,作者采用了admixture分析,来观察基因组成分。通过(F_{ST})值区分cluster并使用AMOVA法进行分析,作者得出六倍体小麦群体中分组数为6-12时最informative的结论。结合不同的K值做出的成分图,作者进行了进一步讨论,如K=12时可将传统地方种分为四类、其中modern Mexican landraces对subgroup 7的elite种质有着更多贡献(相较于subgroup 1)。在四倍体小麦中,作者发现当K=7时解释性较好。作者还对近缘种做了此分析。
在上述“数据描述”过程之后,作者开始尝试寻找“核心种质”。作者通过expected heterozygosity ((He^2)), inbreeding, and Shannon indices来寻找“核心种质”,并筛出了了20%的样本(11235六倍体、3157四倍体和746近缘种)。经过全局多样性分析,验证了三个核心种质之间差异明显。通过对每个个体组的分析,作者找到了一些“离群值”,并通过marker的匹配率识别出了一些误分组的样本。这些误分组的检测结果得到了CIMMYT和ICARDA的确认,证实了这一检测方法的可靠性。
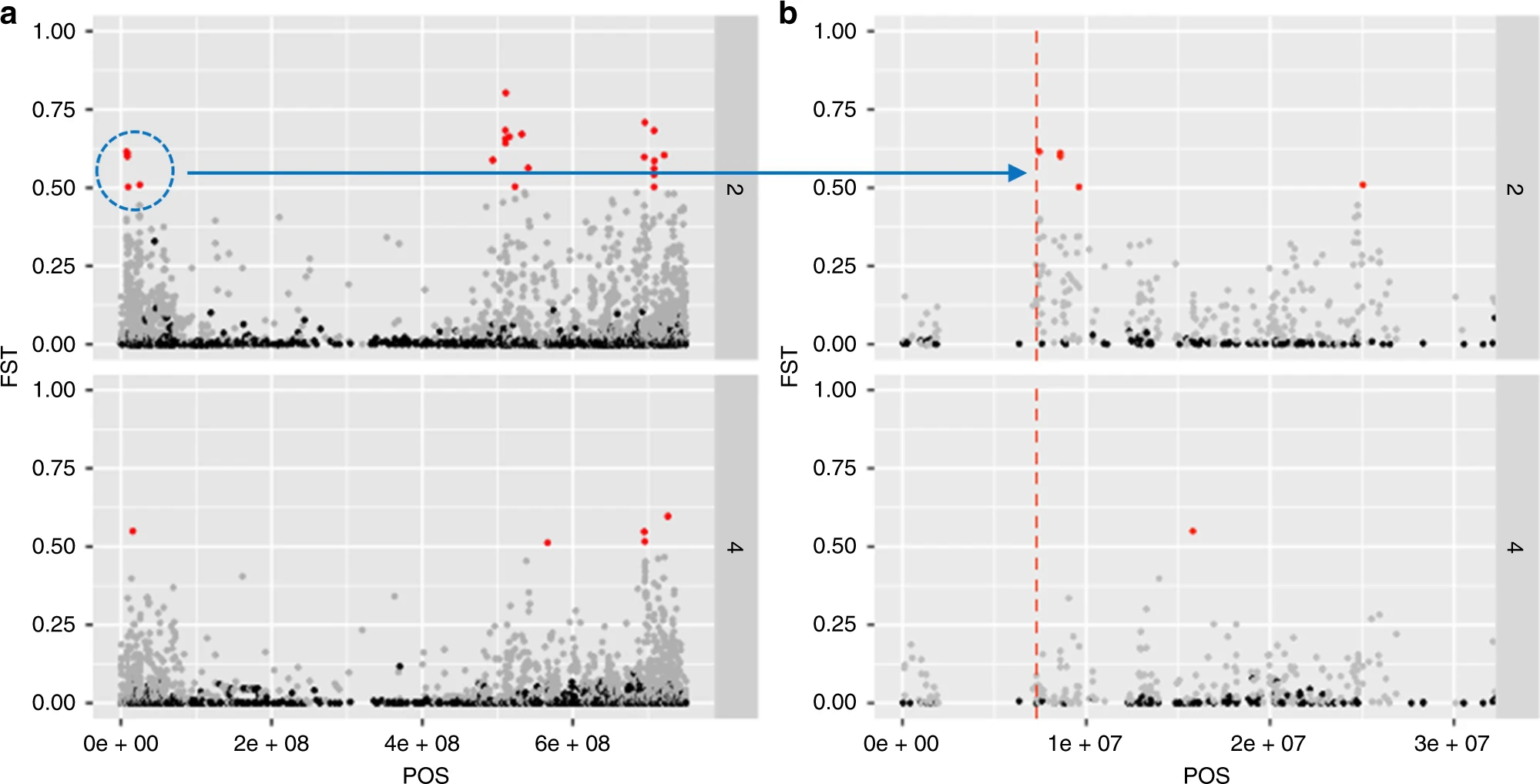
接着,作者通过基因组上的高(F_{ST})区域,来寻找受到正向选择的基因组区域。寻找主要目标在于能够区分传统种质与elite的和能够区分核心种质与elite的区域。该分析识别出了许多与已知关键农艺性状相关的区间,也发现了许多能够帮助解释近代育种史的区间。这一研究还能够为未来的育种提供目标allele。
为了进一步利用数据,作者对3870个样本测定了表型数据并进行了GWAS分析。表型包括GPC(籽粒蛋白含量)和SDS(可代表总面筋质量)。识别出的QTL与一报道的研究一致,证明了这一研究在标记辅助育种中的帮助。