转自:https://www.jianshu.com/p/a7af4f6e50c3
1.原始数据
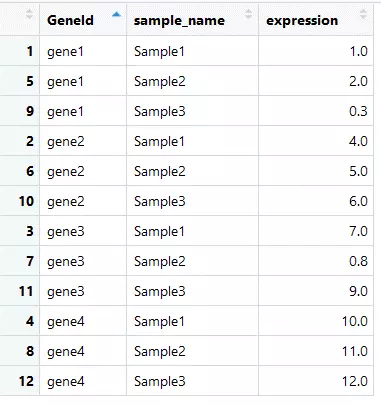
以上是原有的一个,再生成一个新的:
> gene_exp_tidy2 <- data.frame(GeneId = rep("gene5", times = 3),sample_name = paste("sample", 1:3, sep = ""), expression = 2:4) > gene_exp_tidy2 GeneId sample_name expression 1 gene5 sample1 2 2 gene5 sample2 3 3 gene5 sample3 4 > mode(gene_exp_tidy2) [1] "list" > typeof(gene_exp_tidy2) [1] "list"
//这个生成数据框的一句代码好厉害啊,学习了。
2.数据框拼接
# 1.使用R自带的函数rbind拼接 rbind(gene_exp_tidy,gene_exp_tidy2) # 2.使用dplyr的bind_rows函数 bind_rows(gene_exp_tidy,gene_exp_tidy2)
其中对dplyr中拼接函数的说明:

包括按行合并和按列合并。也就是会改变表中y的值。
3.交集
3.1构建一个gene1和gene5组成的数据框
gene_exp_tidy3 <- filter(gene_exp_tidy, GeneId == "gene1")%>% bind_rows(gene_exp_tidy2)
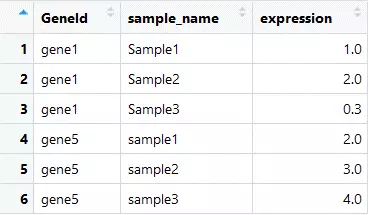
//就是过滤吧,从原来的当中根据列名过滤,列名中的内容=gene1的列,然后进行合并到变量2的行上。结果如上。
3.2求交集
# 求两个数据框的交集
intersect(gene_exp_tidy, gene_exp_tidy3)

3.3并集(自动去重)
# 求两个数据框的并集(自动去除重复)
union(gene_exp_tidy, gene_exp_tidy3)

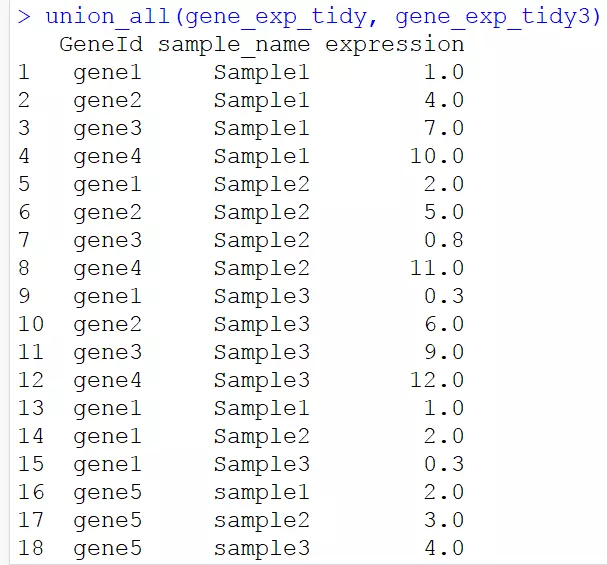
3.3并集(不去重)
# 求两个数据框的并集,不去除重复
union_all(gene_exp_tidy, gene_exp_tidy3)
//是三个元素完全相同,才会判断为相同的。
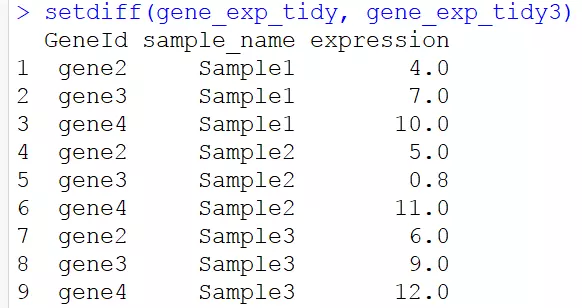
3.4求差集
# 求两个数据框的差集
setdiff(gene_exp_tidy, gene_exp_tidy3)
4表连接操作
4.1内连接
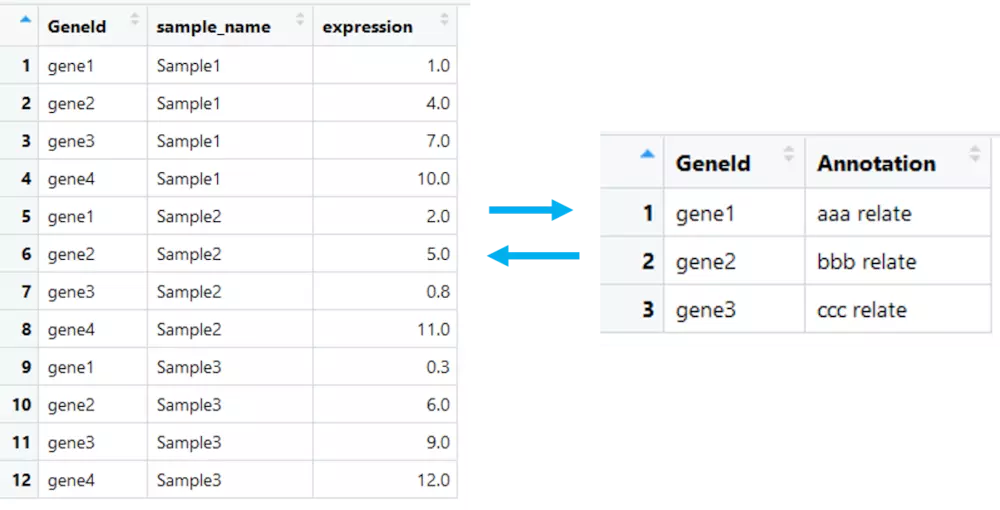
上图中,左边的数据框中有基因的表达信息,右边的数据框中有基因的功能注释,如何把两个数据框相关联,将基因功能注释添加到基因的表达信息中呢?
//也就是以左边的表为基准,查看当前geneid是否在右边表中出现,如果出现,则合并呗。
左连接:以第一个数据框为准,将第二个数据框的相关信息添加进去
# 将两张表以GeneId进行关联 left_join(gene_exp_tidy, gene_anno, by = "GeneId")
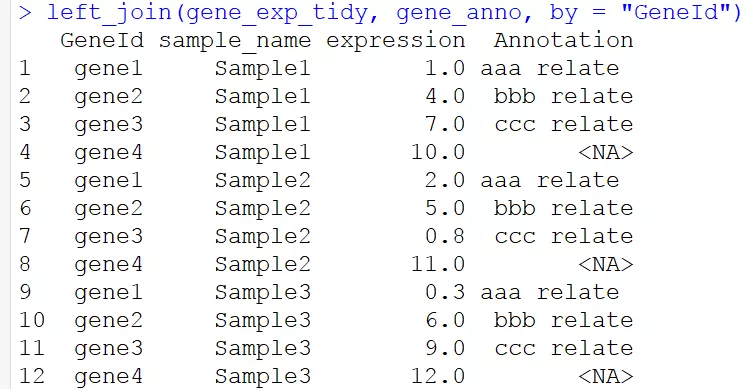
对于如果两个表的列名不恰好相同的话,使用以下语句:
# 使用rename函数将GeneId换成Geneid gene_anno <- rename(gene_anno, Geneid = GeneId) # 将两个数据框关联,设置两个数据框中相同的列名 left_join(gene_exp_tidy, gene_anno, by =c("GeneId" = "Geneid"))
by中是一个向量,左边表的列名=右边表的列名 。结果与上述是一样的结果。
4.2右连接
右连接:以第二个数据框为准,将第一个数据框中的相关信息添加进去
# 右连接 right_join(gene_exp_tidy, gene_anno, by =c("GeneId" = "Geneid"))

4.3内连接:保留两个数据框中共有的GeneId
# 内连接 inner_join(gene_exp_tidy, gene_anno, by =c("GeneId" = "Geneid"))

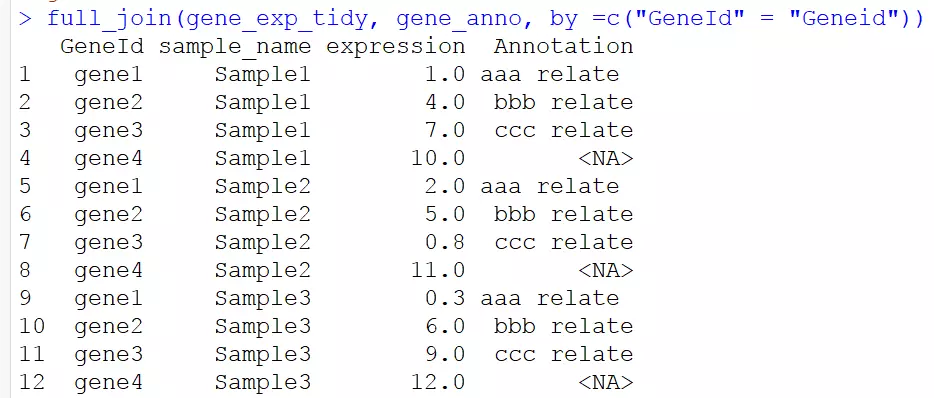
4.4全连接:保留两张表格中所有GeneId的信息
# 全连接 full_join(gene_exp_tidy, gene_anno, by =c("GeneId" = "Geneid"))
4.5半连接
半连接:只保留第二个数据框中包含的GeneId信息
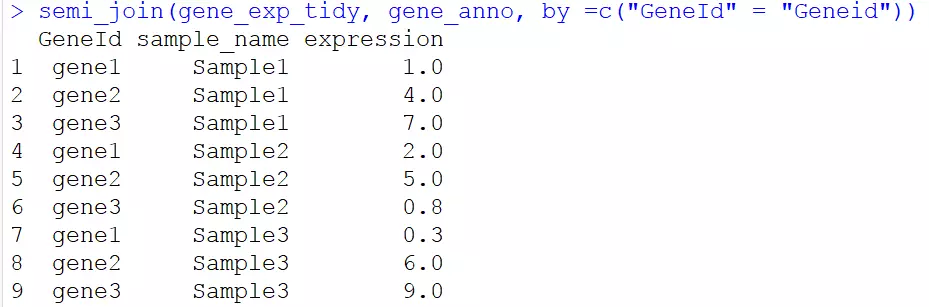
//应该是只根据c中的保留表2中的列,不会进行其他列的信息合并。
4.6反连接:只保留第二个数据框中不包含的GeneId信息
# 反连接 anti_join(gene_exp_tidy, gene_anno, by =c("GeneId" = "Geneid"))
